刚开始学习机器学习,有很多不懂得地方,也就是抱着书本啃,可能有很多理解不到位的地方,希望大家可以给我提出宝贵的意见。
如果有同学需要数据集可以在下面把邮箱留下来。
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
import numpy as np
import matplotlib.pyplot as plt
df_wine = pd.read_csv('H:\python\machine-learning\wine.data')
x,y = df_wine.iloc[:,1:].values, df_wine.iloc[:,0].values #1-13标记为x;第一列为y(0-0就是第一列)
#调用train_test_split分裂数据集,0.3:0.7 stratify=y是确保训练集和测试集具有相同的分类比例
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.3,random_state=0,stratify=y)
#PCA一定要进行标准化数据处理
sc = StandardScaler()
x_train_std = sc.fit_transform(x_train)
x_test_std = sc.transform(x_test)
#计算特征值和特征向量
cov_mat = np.cov(x_train_std.T) #计算表转化数据的协方差矩阵
eigen_vals,eigen_vecs = np.linalg.eig(cov_mat) #用linalg.eig来将特征值分解,产生含有13个特征值,对应的特征向量存储在13*13维度的矩阵中
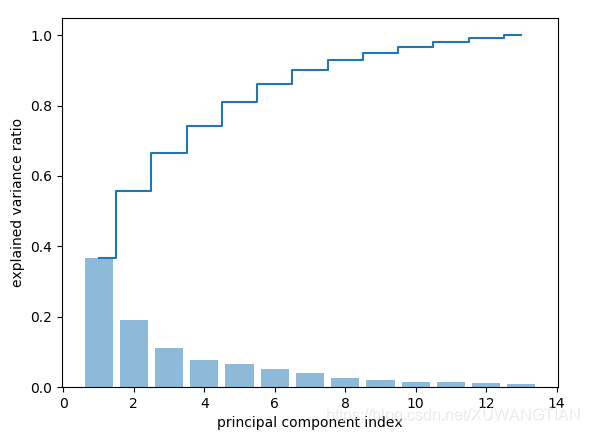
#计算总方差和解释方差
tot = sum(eigen_vals)
var_exp = [(i / tot) for i in sorted(eigen_vals)]
cum_var_exp = np.cumsum(var_exp) #用numpy.cumsum来计算总方差和
plt.bar(range(1,14),var_exp,alpha=0.5,align = 'center', label = 'individual explained variance')
plt.step(range(1,14),cum_var_exp, where = 'mid', label = 'cumulative explained variance')
plt.ylabel('explained variance ratio')
plt.xlabel('principal component index')
plt.show()
图像如下:
2.特征变换
在成功的把协方差矩阵分解成特征对之后,接着完成最后的三个步骤,将葡萄酒数据集变换成新的主成分轴。
(1)选择与前k个特征值相对应的特征向量
(2)构造投影矩阵W
(3)用投影矩阵变换D维输入数据集X已获得K维特征子空间。
通俗的说就是用投影矩阵把数据变换到低维子空间。
搜集前两个最大特征值的特征向量,捕获数据集约60%的方差
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:,i])for i in range(len(eigen_vals))]
eigen_pairs.sort(key =lambda k: k[0],reverse = True)
w = np.hstack((eigen_pairs[0][1][:,np.newaxis],eigen_pairs[1][1][:,np.newaxis]))
print ('Matrix W:\n',w)
输出
Matrix W:
[[ 0.12221148 0.49927666]
[-0.24391049 0.15419073]
[ 0.00369454 0.25170137]
[-0.249647 -0.12269609]
[ 0.13011026 0.31420427]
[ 0.39099441 0.05595656]
[ 0.41649235 -0.02637354]
[-0.31569983 0.07362469]
[ 0.2982478 -0.01094724]
[-0.07304049 0.53961007]
[ 0.31641846 -0.21061646]
[ 0.37253046 -0.23649287]
[ 0.29514562 0.38598117]]
3.主成分分析
下面是主成分分析的全部代码。
首先是分裂数据集0.3:0.7,计算特征值,将特征值最为大的两个特征向量提取。
对train data 进行训练
对test data 进行测试
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.colors import ListedColormap
from sklearn.linear_model import LogisticRegression
from sklearn.decomposition import PCA
df_wine = pd.read_csv('H:\python\machine-learning\wine.data')
x,y = df_wine.iloc[:,1:].values, df_wine.iloc[:,0].values #1-13标记为x;第一列为y(0-0就是第一列)
#调用train_test_split分裂数据集,0.3:0.7 stratify=y是确保训练集和测试集具有相同的分类比例
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.3,random_state=0,stratify=y)
#PCA一定要进行标准化数据处理
sc = StandardScaler()
x_train_std = sc.fit_transform(x_train)
x_test_std = sc.transform(x_test)
#计算特征值和特征向量
cov_mat = np.cov(x_train_std.T) #计算表转化数据的协方差矩阵
eigen_vals,eigen_vecs = np.linalg.eig(cov_mat) #用linalg.eig来将特征值分解,产生含有13个特征值,对应的特征向量存储在13*13维度的矩阵中
#计算总方差和解释方差
tot = sum(eigen_vals)
var_exp = [(i / tot) for i in sorted(eigen_vals, reverse = True)]
cum_var_exp = np.cumsum(var_exp) #用numpy.cumsum来计算总方差和
plt.bar(range(1,14),var_exp,alpha=0.5,align = 'center', label = 'individual explained variance')
plt.step(range(1,14),cum_var_exp, where = 'mid', label = 'cumulative explained variance')
plt.ylabel('explained variance ratio')
plt.xlabel('principal component index')
plt.show()
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:,i])for i in range(len(eigen_vals))]
eigen_pairs.sort(key =lambda k: k[0],reverse = True)
w = np.hstack((eigen_pairs[0][1][:,np.newaxis],eigen_pairs[1][1][:,np.newaxis]))
print ('Matrix W:\n',w)
x_train_std[0].dot(w)
x_train_pca = x_train_std.dot(w) #通过计算矩阵点将整个124*13维的训练集转换成两个主成分
#定义一个函数来完成二维数据决策边界的可视化
def plot_decision_regions(x,y,classifier,resolution=0.02):
#定义marker generator and color ma
markers = ('s','x','o','^','v')
colors = ('red','blue','lightgreen','gray','cyan')
cmap = ListedColormap(colors[:len(np.unique(y))])
#plot the decision surface
x1_min,x1_max = x[:,0].min() - 1, x[:,0].max() + 1
x2_min,x2_max = x[:,1].min() - 1, x[:,1].max() + 1
xx1,xx2 = np.meshgrid(np.arange(x1_min,x1_max,resolution),np.arange(x2_min,x2_max,resolution))
z = classifier.predict(np.array([xx1.ravel(),xx2.ravel()]).T)
z = z.reshape(xx1.shape)
plt.contourf(xx1,xx2,z,alpha=0.4,cmap=cmap)
plt.xlim(xx1.min(),xx1.max())
plt.xlim(xx2.min(),xx2.max())
#plot class samples
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=x[y == cl, 0],
y=x[y == cl, 1],
alpha = 0.6, c = cmap(idx), edgecolor = 'black',marker = markers[idx], label = cl)
#对训练集进行训练
pca = PCA(n_components = 2)
lr = LogisticRegression()
x_train_pca = pca.fit_transform(x_train_std)
x_test_pca = pca.transform(x_test_std)
lr.fit(x_train_pca,y_train)
plot_decision_regions(x_train_pca,y_train, classifier = lr)
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.legend(loc = 'lower left')
plt.show()
#测试集
plot_decision_regions(x_test_pca, y_test, classifier=lr)
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.legend(loc = 'best')
plt.show()
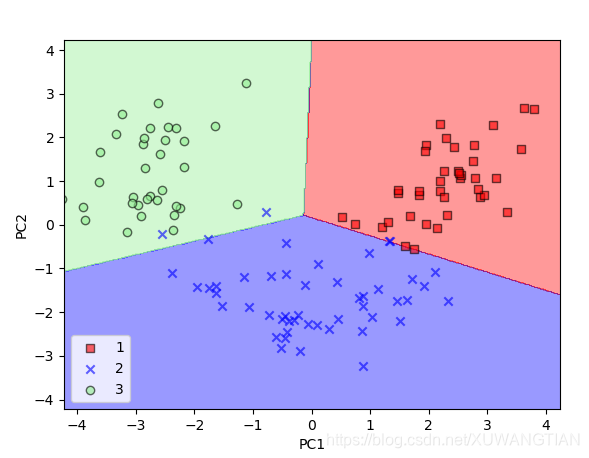
结果如下:
下图是训练集分界可视化
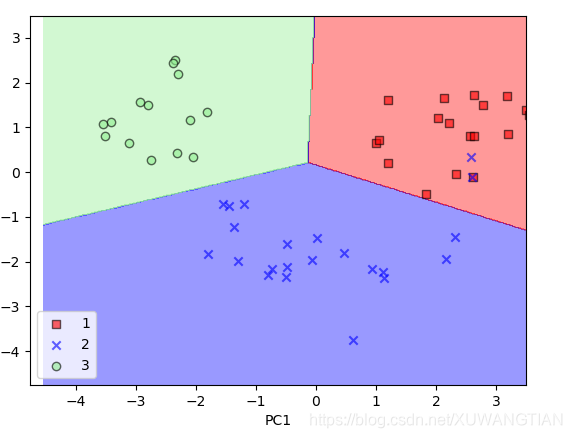
下图是测试集分解可视化






















 2200
2200











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








