1.从file里面读取大分子受体蛋白4zau.pdb,在edit里面加氢,去水,计算电荷,设置原子类型ASSIGN AD4,去水,最后保存为PDBQT的格式。
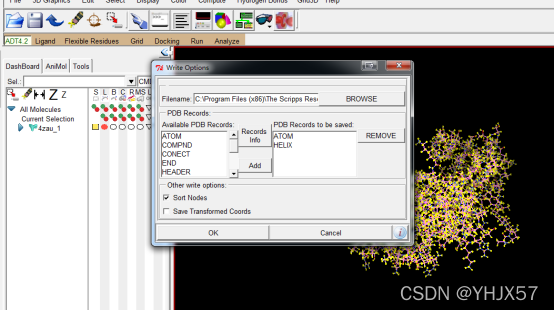
1.PDBQT的格式。
2.从ligand里面open小分子配体azd9291.pdb.弹出提示框

点击确定。然后在ligand里面choose该配体。
接下来判定配体的root。ADT菜单栏:Ligand->Torsion Tree->Detect Root,选择配体可扭转的键。Ligand->Torsion Tree->Choose Torsions

点击done,保存为pdbqt文件。ADT菜单栏:“Ligand->Output->Save as PDBQT”
3.设置grid。
删掉屏幕中的配体,打开受体分子,ADT菜单栏:“Grid->Macromocule->Open”,打开“r.pdbqt”。弹出的对话框如下图,点击Yes及确定




打开配体分子,ADT菜单栏:“Grid->Set Map Types->Open ligand”

设置GridBox。ADT菜单栏:“Grid->Grid Box…”,打开Grid Options对话框。调整X,Y,Z及格子中心坐标。我们这里就设置成如下图
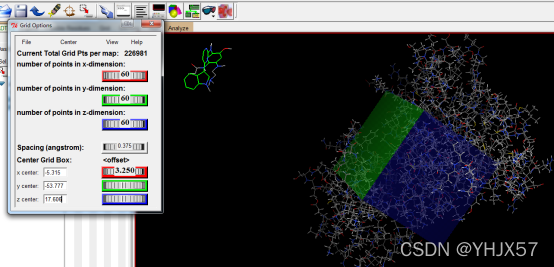
坐标是从pdb文件里找的关键氨基酸残基的CA坐标,大小都是设置成60.(看小木虫里被人说的,60差不多就可以包括附近的关键氨基酸残基了)

设置完成后点击Grid Options 菜单中:File→Close saving current 保存并关闭对话框。
点击File——Close saving current保存盒子信息,选择Grid——Output——Save GPF,保存为protein_ligand.gpf文件(注意Windows下要手动添加文件名后缀);

4.点击Docking——Macromolecule——Set RigidFilename,选择4zau.pdbqt文件,将受体蛋白质设置为刚性;
同样的方法Docking——Ligand——Choose,选择配体,设置初始位置等信息,点击Accept即可;

选择Docking——Output——Lamrckian GA 4.2,选择拉马克遗传算法作为对接算法,保存成为protein_ligand.dpf文件(Windows下手动加后缀),dpf文件中包含了分子对接的信息,默认对接的构象数为10个,可以用文本编辑器打开dpf文件,手动修改对接的构象数目(ga_run 10);

5.至此,分子对接的准备文件就完成了,接下来就是运行Autogrid和AutoDock程序了,
运行autogrid,ADT菜单栏:Run->Run Autogrid

点击launch弹出一个小窗口,瞬间消失
6. 准备DPF文件运行AutoDock
在窗口中删掉受体和配体分子,运行AutoDock程序,
运行autogrid,ADT菜单栏:Run->Run AutoDock

点击lanuch后会出现一个小框瞬间消失了。
但是最后的文件夹里面没收生成dlg文件。我就做到这里了,每次都是这样不出结果。











 本文详细指导如何使用ADT工具从大分子受体蛋白pdb文件处理成PDBQT格式,选取小分子配体并设定根键,设置Grid,进行刚性和初始位置设置,最后通过Autogrid和AutoDock进行分子对接,生成protein_ligand.dpf文件。
本文详细指导如何使用ADT工具从大分子受体蛋白pdb文件处理成PDBQT格式,选取小分子配体并设定根键,设置Grid,进行刚性和初始位置设置,最后通过Autogrid和AutoDock进行分子对接,生成protein_ligand.dpf文件。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








