1、串联重复和非串联重复(一般重复)的定义
串联重复:重复序列以串联方式排列,即一个序列紧接着另一个相同的序列。例如,一个基因的外显子序列正常情况下为【ATGCGT】,如果发生了串联重复,可能变为【ATGCGTATGCGT】。
判断方法:通过将变异后的基因序列与正常基因序列进行比对。如果发现重复的外显子序列是连续排列的,且重复单元之间没有间隔序列,就可以判断为串联重复。例如,使用生物信息学软件IGV展示序列比对结果,会有助于判断重复单元的排列方式。
一般重复:指基因组中其他类型的重复,不遵循串联排列的方式,重复序列不是首尾相连的,而是分散在基因的不同位置。例如,一个基因的外显子序列为【ATGCGT】,发生了非串联重复,可能就变为【ATGCGT……ATGCGT】,它们没有串联在一起,而是被其他序列间隔开。
判断方法:同样进行序列比对,但如果发现重复的外显子序列之间有其他序列间隔,且这些间隔序列与重复单元没有明显的重复关系,就可以判断为一般重复。
由于在临床基因测序数据解读过程中,基因内部的不同重复方式对基因功能的影响不一样;此外,对于该重复的致病性评级也不一样,如果是串联重复,就有可能会影响整个基因的编码,如果打断阅读框,造成了移码,则可以参考ACMG打分规则中的Loss of function (LOF)变异进行打分;所以,如果可以根据现有的数据结果对重复类型进行区分,有很重要的临床意义;
2、通过IGV图判断NF1基因的13-33号外显子的重复为串联重复还是非串联重复
案例背景:基因组版本hg19、WES测序,生信流程分析结果显示chr17:29541367-29587533_dup,为NF1基因13-33号外显子的重复,如下图所示;

2.1. 打开IGV图,根据生信分析结果chr17:29541367-29587533_dup去初步判断断点位置;由于区段较大,展示不完整,因此,可以对头尾两个位置分开查看,比如分别输入包括头尾的两个区段chr17:29530367-29542367和chr17:29587033-29587933;
chr17:29530367-2954236区段展示如下(记得勾选Show soft clips,这样能快速找到比对异常的序列),如下图;

2.2.将展示内容放大,点击29536965位置的[T],会出现下图信息,根据该比对信息对结果进行解读;
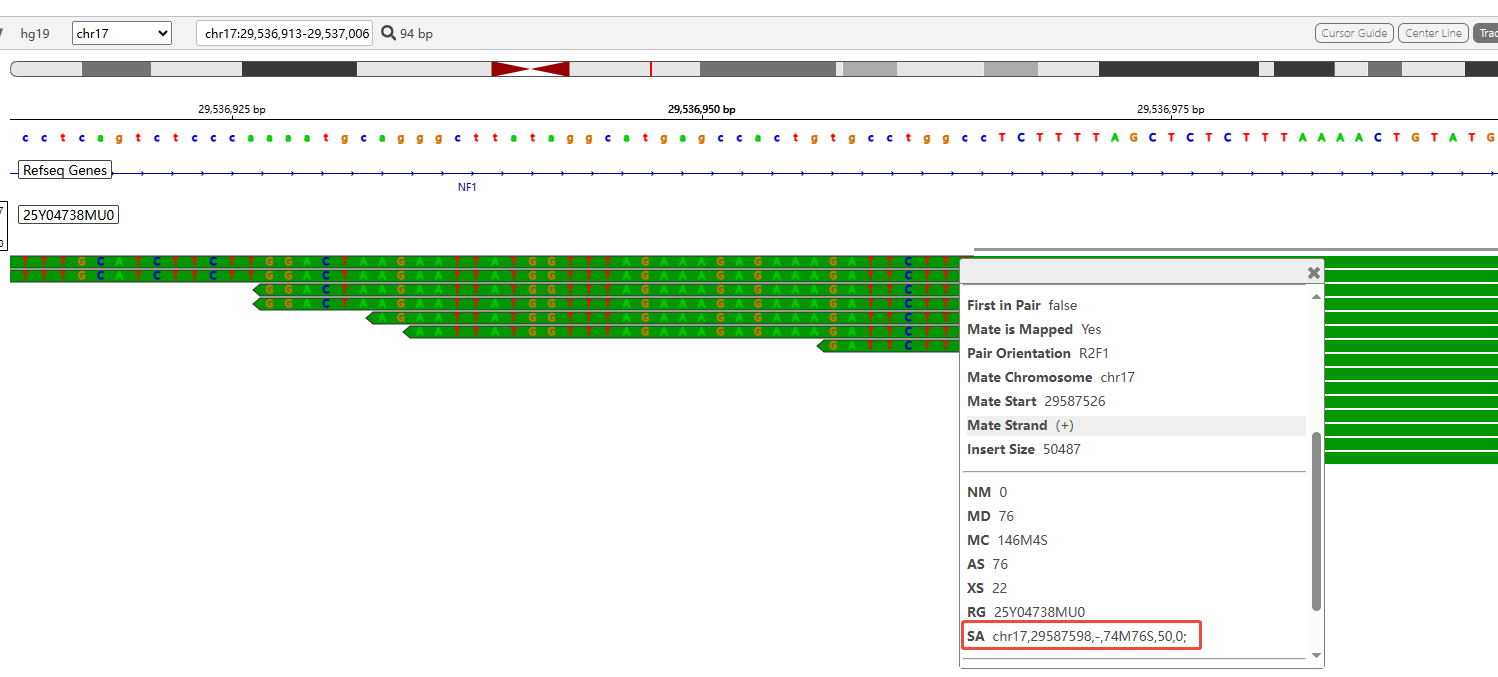
2.3.比对结果各参数信息解释如下:


2.4. 根据变异信息找断点位置:
这里面主要用到的信息有:
Alignment Start:从chr17:29,536,965这个位置开始,有变异发生,上游的断点就在这个位置;
Cigar:74S76M,74个碱基软剪切(不匹配) + 76个碱基完全匹配,代表这一条read的150个碱基里面,有74个碱基没有与参考基因组序列匹配,而有76个碱基与参考序列匹配;
SA:里面的补充比对信息很有用,有利于找到下游断点;补充比对信息的位置,代表Cigar里面与该位置没有匹配到的74个碱基匹配到了chr17:29,587,598位置,而chr17:29,587,598位置为起始位置,加上与chr17:29,587,598匹配上的74bp,则是断点位置(也就是没有匹配上的那76bp断点位置);chr17:29,587,598+74=chr17:29,587,672;于是去查看chr17:29,587,672位置的详细情况,如下图所示;而下游的SA信息为:chr17,29,536,965,-,111S39M,60,0;与上游的断点chr17:29,536,965重合,因此,该重复的具体区域是:chr17:29536965-29587671_dup;

2.5. 如何判断为串联重复还是非串联重复
根据重复位置chr17:29536965-29587671_dup上下游的序列信息,可以看到上游红框中未比对到基因组的序列信息刚好和下游的红色参考基因组序列完全相同,而下游的绿框中未比对到基因组的序列信息刚好和上游的绿色色参考基因组序列完全相同;
上游序列如下图:

下游序列如下图:

由于绿色方框内的reds序列与绿色方框内的基因组序列一致,而由于红色方框内的reds序列与红色方框内的基因组序列一致;而这些红色和绿色方框内的reads其实是在二代测序的时候,每一条reads都是150bp,也就是说,他们是一条reads;在chr17:29536965-29587671_dup的下游29587671位置,测完以后,紧跟上的序列又是29536965位置的序列,因此可以判断该重复为串联重复;
2.6. 临床意义:由于该重复片段大小为29536965-29587671+1=50705bp,打断了阅读框,因此可能会造成NF1基因编框的移码;
3. 实验验证
串联重复
PCR扩增:设计特异性的引物,针对重复单元进行PCR扩增。如果重复是串联的,PCR产物的大小会随着重复次数的增加而增加。例如,正常基因的PCR产物为100bp,如果发生了串联重复,PCR产物可能为200bp、300bp等,且电泳图谱中会出现多个条带,条带大小呈等差数列。
测序验证:对PCR产物进行测序,可以直接观察到重复单元的排列方式。如果测序结果显示重复单元是首尾相连的,就可以判断为串联重复。
一般重复(非串联重复)
PCR扩增:同样设计特异性的引物,但一般重复的基因PCR产物大小可能不会随着重复次数的增加而明显变化,因为重复单元之间有间隔序列。例如,正常基因的PCR产物为100bp,即使发生了重复,PCR产物可能仍为100bp左右,但电泳图谱中可能会出现多个条带,条带大小没有明显的等差数列关系。
测序验证:对PCR产物进行测序,如果测序结果显示重复单元之间有间隔序列,且这些间隔序列与重复单元没有明显的重复关系,就可以判断为一般重复。
总之,判断外显子重复是串联重复还是一般重复,需要综合运用序列比对分析、基因结构分析和实验验证等多种方法。




























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










