算法基本介绍
RANSAC(RAndom SAmple Consensus,随机采样一致)是一种随机参数估计算法,常常应用于二维图像的拟合、分割等等,由于是估计数学模型参数的迭代算法,因此也被用于三维平面、球的估计。
RANSAC算法由Fischler和Bolles于1981年提出,是一种从数据集合中迭代稳健估计模型参数的方法。该算法的基本思想是不断地从数据集合中随机抽取样本集,寻求支持更多局内点的模型参数,然后利用模型余集检验获得的模型参数,通过一定次数的迭代来达到最大的一致性概率,并将该采样样本集作为理解的样本集,且参数解的正确性由样本余集检验支撑。其中数据集合中包含正确数据(即内点inliers)和异常数据(外点outliers)在关键点匹配中为误匹配的点。同时RANSAC假设:在给定一组含有少部分“内点”的数据,存在一个程序可以估计出符合“内点”的模型。
RANSAC与最小二乘拟合不同,最小二乘法考虑整个数据集合中所有的数据,让数据集整体的误差最小,而RANSAC首先假设数据整体具有统一共性(即符合某个模型),为了达到这一共性,可以适当的排除一些数据,为了解决样本中外点的影响,最多可以处理50%的外点情况。
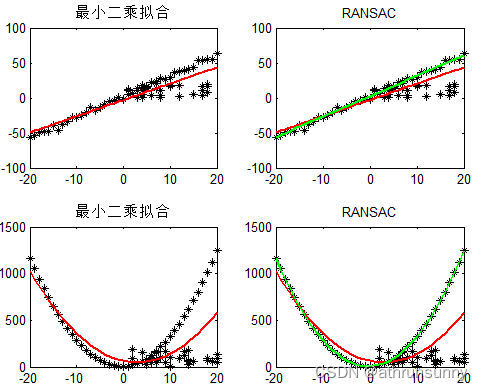
上图中给出了一阶与二阶曲线使用最小二乘法以及RANSAC拟合的效果对比。
算法思想
RANSAC算法的输入:一组观测数据,一个可以解释或者适应于观测数据的参数化模型,一些可信的参数。
RANSAC通过反复选择数据中的一组随机子集来达成目标。被选取的子集被假设为局内点,并用下述方法进行验证:
1.有一个模型适应于假设的内点,即所有的未知参数都能从假设的内点计算得出。
2.用1中得到的模型去测试所有的其它数据,如果某个点适用于估计的模型,认为它也是内点。
3.如果有足够多的点被归类为假设的内点,那么估计的模型就足够合理。
4.然后,用所有假设的内点去重新估计模型,因为它仅仅被初始的假设内点估计过。
5.最后,通过估计内点与模型的错误率来评估模型。
这个过程被重复执行固定的次数,每次产生的模型要么因为内点太少而被舍弃,要么因为比现有的模型更好而被选用。
举一个实际例子:
1)给定一个数据集,从中选择建立模型所需的最小样本数(例如直线最少由两个点确定,所以最小样本数是2,而平面可以根据不共线三点确定,所以最小样本数为3),记选择数据集为
;
2)使用选择的数据集计算得到一个数学模型
;
3)用计算的模型去测试数据集中剩余的点,如果测试的数据点在误差允许的范围内,则将该数据点判为内点(inlier),否则判为外点(outlier),记所有内点组成的数据集为
,表示为
的一致性集合;
4)比较当前模型和之前推出的最好的模型的“内点”的数量,记录最大“内点”数量时模型参数和“内点”数量;
5)重复1-4步,直到迭代结束或者当前模型已经足够好了(“内点数目大于设定的阈值”)。
迭代的次数可通过以下方式估计出来:
假设内点在数据集中的占比为,可表示为
,那么我们每次计算模型使用
个点的情况下,选取的点至少有一个外点的情况(采样失败的概率)就是
, 那么
次迭代计算模型都至少采样到一个外点的概率为
,那么
次采样至少有一次成功的概率记为
,通过
变换并在等式两边同时取对数可以得到
。在基本子集的迭代次数内,至少有一次采样使得采样子集内的
个点均为内点,从而保证在迭代次数中,至少存在一次采样能够取得目标函数的最大值,因此终止循环条件应满足
。
内点的占比是一个先验值,如果一开始不知道这个先验值
,可以采用自适应迭代的方法,用当前的内点的比值来当成
来估算迭代的次数。然后通过不断迭代,内点的比值也逐渐增大,再用新的更大的内点比值去代替
的值。
Pseudocode
伪代码参考自wiki
Given:
data – A set of observations.
model – A model to explain the observed data points.
n – The minimum number of data points required to estimate the model parameters.
k – The maximum number of iterations allowed in the algorithm.
t – A threshold value to determine data points that are fit well by the model (inlier).
d – The number of close data points (inliers) required to assert that the model fits well to the data.
Return:
bestFit – The model parameters which may best fit the data (or null if no good model is found).
iterations = 0
bestFit = null
bestErr = something really large // This parameter is used to sharpen the model parameters to the best data fitting as iterations go on.
while iterations < k do
maybeInliers := n randomly selected values from data
maybeModel := model parameters fitted to maybeInliers
confirmedInliers := empty set
for every point in data do
if point fits maybeModel with an error smaller than t then
add point to confirmedInliers
end if
end for
if the number of elements in confirmedInliers is > d then
// This implies that we may have found a good model.
// Now test how good it is.
betterModel := model parameters fitted to all the points in confirmedInliers
thisErr := a measure of how well betterModel fits these points
if thisErr < bestErr then
bestFit := betterModel
bestErr := thisErr
end if
end if
increment iterations
end while
return bestFitpython demo
定义模型为2D线性回归模型
from copy import copy
import numpy as np
from numpy.random import default_rng
rng = default_rng()
class RANSAC:
def __init__(self, n=10, k=100, t=0.05, d=10, model=None, loss=None, metric=None):
self.n = n # `n`: Minimum number of data points to estimate parameters
self.k = k # `k`: Maximum iterations allowed
self.t = t # `t`: Threshold value to determine if points are fit well
self.d = d # `d`: Number of close data points required to assert model fits well
self.model = model # `model`: class implementing `fit` and `predict`
self.loss = loss # `loss`: function of `y_true` and `y_pred` that returns a vector
self.metric = metric # `metric`: function of `y_true` and `y_pred` and returns a float
self.best_fit = None
self.best_error = np.inf
def fit(self, X, y):
for _ in range(self.k):
ids = rng.permutation(X.shape[0])
maybe_inliers = ids[: self.n]
maybe_model = copy(self.model).fit(X[maybe_inliers], y[maybe_inliers])
thresholded = (
self.loss(y[ids][self.n:], maybe_model.predict(X[ids][self.n:]))
< self.t
)
inlier_ids = ids[self.n:][np.flatnonzero(thresholded).flatten()]
if inlier_ids.size > self.d:
inlier_points = np.hstack([maybe_inliers, inlier_ids])
better_model = copy(self.model).fit(X[inlier_points], y[inlier_points])
this_error = self.metric(
y[inlier_points], better_model.predict(X[inlier_points])
)
if this_error < self.best_error:
self.best_error = this_error
self.best_fit = better_model
return self
def predict(self, X):
return self.best_fit.predict(X)
def square_error_loss(y_true, y_pred):
return (y_true - y_pred) ** 2
def mean_square_error(y_true, y_pred):
return np.sum(square_error_loss(y_true, y_pred)) / y_true.shape[0]
class LinearRegressor:
def __init__(self):
self.params = None
def fit(self, X: np.ndarray, y: np.ndarray):
r, _ = X.shape
X = np.hstack([np.ones((r, 1)), X])
self.params = np.linalg.inv(X.T @ X) @ X.T @ y
return self
def predict(self, X: np.ndarray):
r, _ = X.shape
X = np.hstack([np.ones((r, 1)), X])
return X @ self.params
if __name__ == "__main__":
regressor = RANSAC(model=LinearRegressor(), loss=square_error_loss, metric=mean_square_error)
X = np.array(
[-0.848, -0.800, -0.704, -0.632, -0.488, -0.472, -0.368, -0.336, -0.280, -0.200, -0.00800, -0.0840, 0.0240,
0.100, 0.124, 0.148, 0.232, 0.236, 0.324, 0.356, 0.368, 0.440, 0.512, 0.548, 0.660, 0.640, 0.712, 0.752, 0.776,
0.880, 0.920, 0.944, -0.108, -0.168, -0.720, -0.784, -0.224, -0.604, -0.740, -0.0440, 0.388, -0.0200, 0.752,
0.416, -0.0800, -0.348, 0.988, 0.776, 0.680, 0.880, -0.816, -0.424, -0.932, 0.272, -0.556, -0.568, -0.600,
-0.716, -0.796, -0.880, -0.972, -0.916, 0.816, 0.892, 0.956, 0.980, 0.988, 0.992, 0.00400]).reshape(-1, 1)
y = np.array(
[-0.917, -0.833, -0.801, -0.665, -0.605, -0.545, -0.509, -0.433, -0.397, -0.281, -0.205, -0.169, -0.0531,
-0.0651, 0.0349, 0.0829, 0.0589, 0.175, 0.179, 0.191, 0.259, 0.287, 0.359, 0.395, 0.483, 0.539, 0.543, 0.603,
0.667, 0.679, 0.751, 0.803, -0.265, -0.341, 0.111, -0.113, 0.547, 0.791, 0.551, 0.347, 0.975, 0.943, -0.249,
-0.769, -0.625, -0.861, -0.749, -0.945, -0.493, 0.163, -0.469, 0.0669, 0.891, 0.623, -0.609, -0.677, -0.721,
-0.745, -0.885, -0.897, -0.969, -0.949, 0.707, 0.783, 0.859, 0.979, 0.811, 0.891, -0.137]).reshape(-1, 1)
regressor.fit(X, y)
import matplotlib.pyplot as plt
plt.style.use("seaborn-darkgrid")
fig, ax = plt.subplots(1, 1)
ax.set_aspect(1)
plt.scatter(X, y)
line = np.linspace(-1, 1, num=100).reshape(-1, 1)
plt.plot(line, regressor.predict(line), c="peru")
plt.show()
运行可得到:

当然scikit-image 中也有相应的实现,感兴趣的可以看看。
RANSAC优点与缺点
RANSAC的优点是它能鲁棒的估计模型参数。例如,它能从包含大量外点的数据集中估计出高精度的参数。
RANSAC的缺点是它计算参数的迭代次数没有上限;如果设置迭代次数的上限,得到的结果可能不是最优的结果,甚至可能得到错误的结果。RANSAC只有一定的概率得到可信的模型,概率与迭代次数成正比。RANSAC的另一个缺点是它要求设置跟问题相关的阀值。
RANSAC只能从特定的数据集中估计出一个模型,如果存在两个(或多个)模型,RANSAC不能找到别的模型。

























 2308
2308

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










