首先是数据准备
library(stringr)
load(file = "exp_and_clinical_group.Rdata")
load(file = "candidate_genes.Rdata")
tumor_exp_t<- read.csv("tumor_exp_d.csv",header = TRUE,stringsAsFactors = F)
normal_exp_t <- read.csv("normal_exp_d.csv",header = TRUE,stringsAsFactors = F)
#上面两行防止出现“不是numeric"这种错误
#这里获得的表达数据框的格式是列名为变量名及基因名,行名为样本名,表达值为numeric形式
#前面的步骤要用一下write.table,这里才能用read.csv
tumor_exp_t_candidate <- tumor_exp_t[candidate_genes] #按列名取子集
normal_exp_t_candidate <- normal_exp_t[candidate_genes]
normal_exp_t_candidate <- rbind(tumor_exp_t_candidate,normal_exp_t_candidate)
table(str_sub(rownames(tumor_normal_exp_t_candidate),14,15))
group1 <- ifelse(as.numeric(str_sub(rownames(tumor_normal_exp_t_candidate),14,15))<10,'tumor','normal')
#这里14,15是因为这里使用的是TCGA的数据,TCGA的样本名14,15位表示<10的为肿瘤,>10的为正常组织
table(group1)
group1 <- factor(group1,levels = c("normal","tumor"))
table(group1)
#group1里记录了表达矩阵中患者ID的对应的组织类型,且和表达矩阵的顺序一样
#构建样本号与肿瘤分类对应关系
tumor_normal_exp_group_t_candidate <- data.frame(tumor_normal_exp_t_candidate,group = group1)
class(tumor_normal_exp_group_t_candidate[1,1])
class(tumor_normal_exp_group_t_candidate$group)
#现在tumor_normal_exp_group_t_candidate就是包含需要的特征,样本名为行名,分类因子在最后一列的数据框,里面的数字为numeric形式,目前数据格式应该就符合SVM的要求了
save(tumor_normal_exp_group_t_candidate,file = "predata_SVM.Rdata")然后是用e1071包训练SVM模型
library(dplyr)
library(e1071)
library(caret)#用于交叉验证
library(pROC)
library(ggplot2)
library(ggpubr)
library(caTools)#有sample.split函数,用原图将数据按预定义的比例分成两组,同时保留数据中不同标签的相对比率,用于将数据分为训练和测试子集
library(ROCR)
load(file = "predata_SVM.Rdata")
set.seed(123) #设置随机种子,保证结果可复现
split1 <- sample.split(tumor_normal_exp_group_t_candidate$group,SplitRatio = 0.8)
#0.8的意思是80%为训练集,20%为测试集
train_data1 <- subset(tumor_normal_exp_group_t_candidate,split1 == TRUE)
test_data1 <- subset(tumor_normal_exp_group_t_candidate,split1 == FALSE)
#定义训练集特征和目标变量
X_train1 <- train_data1[,1:(ncol(train_data1)-1)]
Y_train1 <- as.factor(train_data1[,ncol(train_data1)])
#创建并训练SVM模型
svm_model1 <- svm(x = X_train1, y = Y_train1)
#在训练集上进行预测
train_predictions1 <- predict(svm_model1, newdata = X_train1, probability = TRUE)
#输出预测结果
table(Y_train1,train_predictions1)
##参数调整
#创建参数网格
param_grid <- expand.grid(
sigma = c(0.1, 1, 1/35),
C = c(0.1, 1, 10)
)
#这里sigma的0.03是用默认参数1/特征数量也就是1/35得到的
#定义交叉验证的控制参数,这里是10折交叉验证,直接用caret包里的trainControl函数,就不用写for循环了
ctr1 <- trainControl(method = "cv", number = 10, verboseIter = FALSE)
#进行参数调节
#caret包里包含十折交叉验证的函数就是train函数
#举例train(Species ~., data = iris, method = "svmRadial", trControl = trainControl(method = "cv", number = 10))
#"cv"就是cross vailidation交叉验证
#别忘加随机种子set.seed(123)保证结果可重复
tuned_model1 <- train(
x = X_train1,
y = Y_train1,
method = "svmRadial",
tuneGrid = param_grid,
trControl = ctr1,
probability = TRUE
)
#输出最佳参数配置
print(tuned_model1)
#这里得到最佳参数是sigma = 1/35,C = 10
#使用最佳参数训练模型
svm_final_model1 <- svm(x = X_train1, y = Y_train1,kernel = 'radial', sigma = 0.02857143, C = 1.0 ,probability = TRUE)
#在测试集上使用最佳参数配置进行预测
X_test1 <- test_data1[,-ncol(test_data1)]
Y_test1 <- as.factor(test_data1[,ncol(test_data1)])
test_predicitons1 <- predict(svm_final_model1, newdata = X_test1, probability = TRUE, decision.values = TRUE)
#输出训练结果,这个结果的显示就是混淆矩阵
table(Y_test1, test_predicitons1)然后使用pROC包画ROC曲线
#用pROC绘制ROC
prob_estimates <- attr(test_predicitons1, "probabilities")#此行代码提取出模型预测的测试集的概率
data_pre <- data.frame(prob = prob_estimates[,1],obs = test_data1$group)
data_pre <- data_pre[order(data_pre$prob),]
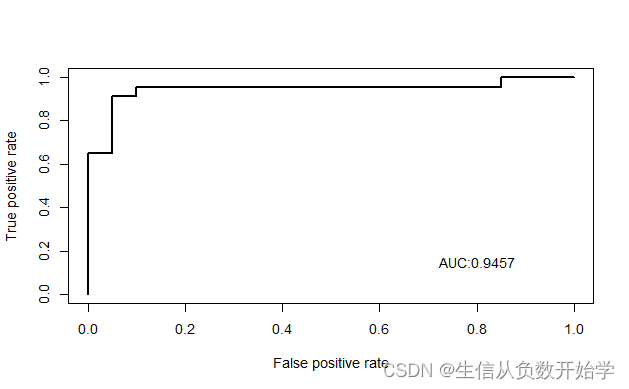
modelroc <- roc(test_data1$group,prob_estimates[,1],
plot = TRUE, legacy.axes = TRUE, xlab = "False positive rate",
ylab = "True postitive rate",print.auc = TRUE)
plot(1-modelroc$specificities, modelroc$sensitivities, type = "l", lty = 1, lwd = 2, xlab = "False positive rate", ylab = "True positive rate")
#参数legacy.axes = TRUE是把纵坐标设置为1-sensitivity,然后方便修改纵坐标
text(0.8,0.15, labels = 'AUC:0.9457', font = 1)
#type,lty,lwd都是线的一些参数,xlab、ylab设置的是横纵坐标的名称最后得到的tpr,fpr的ROC曲线就是下图这个样子,第一次用编程画图像,这个是与训练同一数据集里的数据做的(且测试和训练的case和control的比例被用caTools包里的sample.split函数设置为一样的了)测试集,真测试还得找其他数据集进行验证。
关于SVM的线性可分数学推导可以在B站的马少平老师讲的就很清楚,便于理解SVM的数学思想






















 209
209











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








