
关键词:长读长测序;准确度;DNAscope LongRead;
引言
第三代测序技术凭借其超长读长特性在基因组学研究中发挥重要作用,能够更好地解析复杂区域、结构变异和重复序列,显著提高了基因组组装和变异检测的准确性,但面临着准确性和计算效率的挑战。因此,本文将介绍Sentieon 开发的DNAscope LongRead 。该工具不仅继承了Sentieon一贯的高性能特点,还专门针对长读长数据的特性进行了优化,能够准确处理PacBio HiFi等第三代测序数据。接下来让我们一起了解一下DNAscope LongRead 所展现出的性能优势吧!
文献介绍
- 标题(英文):Sentieon DNAscope LongRead – A highly Accurate, Fast, and Efficient Pipeline for Germline Variant Calling from PacBio HiFi reads
- 标题(中文):Sentieon DNAscope LongRead – 用于 PacBio HiFi 读取的种系变异识别的高度准确、快速且高效的管道
- 发表期刊:bioRxiv
- 作者单位:Sentieon和Pacbio
- 发表年份:2022
- 文章地址:https://doi.org/10.1101/2022.06.01.494452

图1 文献介绍
短读长技术在过去二十年广泛应用,但在解析二倍体基因组的单倍型信息、高度重复序列以及临床相关基因方面存在固有局限。PacBio开发的HiFi测序技术通过CCS测序实现了长读长与高精度的结合,然而早期的长读长数据分析工具在变异检测准确性和计算效率方面仍需优化。Sentieon团队对获奖的变异检测工具DNAscope进行升级,开发了DNAscope LongRead流程。
DNAscope通过改进局部组装算法和引入机器学习模型,显著提升了变异基因分型和过滤的准确性。为更好地适应PacBio HiFi数据特点,对DNAscope进行了优化:扩大了活性区域检测范围,并针对单倍体、二倍体和未分相区域开发了特定处理模型。试验数据表明,优化措施使变异检测的召回率和精确度获得显著提升,尤其在复杂基因组区域的处理方面展现出独特优势。
测序流程
Sentieon提供一站式定制化的基因组数据分析服务,涵盖从比对到变异检测全流程;Sentieon高度优化的算法和企业级的软件工程,能够显著提升NGS数据处理的效率、准确性和可靠性。Sentieon与开源软件结果一致性达到99%以上的同时,在速度和精准度方面都优于开源,能更快更精确地将变异检测结果交付到您的手上。
PacBio HiFi reads的碱基准确率超过99.8%,主要错误来源于同聚物上下文中的插入缺失。针对这一特点,DNAscope设计了三个处理流程:首先未分相reads的重复模型校准与SNV检测;其次进行变异分相及分相区域重复模型校准;最后在分相区域的单倍体变异调用,以及在未分相区域进行二倍体检测。
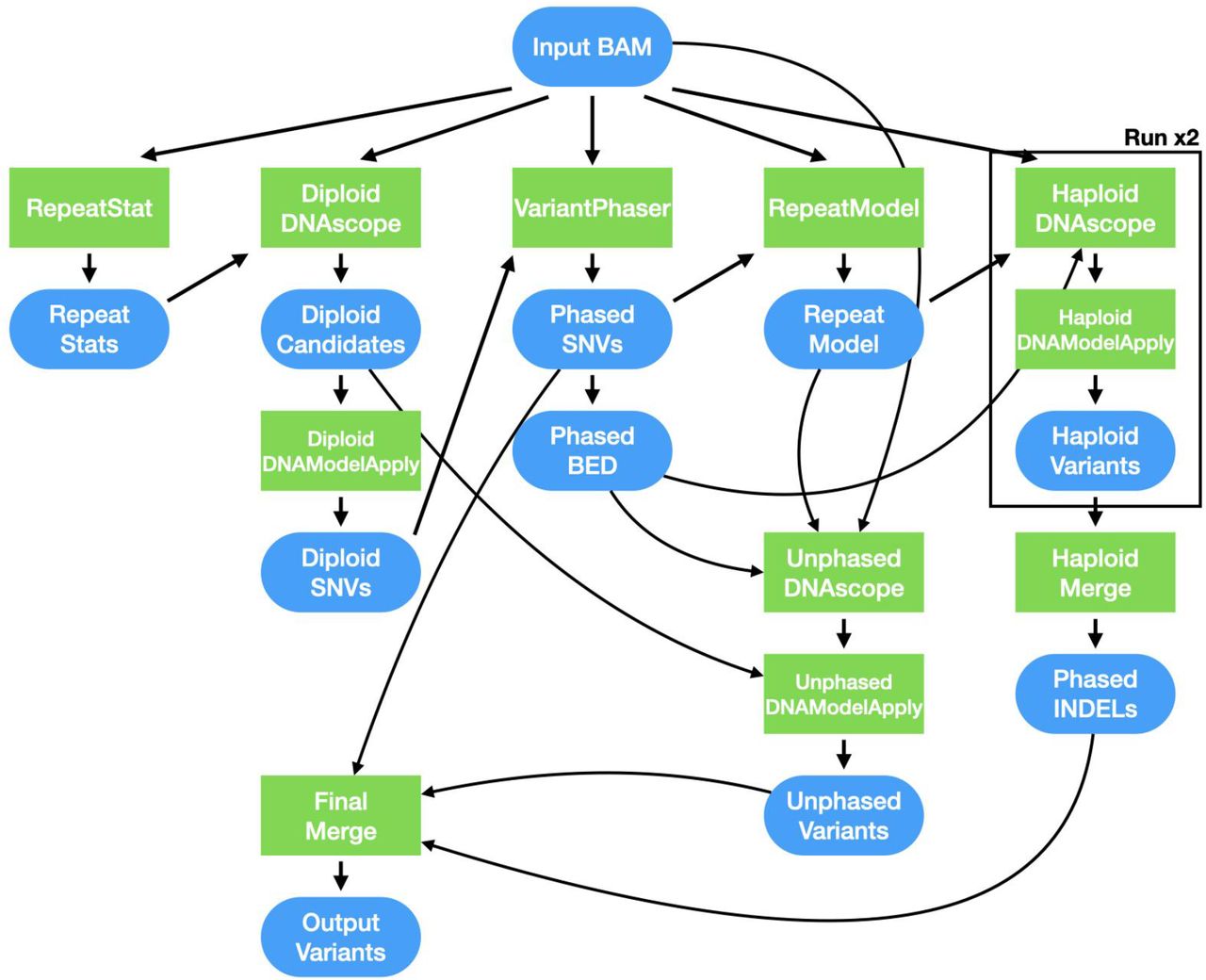
图2 DNAscope LongRead流程概述 该流程从比对的PacBio HiFi测序读段中检测胚系变异
流程可分为三个阶段:第一轮变异检测、变异分型、以及第二轮更精确的基因组分型区域变异检测(分别处理每个单倍体亲本基因组)。在第一轮和第二轮变异检测前,统计模型会根据样本的重复序列内容进行校准,以提高变异检测准确性和流程稳健性。核心变异检测流程在分型或未分型的基因组区域运行DNAscope,并使用DNAModelApply执行基于模型的变异基因分型。使用Python脚本进行VCF文件处理。
在约35x覆盖度的HG002、HG003和HG004样本测试中,DNAscope LongRead展现出了卓越的性能,在GIAB v4.2.1高置信度区域内平均检测到3,859,105个变异,错误数为9,130个,较PacBio HiFi数据流程减少15%。特别是在MHC和难映射区域的错误分别降低了55%和27%,平均F1分数达到0.9988,优于pFDA获胜流程的0.9986。充分证明了DNAscope LongRead在固定测序读段集条件下具有更高的变异检测准确率。
为验证DNAscope LongRead对新化学试剂的适应性,研究团队使用PacBio 2.2化学试剂进行测试,41x覆盖度样本数据表明,DNAscope LongRead检测到9,231个错误,总体F1分数为0.9988,与2.0版化学试剂结果(9,130个错误数,F1分数为0.9988 )相当。证明DNAscope LongRead对上游文库制备和测序变化具有良好的适应性。
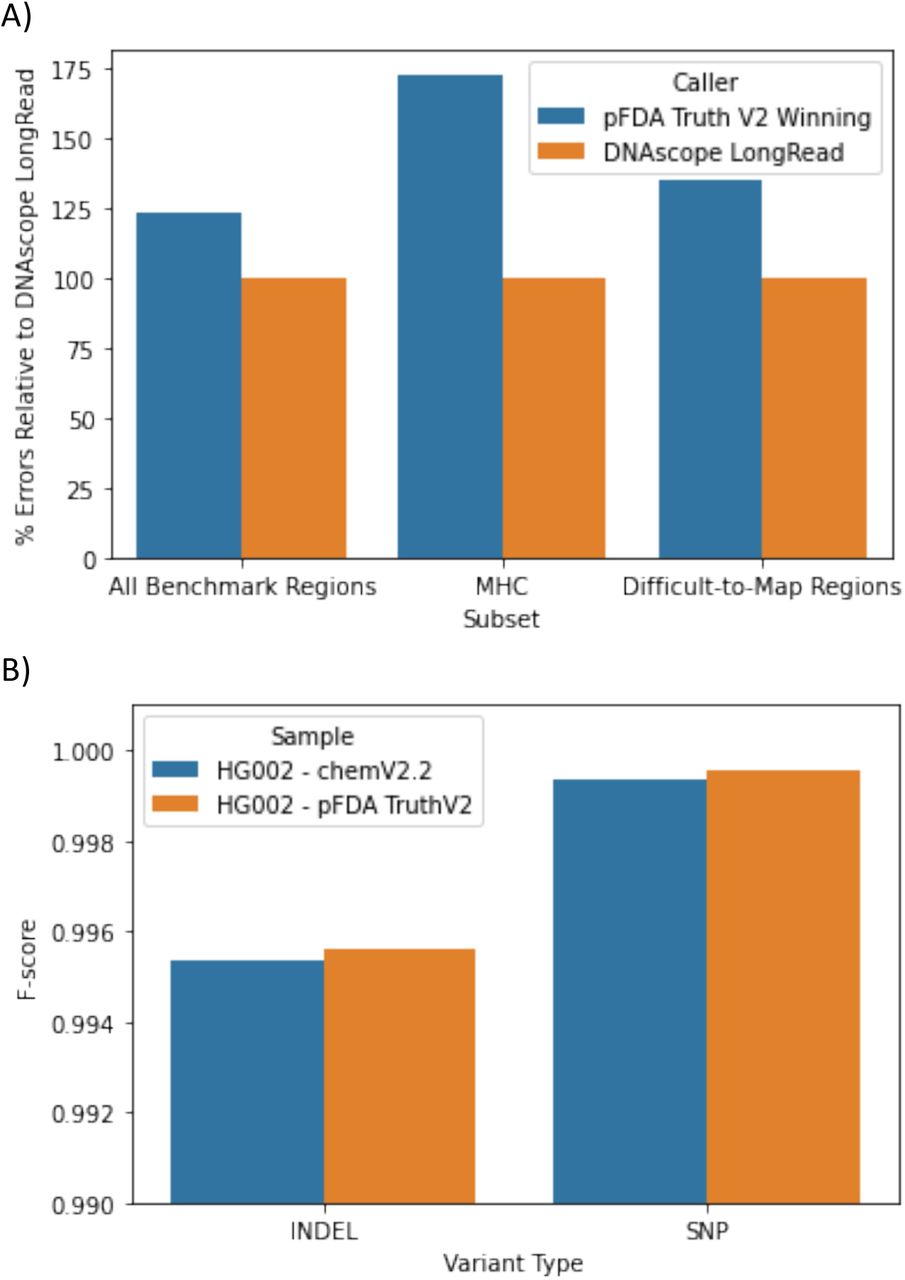
图3 PacBio HiFi样本在PrecisionFDA真实性挑战V2和新化学试剂中的变异检测准确性
(A) 相比PrecisionFDA真实性挑战V2获胜提交结果,DNAscope LongRead流程显著减少了总错误数。在MHC和难以映射的分层中,错误率尤其降低。
(B) DNAscope LongRead流程对文库制备和测序化学试剂的变化具有稳健性。使用新测序化学试剂的chemV2.2样本与模型训练中使用的HG002样本具有相当的准确性。
通过对35x HG003样本进行降采样(从5x到35x,间隔5x)研究发现,在20x-35x覆盖度范围内,非同聚物区域的SNV和indel检测表现稳定,F1分数分别仅下降0.00074和0.0049。总体indel检测受覆盖度影响较大,F1分数在相同覆盖度范围内下降了0.013。
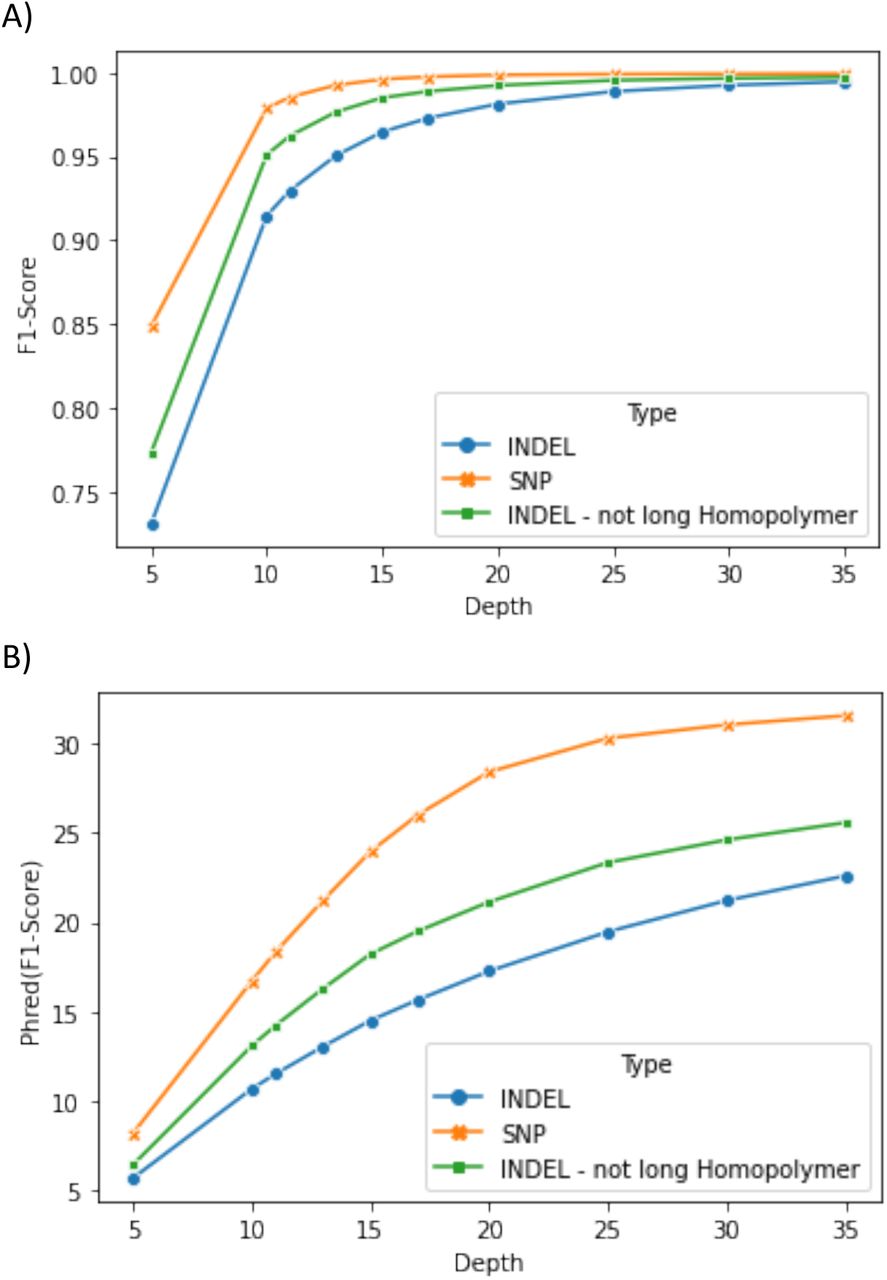
图4 读段覆盖度对DNAscope LongRead流程准确性的影响 展示了序列降采样实验中SNV(x)、indel(点)和非长同聚物区域indel(方块)的F1得分。从35x到20x覆盖度每步降低5x,在10x到20x之间添加了额外样本。在较低覆盖度下,SNV准确性比indel准确性更稳定。数据以两种视图显示,以突出较高覆盖度下的准确性提升:(A)F1得分和(B)phred标度F1得分。
为评估DNAscope LongRead在GIAB基准区域之外的性能,研究团队使用HG002样本与CMRG基准数据集进行比对。结果显示DNAscope LongRead产生了846个总错误。在KMT2C区域表现尤其出色,仅产生62个假阳性,优于DeepVariant-HiFi产生的277个假阳性。证明DNAscope LongRead不仅能扩展到GIAB v4.2.1基准区域之外,还能在独立创建的真实数据集上表现良好。
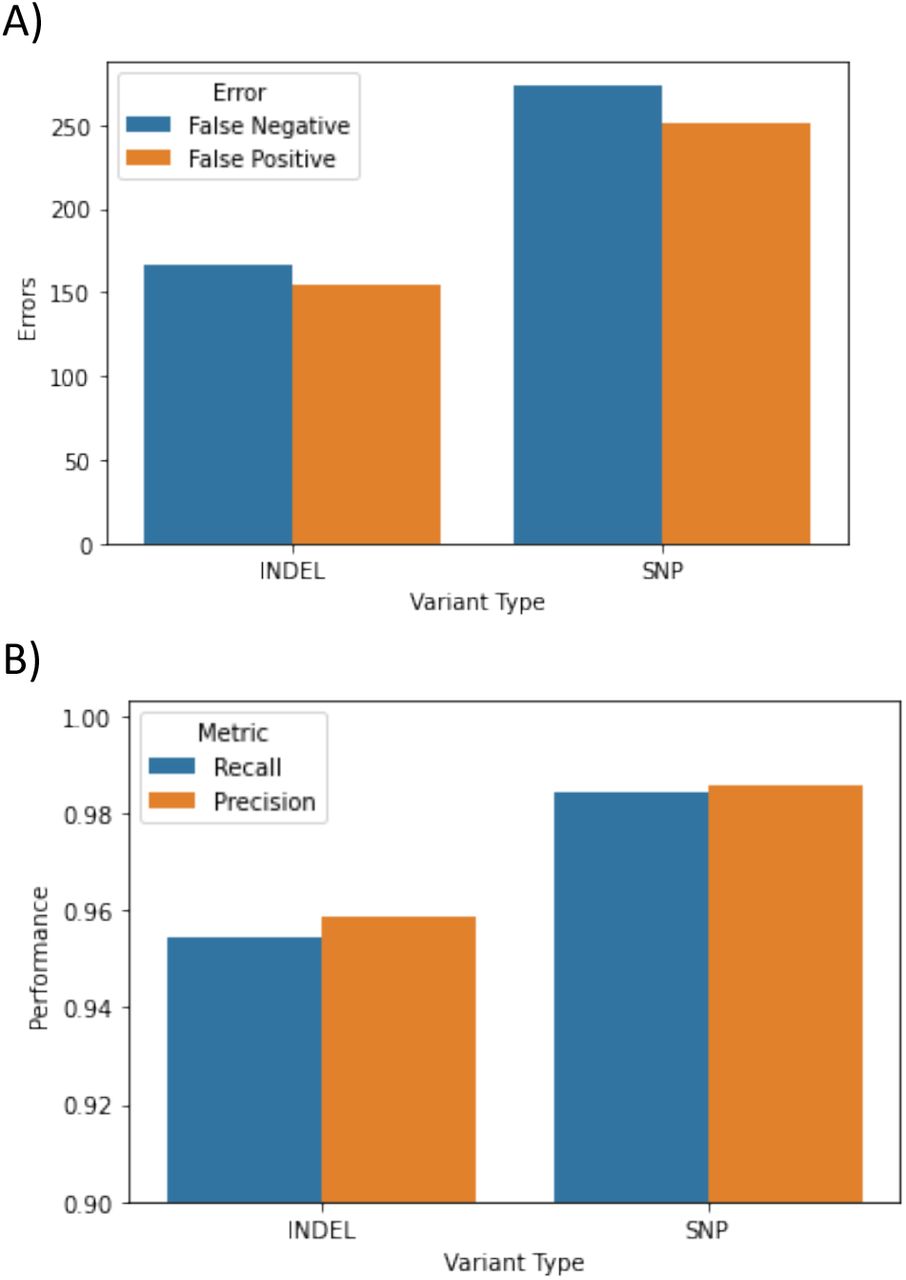
图5 在具有医学相关性的挑战性基因基准测试中对PrecisionFDA Truth V2样本的准确性评估 使用具有医学相关性的挑战性基因基准(CMRG)评估DNAscope LongRead流程的准确性。CRMG基准使用基于组装的方法扩展到v4.2.1 GIAB基准区域之外的更具挑战性区域。(A)错误数量。(B)精确度和召回率。
从计算资源利用效率来看,DNAscope LongRead在30x样本处理中需要120.3核心小时,总运行时间1.71-4.52小时之间。内存使用在8.39-14.8 GB范围内,与样本覆盖度呈正相关。,展现出DNAscope LongRead良好的性价比和资源利用率。
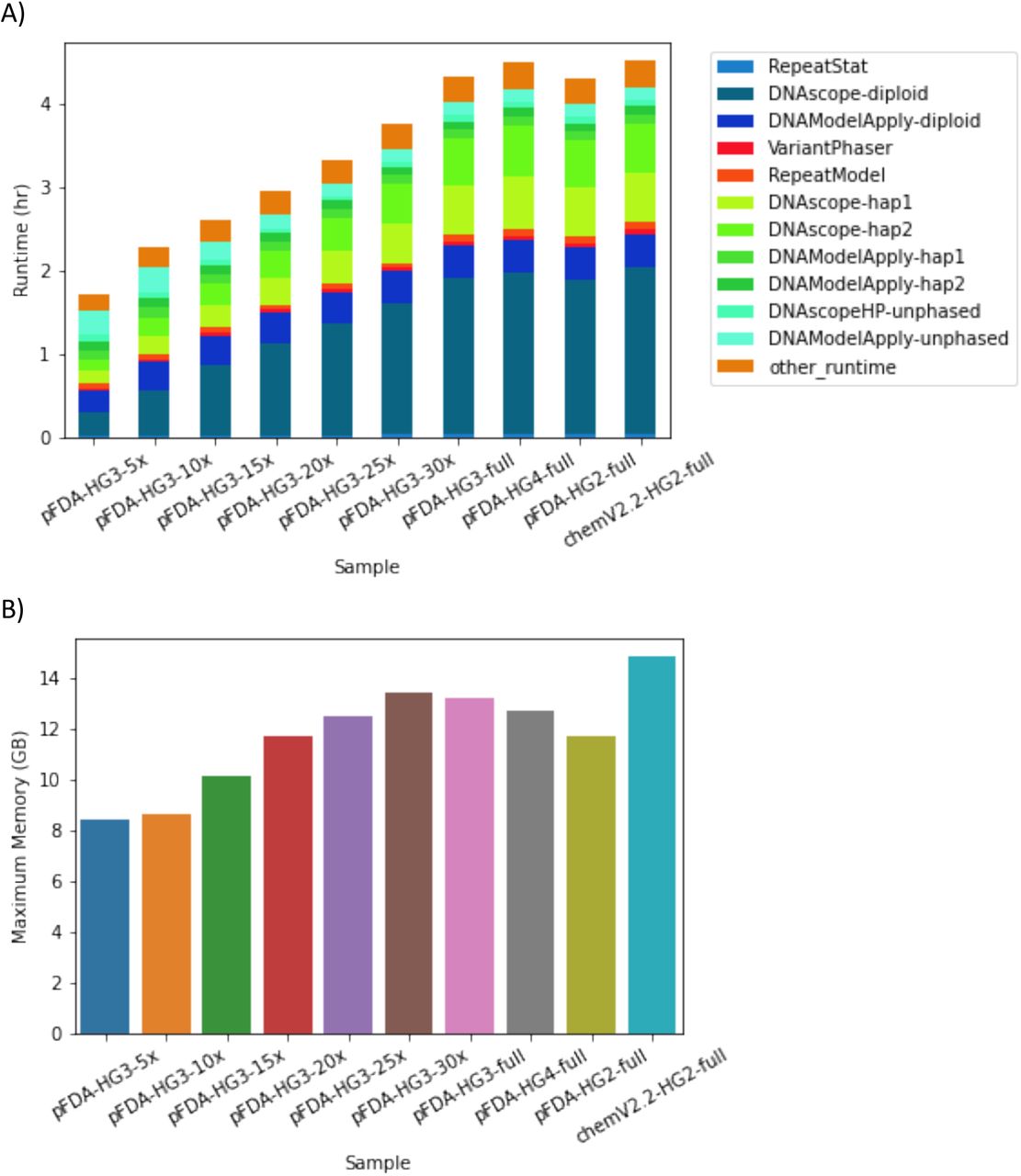
图6 DNAscope LongRead流程的运行时间和最大内存使用
(A) 在32核Intel® Xeon®服务器上各流程阶段的运行时间。阶段按时间顺序排列。第一轮变异检测阶段为深蓝色,变异分型阶段为红色,第二轮变异检测阶段为绿色。"其他运行时间"为简短的VCF处理命令。
(B) 各样本的最大内存使用。高覆盖度样本在变异检测时通常需要更多内存。
Sentieon 软件团队拥有丰富的软件开发及算法优化工程经验,致力于解决生物数据分析中的速度与准确度瓶颈,为来自于分子诊断、药物研发、临床医疗、人群队列、动植物等多个领域的合作伙伴提供高效精准的软件解决方案,共同推动基因技术的发展。截至 2023 年 3 月份,Sentieon 已经在全球范围内为 1300+用户提供服务,被世界一级影响因子刊物如 NEJM、Cell、Nature 等广泛引用,引用次数超过 700 篇。此外,Sentieon 连续数年摘得了 Precision FDA、Dream Challenges 等多个权威评比的桂冠,在业内获得广泛认可。
总结
DNAscope LongRead在处理PacBio HiFi数据的胚系变异检测中实现了在精准性方面、鲁棒性方面,效能方面三大突破,Sentieon未来将进一步优化同聚物区域检测,并探索更广泛的应用场景。
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








