一 问题描述
众所周知,DNA 序列是一个只包含 A、C、T 和 G 的序列,分析 DNA 序列的一个片段非常有用,例如,如果动物的 DNA 序列包含片段 ATC,那么这可能意味着动物可能患有遗传病。到目前为止,科学家已经发现了几个这样的片段,问题是一个物种有多少种 DNA 序列不包含这些片段。
假设一个物种的 DNA 序列是由 A、C、T 和 G 组成的序列,并且序列长度是一个给定的整数 n。
二 输入和输出
1 输入
第一行包含两个整数 m (0<=m<=10) , n (1<=n<=2000000000) 。这里,m 是遗传病片段的数量,n 是序列的长度。
接下来的 M 行每行包含一个 DNA 遗传病片段,这些片段的长度不大于 10。
2 输出
一个整数,DNA 序列数,mod 100000。
三 输入和输出样例
1 输入样例
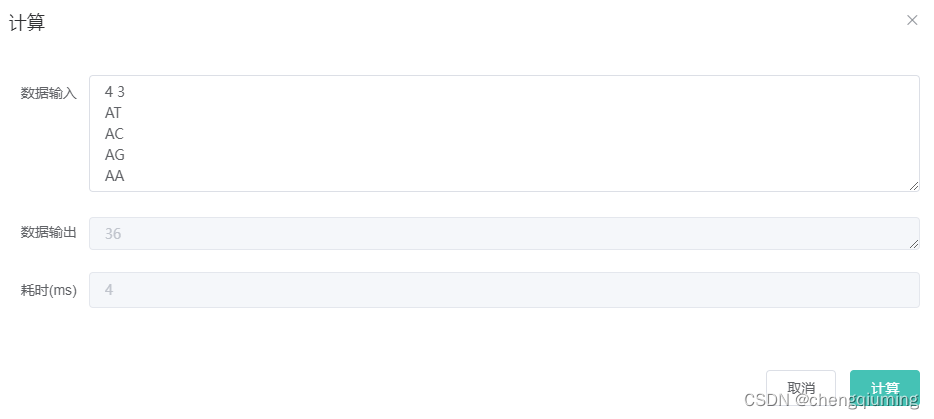
4 3
AT
AC
AG
AA
2 输出样例
36
四 分析和设计
1 分析
有 m 种 DNA 序列是有疾病的,问有多少种长度为 n 的 DNA 序列不包含任何一种有疾病的 DNA 序列。(仅含A、T、C、G 四个字符)。
样例 m = 4,n=3
AA AT AC AG
答案为 36,表示有 36 种长度为 3 的序列可以不包含疾病。
下图例子的病毒序列是
ACG C
构建 trie 图后如下图所示,从每个节点出发都有 4 条边{ A、T、C、G }
从状态 0 出发走 1 步有 4 种走法。
走 A 到状态 1:安全
走 C 到状态 4:危险
走 T 到状态 0:安全
走 G 到状态 0:安全
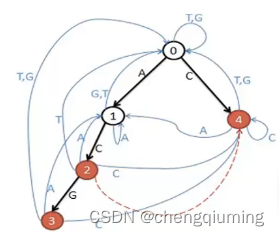
所以当 n = 1 时,答案就是 3。
当 n =2 时,就是从状态 0 出发走 2 步,就形成一个长度为 2 的字符串,只要路径上没有经过危险节点,有几种走法,那么答案就是几种。依次类推走 n 步就形成长度为 n 的字符串。
建立 trie 图的邻接矩阵 M
0 1 2 3 4
0 2 1 0 0 1
1 2 1 1 0 0
2 1 1 0 1 1
3 2 1 0 0 1
4 2 1 0 0 1
M[i,j] 表示从节点 i 到 j 只走一步有几种走法。
那么 M 的 n 次幂就表示从节点 i 到 j 走 n 步有几种走法。
注意:危险节点要去掉,也就是去掉危险节点的行和列。节点 3 和 节点 4 是单词结尾,所以危险,节点 2 的 fail 指针指向 4,当匹配 “AC”时也就匹配了 “C”,所以 2 也是危险的。
矩阵变成 M
2 1
2 1
计算 M[][] 的 n 次幂,然后对 M[0,i] mod 100000 就是答案。
由于 n 很大,可以使用二分来计算矩阵的幂。
2 设计
DNA 序列只包含 A、G、C、T 4种字母,给定 m 个 DNA 遗传病片段,求有多少个长度为 n 的 DNA 序列不包含遗传病片段。
算法步骤:
a 将遗传病片段构建字典树。
b 构建 AC 自动机。需要特别注意,如果当前结点的失败指针 end 有结束标记,当前结点的也要标记结束。
c 构建邻接矩阵。对所有未标记的结点重新编号,根据 AC 自动机构建邻接矩阵。
d 求解矩阵的 n 次幂。可用矩阵快速幂求解。
五 代码
package com.platform.modules.alg.alglib.poj2778;
import java.util.LinkedList;
import java.util.Queue;
public class Poj2778 {
public static int maxn = 105;
public static int K = 4; // 代表 A、C、T 和 G
public static int MOD = 100000;
public static int root; // 字典树的根
public static int L; // 字典树节点个数
public String output = "";
ACAutomata ac = new ACAutomata();
// 矩阵乘法
public static mat mul(mat A, mat B) {
mat C = new mat();
for (int i = 0; i < L; i++)
for (int j = 0; j < L; j++)
for (int k = 0; k < L; k++)
C.a[i][j] = (C.a[i][j] + A.a[i][k] * B.a[k][j]) % MOD;
return C;
}
// A^n
public static mat pow(mat A, int n) {
mat ans = new mat();
for (int i = 0; i < L; i++)
ans.a[i][i] = 1; // 单位矩阵
while (n > 0) {
if ((n & 1) > 0)
ans = mul(ans, A);
A = mul(A, A);
n >>= 1;
}
return ans;
}
public String cal(String input) {
int m; // 病毒数量
int n; // DNA 序列长度
String[] line = input.split("\n");
String[] num = line[0].split(" ");
m = Integer.parseInt(num[0]);
n = Integer.parseInt(num[1]);
int i = 1;
ac.init();
while (m-- > 0) {
ac.insert(line[i++]);
}
ac.build();
output = ac.query(n) + "";
return output;
}
}
class mat {
int a[][] = new int[Poj2778.maxn][Poj2778.maxn];
}
class ACAutomata {
int next[][] = new int[Poj2778.maxn][Poj2778.K];
int fail[] = new int[Poj2778.maxn];
int end[] = new int[Poj2778.maxn]; // 结束标志
int id[] = new int[Poj2778.maxn];
int idx(char ch) { // 转化数字
switch (ch) {
case 'A':
return 0;
case 'C':
return 1;
case 'T':
return 2;
case 'G':
return 3;
}
return -1;
}
int newNode() { // 新建结点
for (int i = 0; i < Poj2778.K; i++)
next[Poj2778.L][i] = -1;
end[Poj2778.L] = 0;
return Poj2778.L++; // 节点个数新增1
}
void init() { // 初始化
Poj2778.L = 0;
Poj2778.root = newNode(); // 根是 0 号节点
}
void insert(String s) { // 插入一个遗传病片段
int len = s.length();
int p = Poj2778.root;
for (int i = 0; i < len; i++) {
int ch = idx(s.charAt(i));
if (next[p][ch] == -1)
next[p][ch] = newNode();
p = next[p][ch];
}
end[p]++;
}
// 构建AC自动机
void build() {
Queue<Integer> Q = new LinkedList<>(); // 队列,BFS使用
fail[Poj2778.root] = Poj2778.root;
for (int i = 0; i < Poj2778.K; i++) {
if (next[Poj2778.root][i] == -1) {
next[Poj2778.root][i] = Poj2778.root;
} else {
fail[next[Poj2778.root][i]] = Poj2778.root;
Q.add(next[Poj2778.root][i]);
}
}
while (!Q.isEmpty()) {
int now = Q.peek();
Q.poll();
if (end[fail[now]] > 0)
end[now]++; // 重要!!如果当前结点的失败指针end有结束标记,当前结点的 end++
for (int i = 0; i < Poj2778.K; i++) {
if (next[now][i] != -1) {
fail[next[now][i]] = next[fail[now]][i];
Q.add(next[now][i]);
} else
next[now][i] = next[fail[now]][i];
}
}
}
int query(int n) {
mat F = new mat();
int ids = 0;
for (int i = 0; i < id.length; i++) {
id[i] = -1;
}
for (int i = 0; i < Poj2778.L; i++) // 对未标记的结点重新编号
if (end[i] == 0)
id[i] = ids++;
for (int u = 0; u < Poj2778.L; u++) {
if (end[u] > 0) continue;
for (int j = 0; j < Poj2778.K; j++) {
int v = next[u][j];
if (end[v] == 0)
F.a[id[u]][id[v]]++;
}
}
Poj2778.L = ids;
F = Poj2778.pow(F, n);
int res = 0;
for (int i = 0; i < Poj2778.L; i++)
res = (res + F.a[0][i]) % Poj2778.MOD;
return res;
}
}六 测试






















 1255
1255











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








