DCA(Decision Curve Analysis)临床决策曲线是一种用于评价诊断模型诊断准确性的方法,在2006年由AndrewVickers博士创建,我们通常判断一个疾病喜欢使用ROC曲线的AUC值来判定模型的准确性,但ROC曲线通常是通过特异度和敏感度来评价,实际临床中我们还应该考虑,假阳性和假阴性对病人带来的影响,因此在DCA曲线中引入了阈概率和净获益的概念。
在既往文章《R语言手动绘制随机森林决策曲线》中,咱们已经介绍了手动绘制随机森林决策曲线,如今我已经把代码整合到了我写的ggscidca包上,咱们可以通过ggscidca包快速绘制随机森林决策曲线,下面我来演示一下
咱们使用下面代码安装ggscidca包,安装了旧版本的在安装一次就可以更新到新版本
install.packages("ggscidca")
安装后以后咱们导入R包和数据,ggscidca绘制决策曲线支持的是randomForest包生成的随机森林,目前还不支持随机森林生存分析,所以咱们要导入randomForest包。数据是使用ggscidca 包自带的LIRI数据。
library(ggscidca)
library(survival)
library(reshape2)
library(ggplot2)
library(randomForest)
LIRI<-LIRI
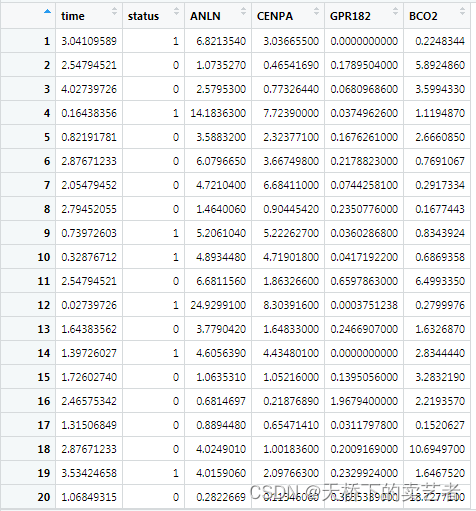
Status是结局变量,其他是一些协变量,这个数据只是演示用,没有什么实际意义。咱们先对数据进行随机拆分
set.seed(1)
index <- sample(2,nrow(LIRI),replace = TRUE,prob=c(0.7,0.3))
traindata <- LIRI[index==1,]
testdata <- LIRI[index==2,]
拆分好数据后,先把结果变量转成因子
traindata$status<-as.factor(traindata$status)
咱们先用训练集建模,具体参数不介绍了,有兴趣看我相关的文章,你需要什么调参的话也是在这一步进行
fit<-randomForest(status ~ANLN+CENPA+GPR182+BCO2 ,data=traindata,ntree=500,
important=TRUE,proximity=TRUE)
建立模型后,咱们先绘制训练集决策曲线
scidca(fit,newdata = traindata)
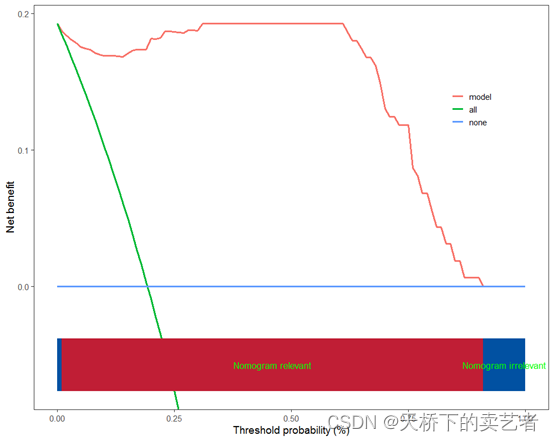
绘制验证集决策曲线
scidca(fit,newdata = testdata )

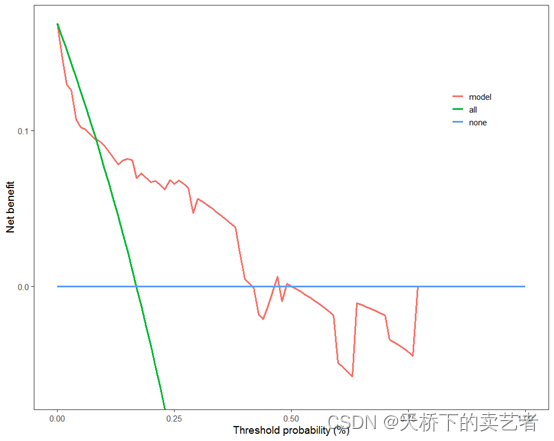
如果只是想要单纯的决策曲线,也是可以的
scidca(fit,newdata = testdata,colbar = F)
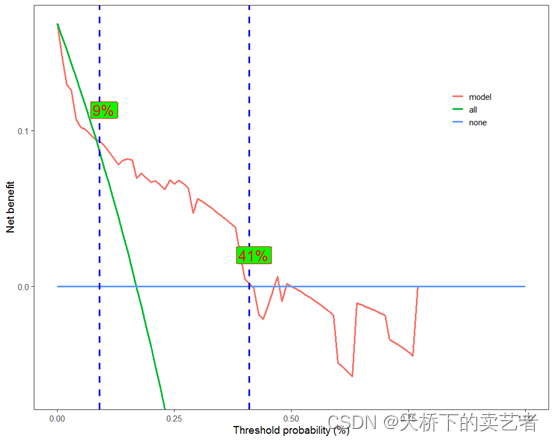
应粉丝的要求在,在新版本ggscidca包添加了虚线功能,你可以在阈值的两点添加虚线,
scidca(fit,newdata = testdata ,threshold.line = T)
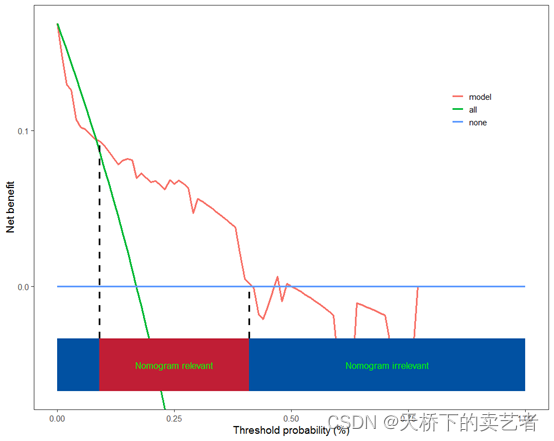
在文章Nomograms in Oncology – More than Meets the Eye中还报道了具体的阈值数,
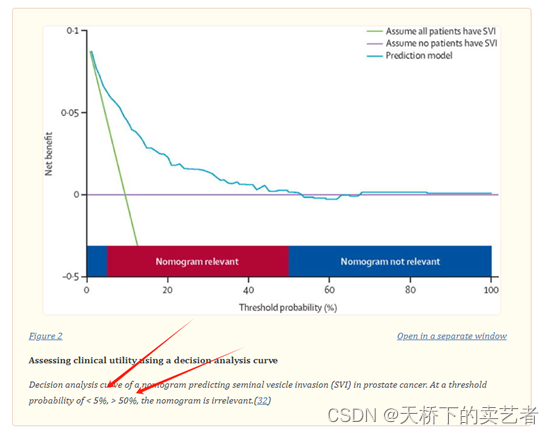
ggscidca包也提供了阈值数的功能,
scidca(fit,newdata = testdata ,threshold.line = T,threshold.text=T)
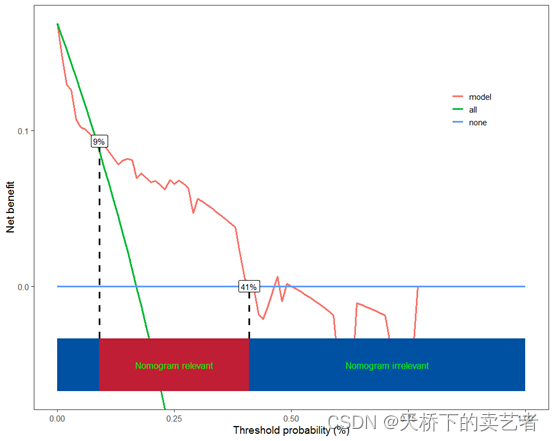
表明预测模型在9%到41%这个阈值是获益的。这两个数值的大小和位置是可以移动的
scidca(fit,newdata = testdata ,threshold.line = T,threshold.text=T,nudge_x = 0.01,nudge_y = 0.02)
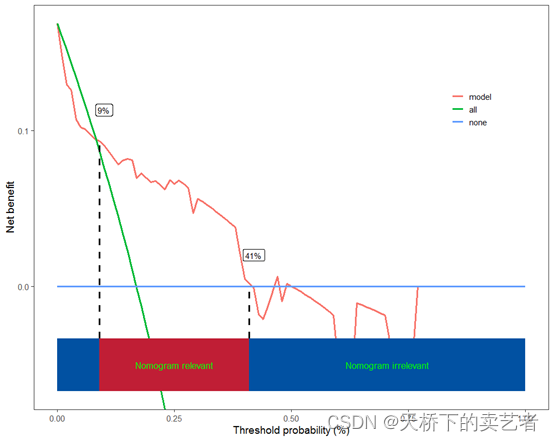
更改字体大小,颜色和背景
scidca(fit,newdata = testdata ,threshold.line = T,threshold.text=T,nudge_x = 0.01,nudge_y = 0.02,
po.text.size=7,po.text.col="red",po.text.fill="green")
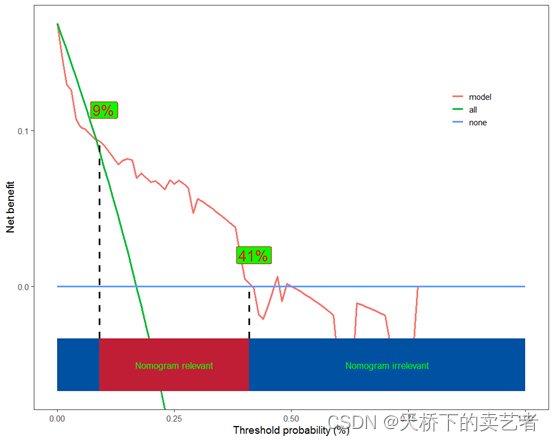
绘制普通的决策曲线,也是可以添加阈值数的
scidca(fit,newdata = testdata,colbar = F,threshold.text=T,nudge_x = 0.01,nudge_y = 0.02,
po.text.size=7,po.text.col="red",po.text.fill="green")
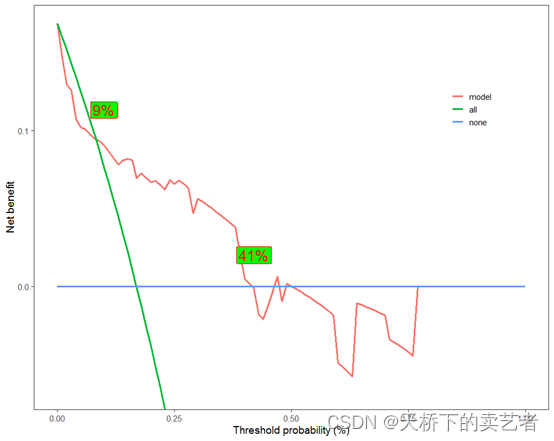
加上线条也是可以的,线条还可以改颜色,线型和大小
scidca(fit,newdata = testdata,colbar = F,threshold.text=T,nudge_x = 0.01,nudge_y = 0.02,
po.text.size=7,po.text.col="red",po.text.fill="green",threshold.line = T,
threshold.linecol="blue")
这个线条加上去好像不是很美观,目前我还没想好怎么优化,如果你有好的建议,也可以私信我。
最后介绍一个手动调控功能,我重新导入一个数据
bc<-Breastcancer
#分类变量转成因子
bc$histgrad<-as.factor(bc$histgrad)
bc$er<-as.factor(bc$er)
bc$pr<-as.factor(bc$pr)
bc$ln_yesno<-as.factor(bc$ln_yesno)
#建立模型
f1<-coxph(Surv(time,status)~er+histgrad+pr+age+ln_yesno,bc)
f2<-coxph(Surv(time,status)~er+histgrad+ln_yesno,bc)
f3<-coxph(Surv(time,status)~ln_yesno,bc)
先做个生存分析的决策曲线
scidca(f2,threshold.line = T,threshold.text=T)
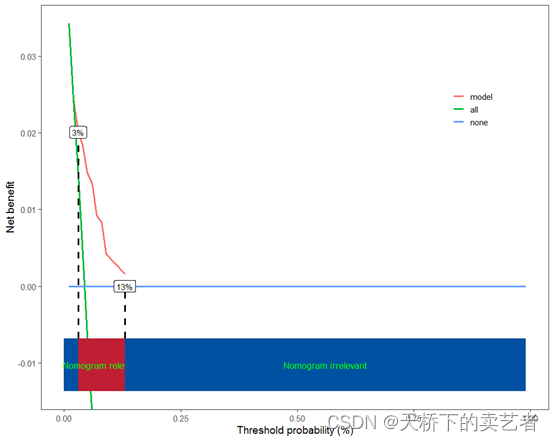
上图看着好像还行,没见啥问题,咱们限制一下X轴的范围
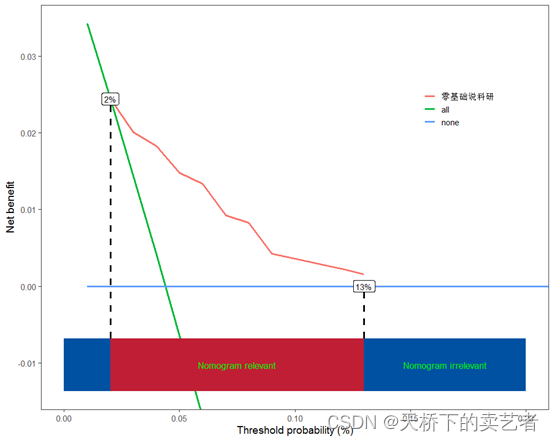
scidca(f2,modelnames = "零基础说科研",xstop=0.20,threshold.line = T,threshold.text=T)
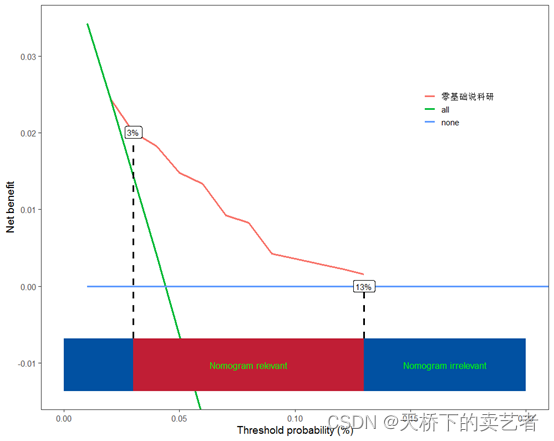
这样问题就来了,虚线其实没有对准,因为X轴很大的时候,虚线向下角度不明显,限制范围后就明显了,这种情况咱们可以手动调整一下,需要liftpec参数,咱们向左挪移一下
scidca(f2,modelnames = "零基础说科研",xstop=0.20,threshold.line = T,threshold.text=T,
liftpec=-1)
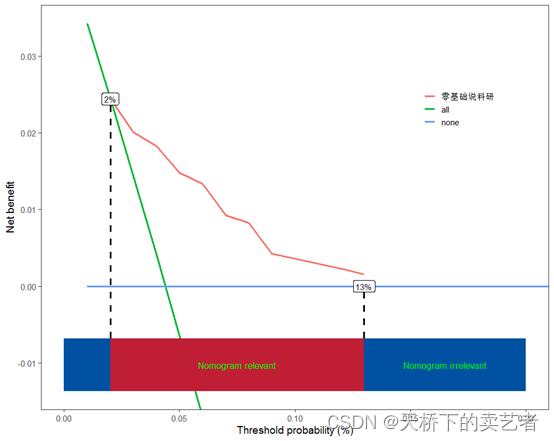
这样就对上了。有报错或者意见或建议,欢迎公众号私信我。

























 648
648











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










