一、前言
本篇文章利用CSP和LDA进行运动想象二分类问题进行分类任务,其中有较为详细的运动想象脑电数据的预处理部分以及事件的选取和方差最大化等内容。因本人能力可能有限,在写本篇文章中也难免会出些错误,本次文章也是对这一段时间内学习的总结,如有错误的地方欢迎评论区指正。
数据集:
本次数据集利用BCI competition IV数据集可从官网下载获取
百度网盘自取:
链接:https://pan.baidu.com/s/15u3J6JrQRyjknvKJB_fiyw
提取码:2424
GitHub自取:
其中本篇文章的源码为EEG_MI_ML.ipynb,数据集为BCICIV_calib_ds1b.mat文件
二、正文
1.导包读取文件
import numpy as np
import scipy.io
from matplotlib import mlab
import matplotlib.pyplot as plt
import scipy.signal
import mne# mat文件中的数据会导致许多维度的复杂 从而使代码复杂
m = scipy.io.loadmat('C:\\Users\\24242\\Desktop\\AI_Reference\\data_bag\\BCICIV\\BCICIV_calib_ds1b.mat',struct_as_record=True)2.数据分析与数据处理
打印m可得
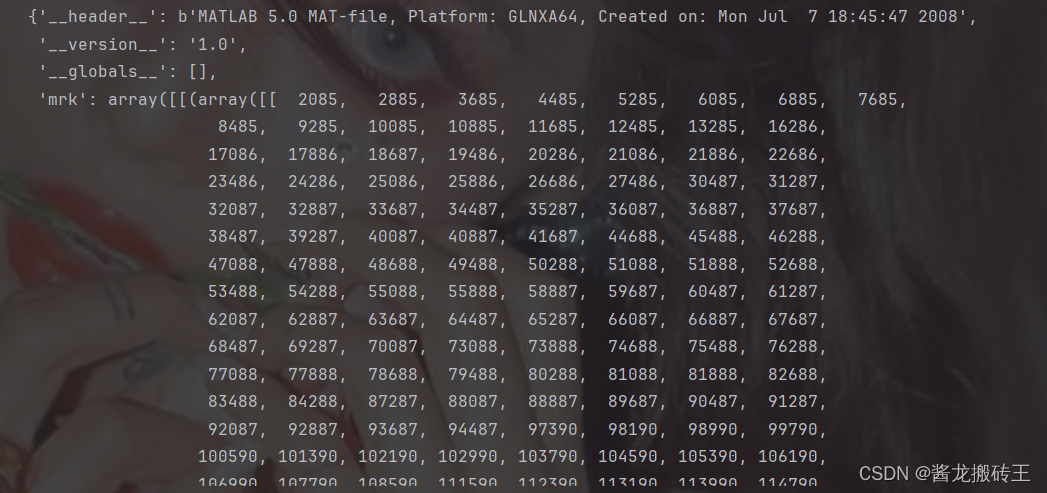
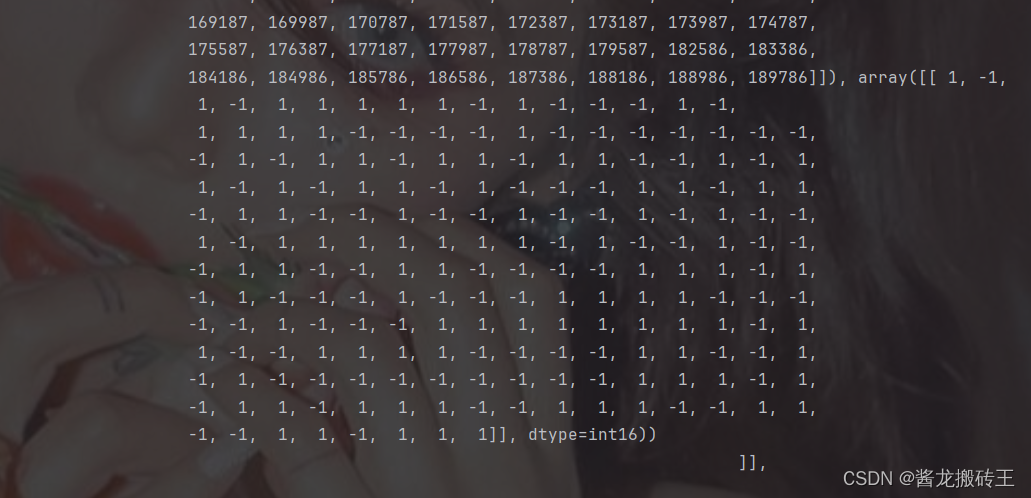
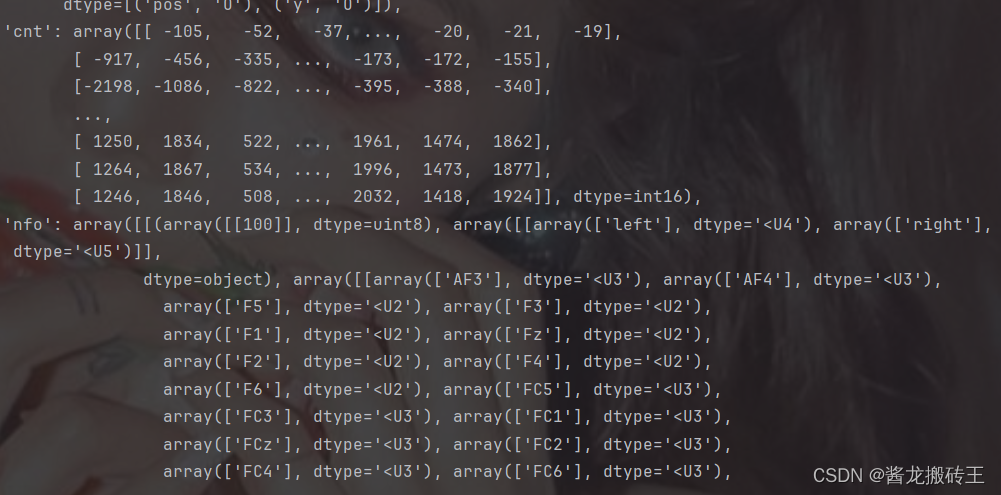
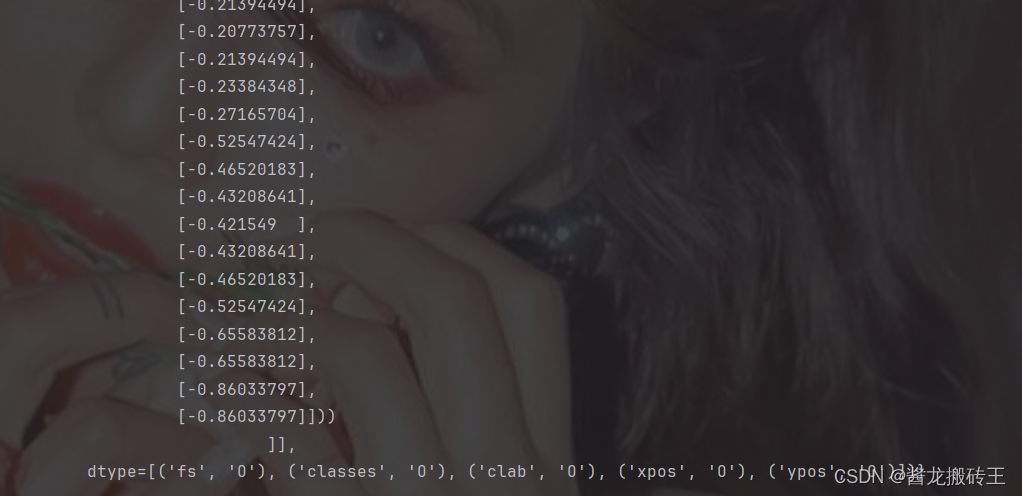
请记住上面这几张图,从上往下我们一一标为图一、二、三、四。
上面是部分关键数据的截图,我们从上面不难发现,m的数据是异常复杂且维度众多的,一不小心我们就会搞混维度,而这才是分析本次数据的关键,不同的维度对应着不同的数据,比如采样通道、EEGdata、采样率等一系列关键数据
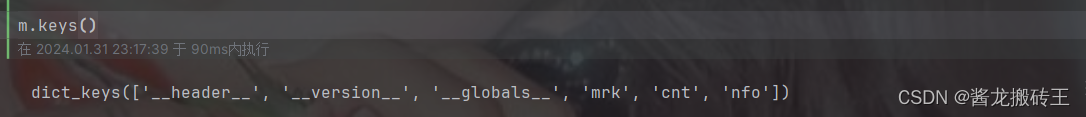
在获取m的属性时,我们发现只有'mrk','cnt','nfo'三个键
我们通过观察可以发现'cnt'属性中有一个矩阵存着我们EEGdata的,此时我们就有一个疑惑了,为什么'cnt'中存在的是我们EEGdata?我看'mrk'中的数据也像呀?仔细观察一下'mrk'中的数据特征,这不是和我们的mark点很像,而'cnt'中的数据有正有负,是不是对应着我们EEGdata。好了,你还会发现'mrk'中还存着一个数组,里面的数据全是[-1,1],这是不是和我们的标签很像。这个时候我们就焕然大悟了,这个'mrk'中包含的就是mark点和其所对应的label。
既然EEGdata和事件的mark、label被我们找到了,那么我们还缺什么呢?没错,缺的东西还挺多的,像这个文件中包含着哪些类别、采样率、通道等数据,那么接下来就要分析令我们头晕的'nfo'文件了。
我们看'nfo',一眼却看到了一个数组中存着100这个数字,那根据经验判断,这个是采样率,那么我们该怎么获取呢?维度这么高,看着我就头晕!
# 获取采样率
sample_rate = m['nfo']['fs'][0][0][0][0]
sample_rate大家可以观察一下,这个第一个维度是不是一个超级超级超级大的array数组,这个数组中存着很多很多的数据
比如'fs':采样率 'classes':类别 ‘clab':通道 ,而我们静下心来看发现采样率就在第四个维度当中,所以我们利用上面的代码进行获取,其他数据获取同理,微笑着面对它,一个一个静心观察。
# 获取EEGdata
EEG = m['cnt'].T
EEG# 通道和样本个数
n_channels, n_samples = EEG.shapechannel_name = [s[0] for s in m['nfo']['clab'][0][0][0]]
channel_name获取通道命就有点复杂了我们可以观察到,每个通道名都是一个二维数组的第一个元素。
# 提取事件对应发生的时间
event_onsets = m['mrk'][0][0][0]
# 提取事件的编码 每个类别都对应了其编码 1代表着左手运动想象 -1代表着右手
event_codes = m['mrk'][0][0][1]# 总有两百个数据 便于我们对连续的数据集事件编码进行分类
event_codes.shape# 创造一个0填充的矩阵
labels = np.zeros((1,n_samples),int)
labels上面的label是用采样个数进行0填充的,为什么要这样做呢,就是为什么给事件打上mark
# 将事件发生对应的事件进行标记编码 理解为当作是一个trigger触发条件
labels[0,event_onsets] = event_codes# 提取事件的名称 二分类任务
cl_lab = [s[0] for s in m['nfo']['classes'][0][0][0]]
cl_lab_left = cl_lab[0]
cl_lab_foot = cl_lab[1]注意了,上面代码是foot其实是right,因为一开始打算做手脚分类的,但是后面还是做了左右手运动想象分类,代码没有改过来,大家注意一下就行,其中类别的提取是和通道提取思想一样的。
# 到底有多少类
n_classes = len(cl_lab)
# 到底有多少个事件
nevents = len(event_onsets)打印处我们现在获取的所有数据
# 目前我们得到了我们基本需要的数据了
print('Shape of EEG:',EEG.shape)
print('Sample rate:',sample_rate)
print('Number of channels:',n_channels)
print('Channel names:',channel_name)
print('Number of events:',len(event_onsets))
print('Event codes:',np.unique(event_codes))
print('Number of classes:',n_classes)
print('Number of events:',nevents)3.绘制PSD频谱图
# 将EEG从连续的信号中分离出来
trials = {}
# 定义时间窗口 这里是以0.5-2.5秒为单位的时间窗口
window = np.arange(int(0.5*sample_rate),int(2.5*sample_rate))# 在时间窗口上的样本 就是为200个数据点
nsamples = len(window)for cl, code in zip(cl_lab,np.unique(event_codes)):
cl_onsets = event_onsets[event_codes == code]
trials[cl] = np.zeros((n_channels,nsamples,len(cl_onsets)))
for i, onset in enumerate(cl_onsets):
trials[cl][:,:,i] = EEG[:,window+onset]上面是获取指定事件窗口中的采样样本
trials[cl_lab_foot].shape,trials[cl_lab_left].shape
trials# 进行频谱图的绘制
def get_psd(trials):
"""
计算出实验的PSD
:param trials: 3d-array(channels,samples,trials)
:return: trials_PSD:3d-array(channels,sample,trials)
freqs:list of floats
"""
ntrials = trials.shape[2]
trials_PSD = np.zeros((n_channels,101,ntrials))
for trial in range(ntrials):
for ch in range(n_channels):
# 计算PSD 每个通道的
(PSD,freqs) = mlab.psd(trials[ch,:,trial],NFFT=int(nsamples),Fs=sample_rate)
trials_PSD[ch,:,trial] = PSD.ravel()
return trials_PSD,freqs频谱图的绘制大家可以去看看matplotlib文档中的mlab,本文不加赘述(绝对不是作者食力有限)。
# 绘制PSD图
def plot_psd(trials_PSD,freqs,chan_ind,chan_lab=None,maxy=None):
"""
plot PSD data
:param trials_PSD:The PSD Data as returned by get_psd()
:param freqs: list of float as returned by get_psd()
:param chan_ind: The indices of the channels to plot
:param chan_lab: (optional)list of names for each channel
:param maxy: (optional)Limit the y-axis to this value
:return: None
"""
plt.figure(figsize=(12,5))
nchans = len(chan_ind)
nrows = np.ceil(nchans/3)
ncols = min(3,nchans)
# Enumerate所有的通道
for i,ch in enumerate(chan_ind):
plt.subplot(int(nrows),ncols,i+1)
for cl in trials.keys():
plt.plot(freqs,np.mean(trials_PSD[cl][ch,:,:],axis=-1),label=cl)
# 所有绘图的下方装饰
plt.xlim(1,30)
if maxy != None:
plt.ylim(0,maxy)
else:
plt.title(chan_lab[i])
plt.grid()
plt.xlabel('Frequency (HZ)')
if chan_lab == None:
plt.title('Channel %d' % (ch+1))
else:
plt.title(chan_lab[i])
plt.legend()
plt.tight_layout()
plt.show()psd_left,freqs = get_psd(trials[cl_lab_left])
psd_foot,freqs = get_psd(trials[cl_lab_foot])
trials_PSD = {cl_lab_left:psd_left,cl_lab_foot:psd_foot}获取左手右手的trails_PSD进行绘图准备
为什么循着Cz C3 C4 这三个通道呢,因为这三个通道对研究运动想象更为有意义
绘制出三个通道的PSD频谱图
# 我们选择Cz C3 C4三个通道进行绘制 C3代表着左边 C4代表着右边 Cz代表着中间
plot_psd(trials_PSD,freqs,[channel_name.index(ch) for ch in ['C3','Cz','C4']],chan_lab=['left','center','foot'],maxy=500)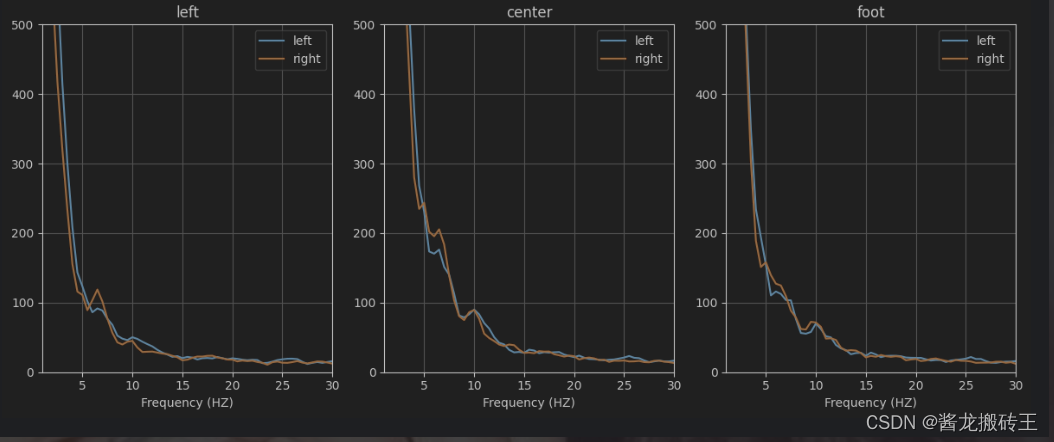 我们都知道左手运动是右脑 右手运动是左脑 脚步运动是由脑部的中央片区控制的
我们都知道左手运动是右脑 右手运动是左脑 脚步运动是由脑部的中央片区控制的
小剧场
利用python的mne包创建一个raw对象玩一玩,试试就逝世。
# 尝试创建一下raw对象
info = mne.create_info(ch_names=channel_name,sfreq=100,ch_types='eeg')
raw = mne.io.RawArray(EEG,info)
print(raw)raw.plot_psd()
玩完之后我们进行我们的滤波操作了,在mne中我们滤波操作可能只需要一个filter就能搞定,但是我们如果自己实现的话就有一些麻烦了,涉及scipy中的signal去构建滤波器,同理查看scipy官方文档了解详细用法。
# 创建带通滤波器
def bandpass(trials, lo, hi, sample_rate):
# IIRfilter滤波器设置 第一个参数为过滤器顺序 第二个参数为临界的标量 为半个周期所以是采样率/2 既是sample_rate/2
a,b = scipy.signal.iirfilter(6,[lo/(sample_rate/2.0),hi/(sample_rate/2.0)])
ntrials = trials.shape[2]
trials_filt = np.zeros((n_channels,nsamples,ntrials))
for i in range(ntrials):
trials_filt[:,:,i] = scipy.signal.filtfilt(a,b,trials[:,:,i],axis=1)
return trials_filt# 进行滤波处理 就像mne包中Epoch.get_data().filter()一样 默认是FIR
trials_filt = {cl_lab_left:bandpass(trials[cl_lab_left],8,15,sample_rate),
cl_lab_foot:bandpass(trials[cl_lab_foot],8,15,sample_rate)}# 只选择8,15该频率观察后
psd_left,freqs = get_psd(trials_filt[cl_lab_left])
psd_foot,freqs = get_psd(trials_filt[cl_lab_foot])
trials_PSD = {cl_lab_left:psd_left,cl_lab_foot:psd_foot}
plot_psd(
trials_PSD,
freqs,
[channel_name.index(ch) for ch in ['C3','Cz','C4']],
chan_lab=['left','center','right'],
maxy=500
)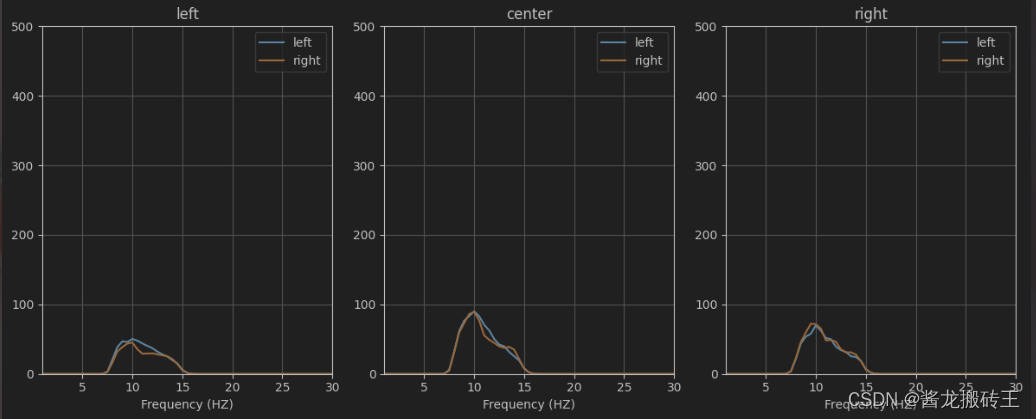
好了现在这个图只显示8-15这个区间的图我们看起来就很舒服了
4.构建CSP方法获取滤波器
在进行脑电信号处理途中,我们经常用的方法可能就是取对数反差了,听到方差这个东西大家脑子里一闪而过,CSP共空间模式,使方差最大化是吗,我们的主要目的是构建出滤波器W,下面就开始关键操作了
# 取信号的对数方差
def logvar(trials):
return np.log(np.var(trials,axis=1))trials_logvar = {cl_lab_left:logvar(trials_filt[cl_lab_left]),
cl_lab_foot:logvar(trials_filt[cl_lab_foot])}# 绘制每个通道的对数方差
def plot_logvar(trials):
plt.figure(figsize=(12,5))
x0 = np.arange(n_channels)
x1 = np.arange(n_channels) + 0.4
y0 = np.mean(trials[cl_lab_left],axis=1)
y1 = np.mean(trials[cl_lab_foot],axis=1)
plt.bar(x0,y0,width=0.5,color='b')
plt.bar(x1,y1,width=0.4,color='r')
plt.xlim(-0.5,n_channels+0.5)
plt.gca().yaxis.grid(True)
plt.title('log-var of each channel/component')
plt.xlabel('channel/components')
plt.ylabel('log-var')
plt.legend(cl_lab)plot_logvar(trials_logvar)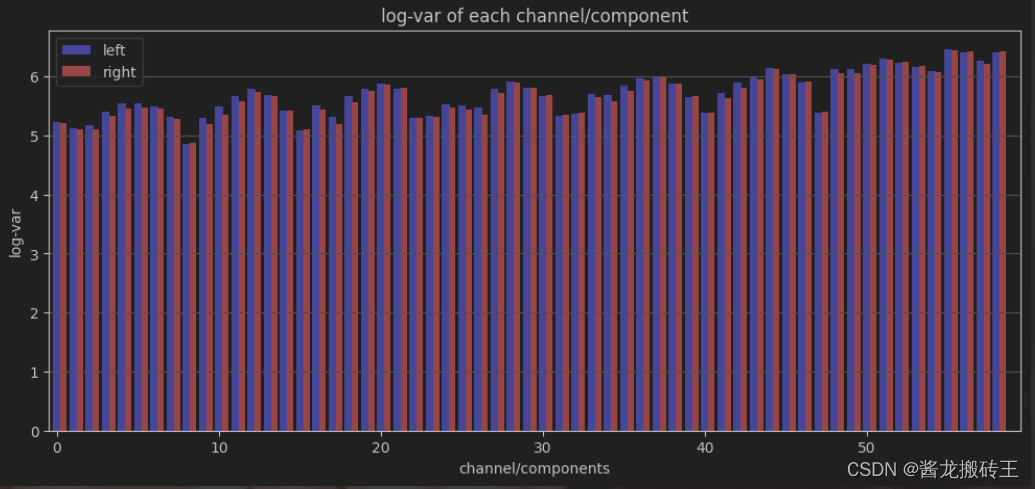
我们可以观察到,这方差一点都不明显,怎么去做分类呀?所以我们就需要构建出空间滤波器使得两者方差最大化。
# 计算协方差
def cov(trials):
ntrials = trials.shape[2]
covs = [trials[:,:,i].dot(trials[:,:,i].T)/nsamples for i in range(ntrials)]
return np.mean(covs,axis=0)# 矩阵的白化
def whitening(sigma):
u,l,_ = np.linalg.svd(sigma)
return u.dot(np.diag(l**-0.5))# 构建csp滤波器
def csp(trials_l,trials_r):
cov_l = cov(trials_l)
cov_r = cov(trials_r)
# 进行矩阵的白化操作
p = whitening(cov_l + cov_r)
B,_,_ = np.linalg.svd(p.T.dot(cov_r).dot(p))
W = p.dot(B)
return W# 进行滤波处理后方差最大化数据
def apply_mix(W,trials):
ntrials = trials.shape[2]
trials_csp = np.zeros((n_channels,nsamples,ntrials))
for i in range(ntrials):
trials_csp[:,:,i] = W.T.dot(trials[:,:,i])
return trials_cspW = csp(trials_filt[cl_lab_left],trials_filt[cl_lab_foot])
trials_csp = {cl_lab_left:apply_mix(W,trials_filt[cl_lab_left]),
cl_lab_foot:apply_mix(W,trials_filt[cl_lab_foot])}trials_logvar = {cl_lab_left:logvar(trials_csp[cl_lab_left]),
cl_lab_foot:logvar(trials_csp[cl_lab_foot])}
plot_logvar(trials_logvar)
经过上面一大堆巴拉巴拉的操作,现在我们利用使得两者方差最大化,在图中也能表现出来,如果不知道CSP工作原理的,推荐脑机接口社区的CSP共空间的公式推导,这里也不给大家继续推了。
psd_l,freqs = get_psd(trials_csp[cl_lab_left])
psd_r,freqs = get_psd(trials_csp[cl_lab_foot])
trials_PSD = {cl_lab_left:psd_l,cl_lab_foot:psd_r}
plot_psd(trials_PSD,freqs,[0,58,-1],chan_lab=['first component','middle component','last component'],maxy=0.75)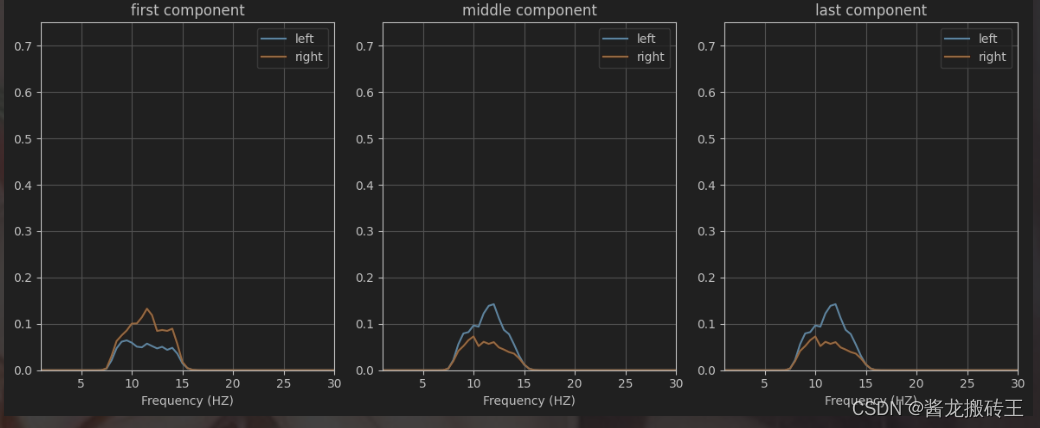
现在我们观察频谱图的时候是不是比之前顺眼多了,两者的差距更加明显,好了,我们在画画散点图试试看,这两类数据到底是什么样的。
# 画出经过csp方法处理后使两类差异最大化的散点图
def plot_scatter(left,right):
plt.figure()
# 将第一维度中的第一行作为x 最后一行作为y进行散点图的绘制 这也是一直没搞清楚绘图原理的原因
plt.scatter(left[0,:],left[-1,:],color='b')
plt.scatter(right[0,:],right[-1,:],color='r')
plt.xlabel('Last component')
plt.xlabel('First component')
plt.legend(cl_lab)# 观察一下数据
print(trials_logvar[cl_lab_left][0, :])
print(trials_logvar[cl_lab_left][-1,:])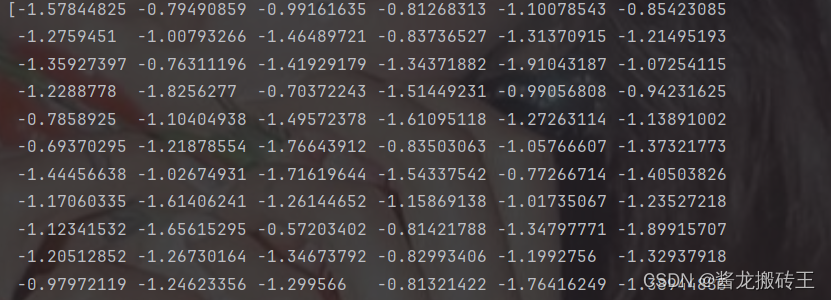
plot_scatter(trials_logvar[cl_lab_left],trials_logvar[cl_lab_foot])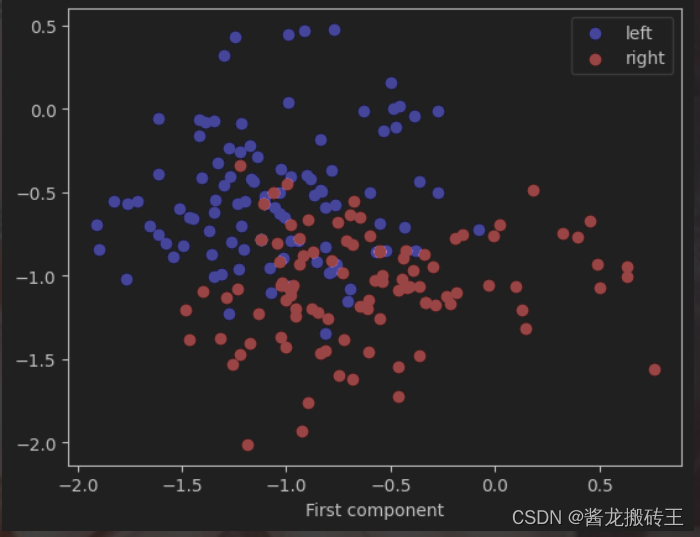
通过CSP处理之后将之前EEG的数据从200多、1000多变成了-2~0之间了,这样归一化数据观察起来更加方便,也防止奇异样本数据对实验造成影响,对分类更加有利。
5.对数据进行分类
# 用滤波后的数据分出训练集和测试集
train_percentage = 0.5
ntrain_l = int(trials_filt[cl_lab_left].shape[2]*train_percentage)
ntrain_r = int(trials_filt[cl_lab_foot].shape[2]*train_percentage)
ntest_l = trials_filt[cl_lab_left].shape[2] - ntrain_l
ntest_r = trials_filt[cl_lab_foot].shape[2] - ntrain_r
train = {cl_lab_left:trials_filt[cl_lab_left][:,:,:ntrain_l],
cl_lab_foot:trials_filt[cl_lab_foot][:,:,:ntrain_r]}
test = {cl_lab_left:trials_filt[cl_lab_left][:,:,ntrain_l:],
cl_lab_foot:trials_filt[cl_lab_foot][:,:,ntrain_r:]}
# 得到训练集和测试集数据的滤波器
W = csp(train[cl_lab_left],train[cl_lab_foot])train[cl_lab_left] = apply_mix(W,train[cl_lab_left])
train[cl_lab_foot] = apply_mix(W,train[cl_lab_foot])
test[cl_lab_left] = apply_mix(W,test[cl_lab_left])
test[cl_lab_foot] = apply_mix(W,test[cl_lab_foot])# 定义两个组件0和-1 也就是第一行数据和最后一行数据得到两个组件
component = np.array([0,-1])
train[cl_lab_left] = train[cl_lab_left][component,:,:]
train[cl_lab_foot] = train[cl_lab_foot][component,:,:]
test[cl_lab_left] = test[cl_lab_left][component,:,:]
test[cl_lab_foot] = test[cl_lab_foot][component,:,:]大家看这个component是不是很熟悉,这不是MNE包中CSP方法中的n_component中的成分吗?
现在我们取最前面一个和最后面一个数据作为我们的成分。
train[cl_lab_left] = logvar(train[cl_lab_left])
train[cl_lab_foot] = logvar(train[cl_lab_foot])
test[cl_lab_left] = logvar(test[cl_lab_left])
test[cl_lab_foot] = logvar(test[cl_lab_foot])def train_lda(class1,class2):
nclasses = 2
nclass1 = class1.shape[0]
nclass2 = class2.shape[0]
prior1 = nclass1/float(nclass1+nclass2)
prior2 = nclass2/float(nclass1+nclass1)
mean1 = np.mean(class1,axis=0)
mean2 = np.mean(class2,axis=0)
class1_centered = class1 - mean1
class2_centered = class2 - mean2
cov1 = class1_centered.T.dot(class1_centered)/(nclass1-nclasses)
cov2 = class2_centered.T.dot(class2_centered)/(nclass2-nclasses)
W = (mean2-mean1).dot(np.linalg.pinv(prior1*cov1)+prior2*cov2)
b = (prior1*mean1 + prior2*mean2).dot(W)
return (W,b)构造LDA分类器来获取我们的w权重和b偏置项,再利用其进行图像的绘制。
def apply_lda(test,W,b):
ntrials = test.shape[1]
prediction = []
for i in range(ntrials):
result = W.dot(test[:,i]) - b
if result <= 0:
prediction.append(1)
else:
prediction.append(2)
return np.array(prediction)# 得到权重和偏置项
W,b = train_lda(train[cl_lab_left].T,train[cl_lab_foot].T)
print('W',W)
print('b',b)# 画出分类后的图像
plot_scatter(train[cl_lab_left],train[cl_lab_foot])
x = np.arange(-5,1,0.1)
y = (b-W[0]*x) / W[1]
plt.plot(x,y,linestyle='--',linewidth=2,color='k')
plt.xlim(-5,1)
plt.ylim(-2.2,1)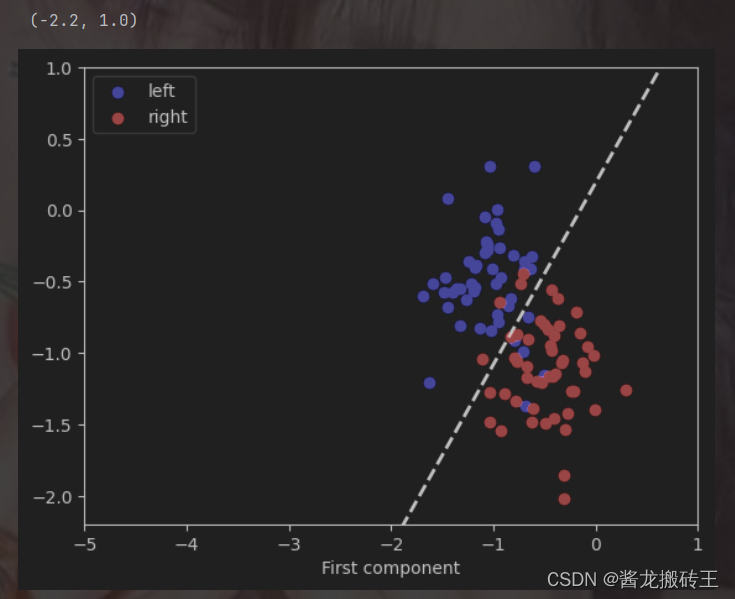
ok了,至此我们的数据已经分类完了,本次实验也接近尾声了。
三、总结
于我个人而言,本次实验加深了我对CSP更深的理解,特别是n_component代表着什么,我之前看到有人问如果CSP中的n_component=6的话,那么是不是3个为左边的特征3个为右边的特征呢?我们理想情况希望下是这样的,实际问题中可能会产生偏差,所以n_component的设置也会影响实验的最终的精确度,大家也可以去看我上一篇文章中,编写了n_component对实验acc的影响。最后感谢大家的耐心阅读,更新文章是我学习的动力,能够帮助大家也是我的初心。如果大家有什么意见或者看法欢迎评论区交流,大家一起进步。





















 2832
2832











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








