这里将会讨论聚类,聚类是指给定一组没有标签的样本集 D D D ,将 D D D 划分为互不相交的k个子集,即k个样本簇。
KMeans
k-平均聚类的目的是:把n个点(可以是样本的一次观察或一个实例)划分到k个聚类中,使得每个点都属于离他最近的均值(此即聚类中心)对应的聚类,以之作为聚类的标准。这个问题将归结为一个把数据空间划分为Voronoi cells的问题。
已知观测集
{
x
1
,
x
2
,
⋯
,
x
n
}
\left \{ x_{1},x_{2},\cdots,x_{n}\right \}
{x1,x2,⋯,xn},其中每个观测都是一个
d
d
d-维实向量,
k
k
k-平均聚类要把这
n
n
n个观测划分到
k
k
k个集合中(k≤n),使得组内平方和(WCSS within-cluster sum of squares)最小。换句话说,它的目标是找到使得下式满足的聚类
S
i
S_{i}
Si,
a
r
g
m
i
n
∑
i
=
1
k
∑
x
∈
S
i
∥
x
−
μ
i
∥
2
argmin\sum_{i=1}^{k}\sum_{x\in S_{i}}\left \| x-\mu_{i} \right \|^{2}
argmini=1∑kx∈Si∑∥x−μi∥2
其中
μ
i
\mu _{i}
μi 是
S
i
S_{i}
Si中所有点的均值。
我们来测试一下Kmeans算法
%pylab inline
import matplotlib.pyplot as plt
import numpy as np
#加载数据
from sklearn.datasets import make_blobs
blobs, classes = make_blobs(500, centers=3)#样本数据和类别
#绘制数据
f, ax = plt.subplots()
rgb = np.array(['r', 'g', 'b'])
ax.scatter(blobs[:, 0], blobs[:, 1], color=rgb[classes])
ax.set_title("Blobs")
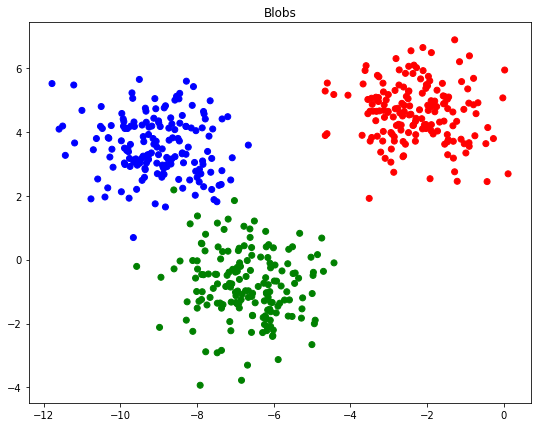
这里我们是知道每个样本类别的,实际情况是我们并不知道每个样本的类别。
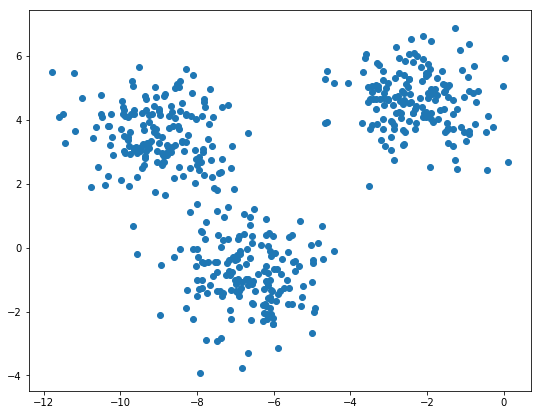
我们使用KMeans算法来进行聚类。
from sklearn.cluster import KMeans
kmean = KMeans(n_clusters=3)#为了测试方便,假设为3
kmean.fit(blobs)
kmean.cluster_centers_
kmean.cluster_centers_就是聚类后每个类别的中心,我们画出来
f, ax = plt.subplots()
ax.scatter(blobs[:, 0], blobs[:, 1], color=rgb[classes])
ax.scatter(kmean.cluster_centers_[:, 0],kmean.cluster_centers_[:, 1], marker='*', s=250,color='black', label='Centers')
ax.set_title("Blobs")
ax.legend()

可以看到还是很符合我们的观测的。
我们也可以打印真实的类别classes和聚类后的类别kmean.labels_ 有多少相同
np.sum(kmean.labels_ == classes)
我测试的是0 。当时我就傻了,后来打印出来发现其实都是对应的,只不过聚类后的类别标号改了,
kmean.labels_[:10]
Out:array([0, 0, 1, 2, 1, 1, 1, 0, 1, 2])
classes[:10]
Out:array([2, 2, 0, 1, 0, 0, 0, 2, 0, 1])
比如原始数据的类别0对应的类别1,类别1对应类别2,类别2对应类别0,如此对应之后会发现非常的一致。
tr = np.array([2,0,1])
new_labels = tr[kmean.labels_]
np.sum(new_labels == classes)
Out:497
可以看到真的是非常准确。
还有一个很有用的函数是transform,
kmean.transform(blobs)
这个函数会计算出每个样本与各个簇中心的距离
Out:array(
[[ 6.47297373, 1.39043536, 6.4936008 ],
[ 6.78947843, 1.51914705, 3.67659072],
[ 7.24414567, 5.42840092, 0.76940367],
[ 8.56306214, 5.78156881, 0.89062961],
[ 7.32149254, 0.89737788, 5.12246797]])
Silhouette distance
Silhouette距离是一种数据簇相似性(相同簇越密集越好,不同簇越分散越好)的度量方式。
假设数据已经根据某些聚类算法分为k类
C
=
{
C
1
,
C
2
⋯
,
C
k
}
C= \{ C_{1},C_{2}\cdots ,C_{k} \}
C={C1,C2⋯,Ck},对每一个样本
x
(
i
)
x^{(i)}
x(i),计算
x
(
i
)
x^{(i)}
x(i)与同一类中其他所有的数据距离的均值
a
(
i
,
x
i
∈
c
)
=
1
N
c
Σ
j
=
1
N
c
d
i
s
t
(
x
c
(
i
)
,
x
c
(
j
)
)
a(i,x_{i} \in c) = \frac{1}{N_{c}}\Sigma_{j=1}^{N_{c}} dist(x_{c}^{(i)},x_{c}^{(j)})
a(i,xi∈c)=Nc1Σj=1Ncdist(xc(i),xc(j))
其中假设
x
i
x_{i}
xi属于第c类,
N
c
N_{c}
Nc为第c类样本的总个数.
在计算样本
x
i
x_{i}
xi到所有其他簇的所有样本平均距离的最小值
b
(
i
)
=
m
i
n
{
a
(
i
,
x
i
∉
c
)
}
b(i) = min\{ a(i,x_{i} \notin c)\}
b(i)=min{a(i,xi∈/c)}
现在定义
s
(
i
)
=
b
(
i
)
−
a
(
i
)
m
a
x
{
a
(
i
)
,
b
(
i
)
}
s(i) = \frac{b(i)-a(i)}{max\{a(i),b(i)\}}
s(i)=max{a(i),b(i)}b(i)−a(i)
可以看到
−
1
⩽
s
(
i
)
⩽
1
-1\leqslant s(i) \leqslant 1
−1⩽s(i)⩽1 。我自己认为,可以将
b
(
i
)
−
a
(
i
)
b(i)-a(i)
b(i)−a(i)看作是类内间距,
{
m
a
x
{
a
(
i
)
,
b
(
i
)
}
\{max\{a(i),b(i)\}
{max{a(i),b(i)}看作类间距离,他们的比值
s
(
i
)
s(i)
s(i)越接近1,说明类内间距小,同类样本密集,类间距离大,不同类的样本分开的更散。
我们可以计算出所有样本的Silhouette距离。
from sklearn import metrics
silhouette_samples = metrics.silhouette_samples(blobs,kmean.labels_)
f, ax = plt.subplots()
ax.set_title("Hist of Silhouette Samples")
ax.hist(silhouette_samples,bins=100);
可以看到silhouette_samples 都非常接近1.打印一下均值silhouette_samples.mean()。到这里就都结束了,那么我们如何知道我们的模型到底好不好呢?这个测试中我们比较了kmean.labels_和classes。但是实际上我们是不知道真实的类别的,所以我们可以用metrics.silhouette_score 来指示模型的好坏,这个函数得到的其实就是所有样本silhouette距离的均值,值越大说明有越多的样本分的越准确。这里我们试着改变类别的个数k,来观测k值为多少的时候模型最好。
sillhouette_avgs = []
# this could take a while
for k in range(2, 60):
kmean = KMeans(n_clusters=k).fit(blobs)
sillhouette_avgs.append(metrics.silhouette_score(blobs,kmean.labels_))
f, ax = plt.subplots()
ax.plot(sillhouette_avgs)
sillhouette_avgs.index(max(sillhouette_avgs))+2 #+2是从2开始的
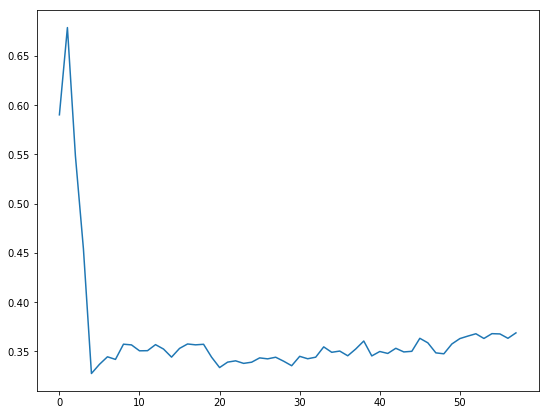
MiniBatch KMeans
由于KMeans算法的实现原理,当数据很多的时候常规的计算会非常的耗时,我们使用MiniBatch KMeans来进行加速计算。
from sklearn.datasets import make_blobs
blobs, labels = make_blobs(int(1e6), 3)
from sklearn.cluster import KMeans, MiniBatchKMeans
kmeans = KMeans(n_clusters=3)
minibatch = MiniBatchKMeans(n_clusters=3)
我们用两种方式来测试一下,看看到底有没有计算变快
%time kmeans.fit(blobs)
Out : Wall time: 5.55 s
%time minibatch.fit(blobs)
Out : Wall time: 1.21 s
可以看到明显加速了,但是光有计算速度是不行的,看看结果都如何
minibatch.cluster_centers_
kmeans.cluster_centers_
我们也可以直接看看中心距离差了多少
np.diag(pairwise.pairwise_distances(kmeans.cluster_centers_, minibatch.cluster_centers_))
可以看到两种方式计算的中心结果相差不多。(应该和 批梯度下降法,随机梯度下降法类似)
利用聚类模糊图片
这里讲一个有趣的应用,利用聚类模糊图像。一个图片由高Height,宽Width,RGB构成,例如一个图片大小为100200像素,每个像素为(r,g,b)那么总共就有1002003个数据需要存储,如果我们将图片img.reshape(wh,3),那么就相当于有w*h个样本,每个样本3个属性,我们通过聚类将这些样本聚成k类,求出每个类的中心值,然后所有这个类的样本都用中心值代替,再将这些数据reshape(w,h,3)还原成图片,那么我们只需要记录k个中心值就可以了,这样就是图片模糊化了。
from scipy import ndimage
img = ndimage.imread("e:\\girl.jpg")
plt.imshow(img)

x, y, z = img.shape
long_img = img.reshape(x*y, z) #将图片格式转换有3个属性的大小为x*y个样本集
使用聚类算法
from sklearn import cluster
k_means = cluster.KMeans(n_clusters=5)
k_means.fit(long_img)
将计算后的每个类的中心代替每个类中每个样本的值,绘制出来(不忍直视)
centers = k_means.cluster_centers_
labels = k_means.labels_
plt.imshow(centers[labels].reshape(x, y, z))

绘制近邻点
我们可以计算出给定的点与其他点的距离,绘制出近邻的点。计算距离的函数用pairwise_distances 。
from sklearn.metrics import pairwise
from sklearn.datasets import make_blobs
points, labels = make_blobs()
distances = pairwise.pairwise_distances(points)
得到的distances 就是任意两个点距离的矩阵,例如distances[0] 就是第0个点与其他所有点的距离,假如我们想要得到与第一个样本点的最近的5个点,我们可以
ranks = np.argsort(distances[0])
near = points[ranks][:5]
argsort 函数返回的是数组值从小到大的索引值,我们可以得到前五个最近的值。
f, ax = plt.subplots()
ax.scatter(points[:,0],points[:,1],c='r')
ax.scatter(near[:,0],near[:,1],c='b')
ax.scatter(points[0,0],points[0,1], marker='*', s=250,color='black', label='Centers')
ax.legend()
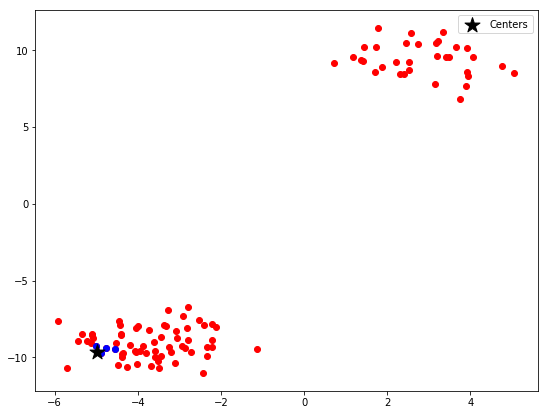
这里的距离默认是欧氏距离,即
L
2
L_{2}
L2。当然我们也可以使用其他距离,
- cityblock
- cosine
- euclidean
- L1
- L2
- manhattan
使用KMeans进行异常检测
有时候我们的样本会有一些异常点,这些点的存在是我们的模型变得非常糟糕,我们使用KMeans进行检测。异常值检测是通过找出簇的质心,然后通过距离质心的距离识别潜在离群值的点,也就是把距离最远的中心最远的几个点去掉。为了测试方便,我们生成一个中心的的簇。
from sklearn.datasets import make_blobs
import numpy as np
from sklearn.cluster import KMeans
X, labels = make_blobs(100, centers=1)
kmeans = KMeans(n_clusters=1)
kmeans.fit(X)
然后绘制出来
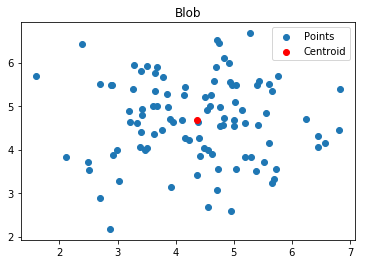
f, ax = plt.subplots()
ax.set_title("Blob")
ax.scatter(X[:, 0], X[:, 1], label='Points')
ax.scatter(kmeans.cluster_centers_[:, 0],kmeans.cluster_centers_[:, 1], label='Centroid',color='r')
ax.legend()
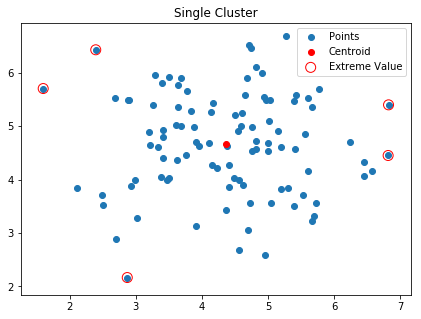
接着计算出所有点与中心的距离,我们得到最大的5个
distances = kmeans.transform(X)
sorted_idx = np.argsort(distances.ravel())[::-1][:5]
将其标注出来
f, ax = plt.subplots(figsize=(7, 5))
ax.set_title("Single Cluster")
ax.scatter(X[:, 0], X[:, 1], label='Points')
ax.scatter(kmeans.cluster_centers_[:, 0],kmeans.cluster_centers_[:, 1],label='Centroid', color='r')
ax.scatter(X[sorted_idx][:, 0], X[sorted_idx][:, 1],label='Extreme Value', edgecolors='r',facecolors='none', s=100)
ax.legend(loc='best')
然后将其删除
new_X = np.delete(X, sorted_idx, axis=0)
我们将新旧中心都画出来看看
new_kmeans = KMeans(n_clusters=1)
new_kmeans.fit(new_X)
f, ax = plt.subplots()
ax.set_title("Extreme Values Removed")
ax.scatter(new_X[:, 0], new_X[:, 1], label='Pruned Points')
ax.scatter(kmeans.cluster_centers_[:, 0],kmeans.cluster_centers_[:, 1], label='Old Centroid',color='r', s=80, alpha=.5)
ax.scatter(new_kmeans.cluster_centers_[:, 0],new_kmeans.cluster_centers_[:, 1], label='New Centroid',color='m', s=80, alpha=.5)
ax.legend(loc='best')
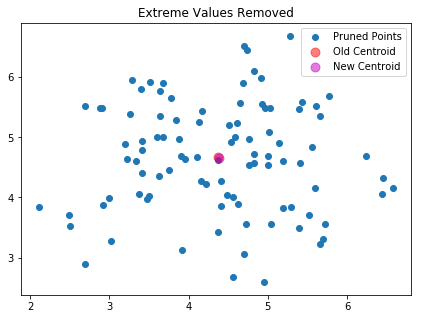
不得不说,新旧的中心这个真的是分不开啊。不过你可以已知删除,知道你满意。
使用KNN进行回归
KNN(k-Nearest Neighbors)其实非常的简单,就是物以类聚,人以群分。你想知道某个人的薪资,你看看他身边的人的薪资,平均一下差不多就是他的薪资了。回归时,你想知道某个样本的输出值,你可以看看这个样本周围最近的k个点的输出值时多少,这个样本点的输出值也不会偏离太多。
我们使用iris数据集进行测试
from sklearn import datasets
iris = datasets.load_iris()
iris.feature_names
Out:['sepal length (cm)','sepal width (cm)','petal length (cm)','petal width (cm)']
我们使用sepal length 和sepal width来预测petal length,
X,y=iris.data[:,[0,1]],iris.data[:,2]
from sklearn.linear_model import LinearRegression
lr = LinearRegression()
lr.fit(X, y)
print("The MSE is: {:.2}".format(np.power(y - lr.predict(X),2).mean()))
Out:The MSE is: 0.41
然后使用KNN进行测试,
from sklearn.neighbors import KNeighborsRegressor
knnr = KNeighborsRegressor(n_neighbors=10)
knnr.fit(X, y)
print("The MSE is: {:.2}".format(np.power(y - knnr.predict(X),2).mean()))
Out:The MSE is: 0.17





















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








