目录
实验目的
给定基因组序列(大肠杆菌E. Coli, 长度~5Mbp)作为参考基因组。给定一定数量来自于大肠杆菌的基因组测序片段(共282224条,pair-end,70 bp/read)作为输入。建立算法和系统,可以完成对基因组测序片段的比对,重构片段位置(基因组坐标,正/反链)和基因组片段与参考序列的差异。
1.完整地完成算法所有模块
2.支持编辑距离3以上的片段比对
3.正确重构片段基因组位置与局部差异
4.支持SAM格式输出
5.程序运行正确,程序代码优化较好
6.所有文档完整且写作规范
实验原理
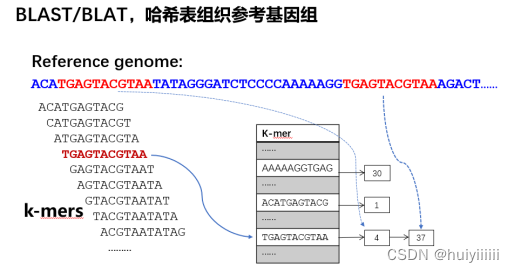
SNAP算法是基于一个seed的基因组(或其他参考数据库)短子串的
哈希索引。给定要对齐的读取,SNAP从中绘制多个长度为s(seed长度)的子字符串,并在哈希索引中执行精确查找,以查找数据库中包含相同子字符串的位置。然后计算读取位置和每个候选位置之间的编辑距离,以找到最佳对齐方式。
其中有两个特性使SNAP不同于以前的算法,并提高了它的速度:使用更大的索引来交换内存以减少计算,以及对局部对齐步骤的一组优化,这大大降低了根据候选位置测试读取的成本。
SNAP的索引是一个哈希表,从固定长度的seed字符串到引用数据库中出现这些字符串的位置列表。它在两个方面与BLAST中的参数不同:
1. 更长的seed,SNAP的索引的哈希表的seed的长度典型值为20个碱基对,较长的种子意味着从read中提取的seed与参考数据库精确匹配的可能性较小,但也大大减少了假阳性种子匹配的数量。通过相应概率计算最后将seed的长度设计成为20。但是在论文中作者是以人类基因组且较大的read长度(100bp)分析的,在该实验中用的是大肠杆菌的基因组而且read长度不是很大(70bp),理论上不需要这么长的seed索引,但是为了能尽可能还原这个算法,我仍然将seed的长度设计为20。
![]()
2.重叠种子:为了节省内存,许多索引只索引参考数据库的非重叠s-mer(例如,前10个碱基,然后是下10个碱基,等等),因此当种子长度为s时,索引中只出现数据库中1/s的位置。这种方法的缺点是每次读取必须尝试更多seed,以找到与索引中使用的步幅匹配的seed。作者从我看不懂的计算机组成原理得出了一些关于查找代价的结论,最终确定了SNAP通过索引重叠的s-mer(即,在数据库上每一次滑动一个长度作为一个seed)来避免这一成本。这需要一个更大的索引,但它仍然在现代服务器的能力范围内,但好像是不在我的计算机的能力范围内,为了能尽可能还原这个算法,我将实验中的hash表索引做了一些改动,使其节约了很多的空间但又不违反seed 20bp长度的索引,虽然这样做会比标准的SNAP索引慢一些,但影响的速度为可以接受的,而且可以在我的计算机运行下去:
若seed大小为20bp,则一个20bp的hash表索引,需要大小420这么大的表头,这很明显很大,于是我这样改进了实验的hash表索引:
在哈希表中允许碰撞产生,在计算机的能力范围内设置一个很大的质数尽量减少碰撞同时防止占用空间过大。
![]()
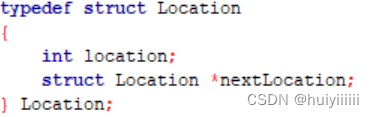
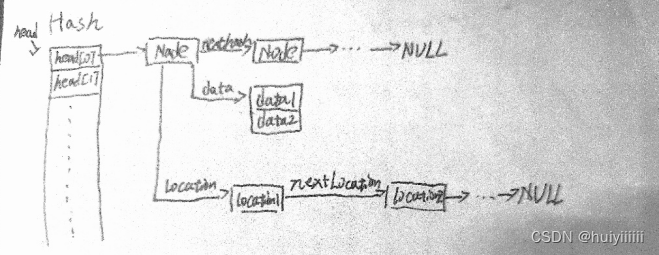
Hash表中有一个大小,长度,WNode类型(表头)的数组head[]。
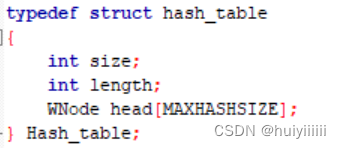
表头只包括一个node类型(节点)的指针nexthash。
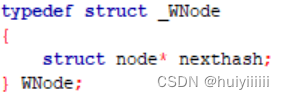
节点包括一个Element类型data(seed数据)的结构体指针,指向同索引的下一个节点的指针nexthash。一个Location类型(位置数据)的指针location,代表该节点位置数量的num。

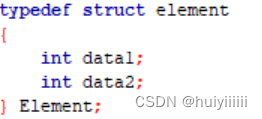
Element结构体用于存储seed的信息,将20bp按A-00,C-01,G-10, T-11的方式编码,并取前30位转化为data1(int类型),后10位转化位data2(int类型)。
Location结构体用于存储位置信息,包括一个location(int类型)代表该seed在参考基因组的一个位置,Location类型的指针nextLocation指向下一个位置信息。
最终hash表结构如下图所示:
SNAP的伪代码:

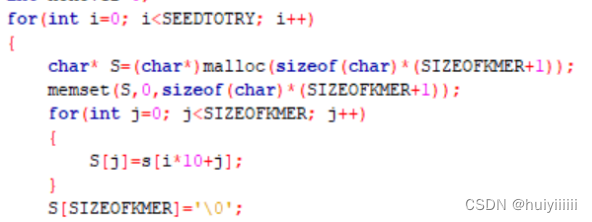
算法的第三行第四行:尝试n个read的seed从尽可能多的连续非重叠种子开始,然后用s/2、s/4、3s/4等偏移下一组种子。在实验中我在一个read中选取了6个seed,从开始开始,每隔10bp选择一个。
算法的第五行,判断seed的位置数量是否小于等于hmax,在hash表中找到seed对应的节点,节点上的num即seed的位置数量。
![]()
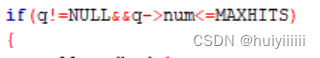
算法第六行第七行第八行,记录不同的位置的数量,将其存储位两个数组,可以减少时间与空间的消耗。其中seedhitting记录位置信息,seedhittings记录对应位置的hit次数信息,每次有新hit都遍历一下seedhitting。若hit的位置不存在,则将其记录在seedhitting中,并设置seedhittings对应的次数为1;否则,直接在对应的次数上加一。
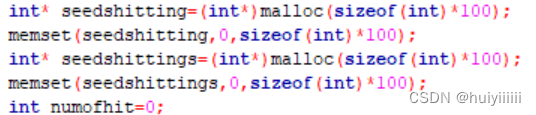

算法第十行,寻找最多hit的位置与次数,遍历seedhittings即可。

算法第18行,计算编辑距离,实验中采用的是动态规划的方法,不同的是当运算过程中一旦中途结果大于dlomit,立刻跳出。
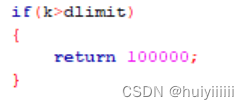
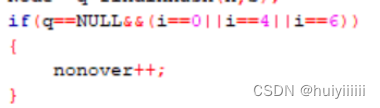
算法第23行,利用鸽笼原理当hash表无法索引到的非重叠seed大于等于dbest+c时,直接忽略未计算的seed直接退出该条read的计算。
![]()





 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章
















 1980
1980

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








