- 期刊名: Molecular Therapy Nucleic Acids
- 期刊名缩写:NUCLEIC ACIDS RES
- 国际刊号:0305-1048
- 2021年影响因子/JCR分区:16.971/Q1
- 出版国家或地区:ENGLAND
- 中科院分区:
- 大类学科及分区:生物
- 生物:一区
- 是否TOP期刊:是
- 小类学科及分区:生化与分子生物
背景介绍
5-甲基胞嘧啶(5-Methylcytosine, m5C)是一种重要的转录后修饰子,广泛存在于多种RNA中。许多研究表明,m5C在许多生物学功能中发挥着重要作用,如RNA结构稳定性和代谢方面。 m5C是mRNA中最重要的修饰之一。美国食品和药物管理局(FDA)于2020年12月11日授权一款运用mRNA(信使核糖核酸)技术研制的新冠疫苗的紧急使用许可。因此,能够准确识别m5C位点的计算方法将是非常有价值的。计算方法是一种从高通量RNA序列数据中识别m5C位点,并有助于解释这一重要修饰的功能机制。然而,准确识别m5C是一个挑战。由于mRNA分子的不稳定性,比较活跃,存在时间又短。高通量测序技术通常无法在单核苷酸分辨率上准确识别m5C位点。为了解这一重要的RNA修饰的功能。在这篇文章中提出了一种新的物种特异性计算方法Staem5,用于准确预测小鼠类和拟南芥的RNA m5C位点。
问题探讨
本研究提出了一种新的物种特异性计算方法Staem5,用于准确预测小鼠类和拟南芥(拟南芥是植物领域应用较为广泛的模式生物。由于具有较小的基因组(135 Mbp),生命周期短,可以在多种环境下生长,一直被视为研究植物遗传学、进化、种群遗传学和植物生长发育的系统。)的RNA m5C位点。Staem5是通过采用特征融合策略来利用信息序列剖面来开发的,首先选择了堆叠集成学习框架结合了5种流行的机器学习算法。那作者又是怎样选择的这些机器学习的模型?堆叠策略又是什么?通过什么方法来证明:Staem5大量的基准测试表明,在交叉验证和独立测试中,Staem5的表现优于最先进的方法。(说明m5c位点重要,用一种最优的组合方式来预测两个数据集里面M5c位点的准确性)

方法介绍
许多基于序列衍生信息和机器学习算法的方法已经被开发出来,用于预测四个物种的m5C位点,包括智人(homo sapiens)、小家鼠(Mus musculus)、酿酒酵母(Saccharomyces cerevisiae)和拟南芥(Arabidopsis thaliana)。根据机器学习算法,这些方法可以分为两类:(1)基于支持向量机的预测器,包括m5C-PseDNC、M5C-HPCR、RNAm5CPred、m5CPred-SVM和iRNA-m5C_SVM;(2)基于随机森林(RF)的方法,包括PEA-m5C、iRNA-m5C。Table1从几个方面总结了这两类专门为m5C设计的预测器,包括特征提取、性能评估策略、种类、网络服务器或软件可用性和基准数据集。我们发现大多数方法都是为智人。只有少数预测因子被设计和测试m5C位点。如iRNA-m5C、iRNA-m5C_SVM、RNAm5Cfinder和m5cpred - svm。此外,m5C位点与预测智人的预测性能家鼠与拟南芥与智人相比不好。例如,m5CPred-SVM、iRNA-m5C_SVM和iRNA-m5C都是在拟南芥的同一个基准数据集上开发的。在交叉验证的平均准确性方面分别达到了71.8%、73.06%和70.7%。原因可能是这些预测器是基于单一的RF或SVM算法开发的。随着集成学习策略在生物信息学中用于开发稳健预测模型的最新进展,我们有动力利用集成学习技术来提高M. mususanda的m5C预测拟南芥。
数据
https://github.com/Cxd-626/Staem5/tree/main/data
代码
Staem5/m5C_predict.py at main · Cxd-626/Staem5 · GitHub
结果
集合了四种编码:PSP、K-mer(k=1、2、3)、PCPseDNC、PseEIIP,采用贝叶斯算法进行优化。将SVM、XGBoost、LightGBM、ExtraTree、GBDT等基分类器组合采用堆叠策略。同时,利用F评分降低特征的维数和计算时间,与训练数据集和独立数据集相比,Staem5显示出了与其他现有方法相比的优势。
分析异同点
使用Two Sample Logo分析包含m5C位点的序列片段的核苷酸偏好,为了分析两个数据集有没有一个共同的特点。
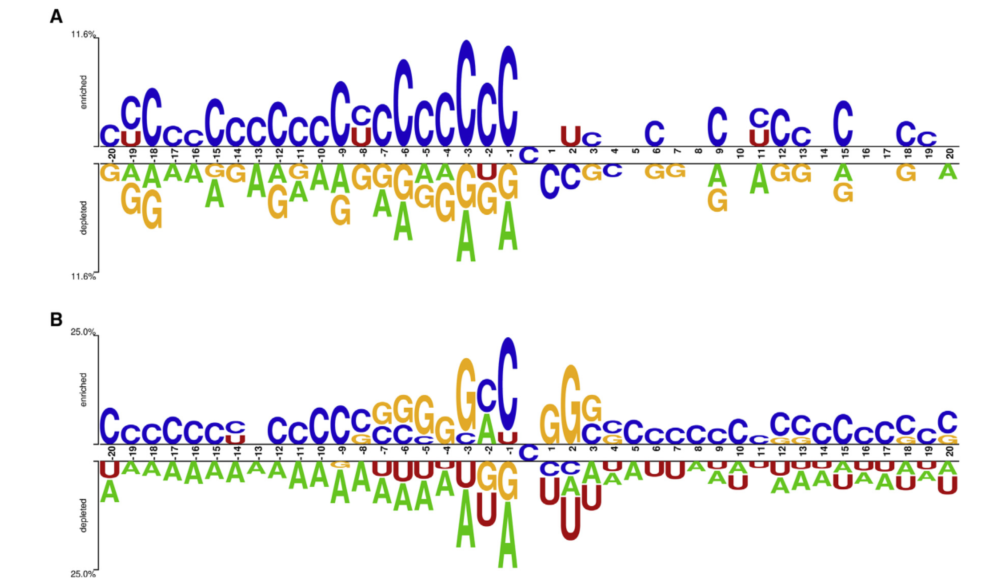
因此,建立一个跨物种预测的通用模型是很困难的,有必要建立物种特异性模型。
分析过程
选择五种机器学习算法:SVM,GBDT,XGBoost,LightGBM,and ExtraTree
预测:拟南芥与家鼠的数据集,首先设置了超参数然后用贝叶斯算法进行优化,通过10倍交叉验证来做的五个分类器的比较,展示出了设置超参数前后之间的对比。通过柱状图可以看出来ExtraTree与LightGBM变化并不是很明显,SVM与GBDT无论是拟南芥的数据集还是家鼠的数据集都有很大的变化

其次,选择堆叠策略进行,测试组合选择SVM与XGBoost进行组合发现ACC并没有提高多少仅从73.62%提高到73.85%。再继续整合LightGBM之后ACC从73.85%提升到73.89%,再继续整合GBDT和ExtraTree的ACC不升反降。所以选择SVM、XGBoost和LightGBM作为堆叠模型的基分类器,其准确率达到73.89%,MCC达到0.479。
进一步的将堆叠策略与投票策略进行比较发现堆叠策略各项指标好于投票策略。



最终结果
将Staem5与iRNA-m5C、m5CPredSVM和iRNA-m5C_SVM进行了比较通过表中可以看出结果,staem5好于其他几种结果。 在两个 A. thaliana and M. musculus,更适合预测m5c位点,各项指标也比较突出。最后选择SVM,XGBoost,LightGBM作为新定义的steam5。








 研究提出Staem5,一种基于特征融合和堆叠集成学习的计算方法,用于准确预测小鼠和拟南芥RNA的5-甲基胞嘧啶(m5C)位点。该方法通过结合SVM、XGBoost和LightGBM算法,优化性能并超越现有预测工具。实验结果显示,Staem5在交叉验证和独立测试中表现出优越的准确性,为m5C功能研究提供有力工具。
研究提出Staem5,一种基于特征融合和堆叠集成学习的计算方法,用于准确预测小鼠和拟南芥RNA的5-甲基胞嘧啶(m5C)位点。该方法通过结合SVM、XGBoost和LightGBM算法,优化性能并超越现有预测工具。实验结果显示,Staem5在交叉验证和独立测试中表现出优越的准确性,为m5C功能研究提供有力工具。
















 948
948

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








