这次复现一篇Cell文章的图表,原文中提供了部分数据,有部分数据是小编为了作为创造的,大体上和原文是一致的,重点是学习过程。要做到复现图上的每一个元素。原文图片如下:
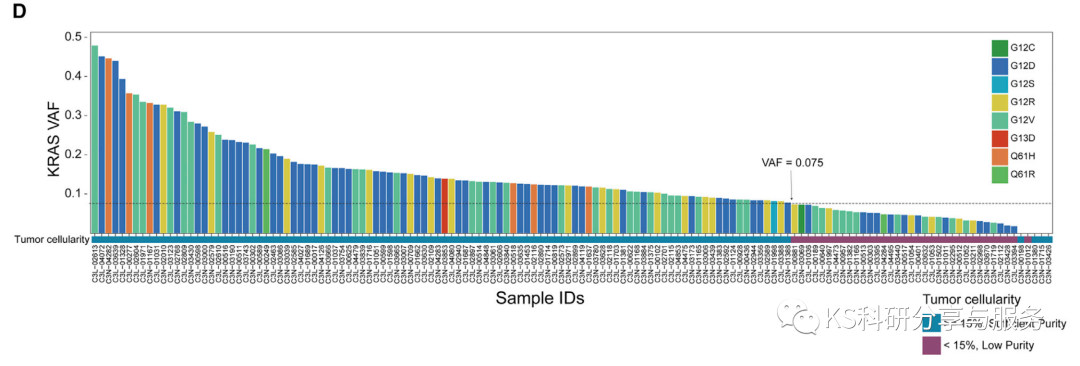
(Reference:Proteogenomic characterization of pancreatic ductal adenocarcinoma)
我的复现结果如下:
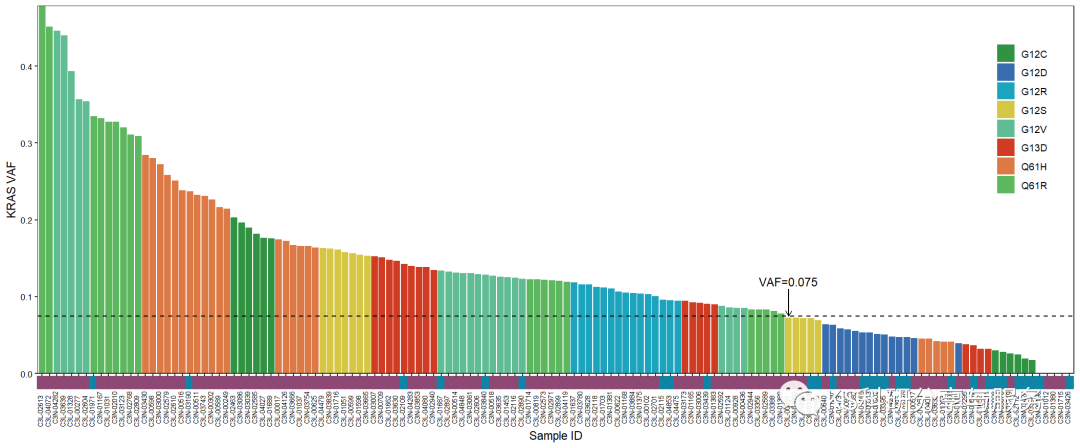
image.png
接下来慢慢盘它:
先做柱状图
setwd('E:/生物信息学/复现Cell柱状图加注释')
A <- read.csv('Tumor.CSV',header = T)
library(ggplot2)
library(ggh4x)
A$cellularity <- ''
A$cellularity[which(A$Tumor.cellularity >=15)] = '>=15,SP'
A$cellularity[which(A$Tumor.cellularity <15)] = '<15,LP'
p <- ggplot(A, aes(x=reorder(case_id,-KRAS_VAF),y=KRAS_VAF))+
geom_bar(aes(fill=group), stat = 'identity')+
labs(x='Sample ID',y='KRAS VAF')+
scale_fill_manual(values = c("#309342","#376CAE","#1CA4BF","#D5C643",
"#5FBC93","#D03B23","#DC7944","#5DB75E"))+
theme_bw()+
scale_y_continuous(expand = c(0,0))+
theme(panel.grid = element_blank(),
legend.position = c(0.95, 0.7),
legend.title = element_blank(),
axis.text.x = element_text(colour = "black", size=6,
angle = 90, hjust = -3, vjust = 0.1),
axis.text.y = element_text(colour = 'black',size = 8),
axis.title.x = element_text(margin =
margin(0.5,1,0,1,'cm')))+
geom_hline(yintercept = 0.075, linetype=2, cex=0.5)+
annotate(geom = 'text', label="VAF=0.075", x=102, y=0.12)+
geom_segment(aes(x = 102, y = 0.11, xend = 102, yend = 0.075),
arrow = arrow(length = unit(0.2, "cm")))
添加分组注释
B <- A
B <- B[order(-B$KRAS_VAF),]
library(forcats)
B$case_id <- as.factor(B$case_id)
B$case_id <- fct_inorder(B$case_id)
Tumor.cellularity <- B$case_id %>% as.data.frame() %>%
mutate(group=B$cellularity) %>%
mutate(p="")%>%
ggplot(aes(p,.,fill=group))+
geom_tile() +
scale_y_discrete(position="right") +
scale_fill_manual(values = c("#1084A4",
"#8D4873"))+
theme_minimal()+xlab(NULL) + ylab(NULL) +
theme(axis.text.y = element_blank(),
axis.text.x =element_blank(),
axis.ticks.x = element_blank(),
legend.position = 'none')+
labs(fill = "Tumor.cellularity")+
coord_flip()
bottom <- ggplotGrob(Tumor.cellularity)
p+annotation_custom(bottom,xmin=-1,xmax=141.5,ymin=-0.03,ymax=0.01)
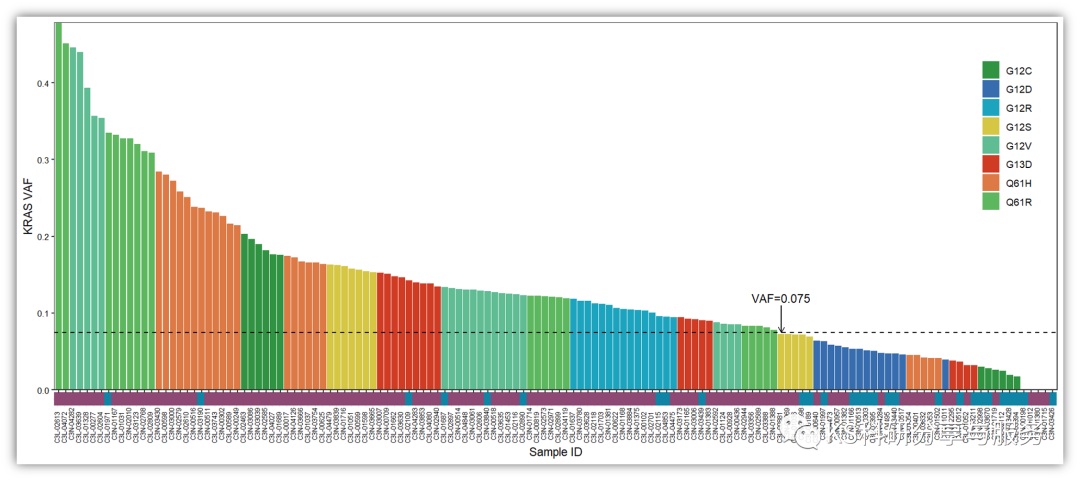
image.png
虽然分组注释是强行加上去的(因为没有想到好的办法,直接在坐标轴下面添加,小伙伴有好办法可私信),也有一个遗憾,就是分组的legend需要后期手动添加,但是整体复现了这个结果,还是不错的,整个图还是能够学到不少东西的。注释代码及数据已上传群文件!
如果觉得分享的只是有用,点个赞。更多精彩可关注我的公众好《KS科研分享与服务》!





















 5419
5419











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








