引言
亲爱的读者们,您是否在寻找某个特定的数据集,用于研究或项目实践?欢迎您在评论区留言,或者通过公众号私信告诉我,您想要的数据集的类型主题。小编会竭尽全力为您寻找,并在找到后第一时间与您分享。
一、研究意义
脑肿瘤图像的研究具有深远的意义,主要体现在以下几个方面:
早期诊断:脑肿瘤图像的准确分析可以帮助医生实现脑肿瘤的早期诊断。早期诊断对于患者的治疗至关重要,因为早期治疗往往能够提高治愈率,并减少并发症的发生。
治疗方案制定:通过脑肿瘤图像的详细分析,医生可以了解肿瘤的具体位置、大小、形状以及其与周围组织的关系,从而制定出更精确、更有效的治疗方案。这有助于实现个体化治疗,提高治疗效果。
疗效评估:在治疗过程中,脑肿瘤图像的跟踪监测可以帮助医生评估治疗效果。通过比较不同时间点的图像,医生可以了解肿瘤是否缩小、消失或扩散,从而及时调整治疗方案。
疾病研究:脑肿瘤图像的研究有助于深入了解脑肿瘤的发病机制、生长规律以及影响因素等。这有助于推动脑肿瘤研究的深入发展,为临床诊断和治疗提供新的思路和方法。
技术进步:脑肿瘤图像的研究促进了医学影像技术的不断发展。随着计算机视觉、人工智能等技术的不断成熟,脑肿瘤图像的自动分割、识别和分类等任务得到了更好的解决,提高了诊断的准确性和效率。
患者教育:脑肿瘤图像的可视化有助于患者了解自己的病情,增强对治疗的信心。通过向患者展示肿瘤图像,医生可以更好地解释病情和治疗方案,从而增强医患之间的沟通和信任。
总之,脑肿瘤图像的研究对于提高脑肿瘤的诊断和治疗水平、推动医学影像技术的发展以及促进患者教育等方面都具有重要意义。
二、应用
脑肿瘤图像的应用广泛且重要,主要体现在以下几个方面:
快速、准确的诊断:医学图像处理算法可以快速、准确地检测脑部肿瘤,减少了人为误差的可能性,提高了诊断的准确性。例如,基于深度学习的脑肿瘤自动分割方法已成为研究热点,它利用高分辨率的MRI图像进行训练,通过卷积神经网络模型学习特征表示,并结合传统的分割方法实现脑肿瘤分割。
提供全面的信息:医学图像处理算法能够准确地测量和计算肿瘤的大小、形状、位置和组织密度等关键指标,为医生提供详尽的信息,有助于更好地了解肿瘤的生长和发展情况,为后续的治疗方案制定提供参考依据。
辅助手术:在脑肿瘤手术中,医学图像处理技术如仿真导航技术和各种影像学技术起到了至关重要的作用。它们能够提供高清晰度的图像,帮助医生在实时图像中准确切除脑肿瘤,避免误伤周边结构。
术后评估:术后评估是脑肿瘤治疗的重要环节,影像学在此起到了关键作用。通过术后影像学检查,医生能够评估手术效果、分析手术后并发症以及进行随访和观察。常见的术后影像学检查方法包括CT扫描、MRI和PET-CT等。
分类和诊断:医学影像分析结合机器学习算法可以对脑瘤进行分类和诊断。通过构建合适的特征向量和分类模型,可以自动判断脑瘤的良恶性,辅助医生制定更合理的治疗方案。
特征提取和分析:医学影像分析可以提取脑瘤的生物学和形态学特征,例如肿瘤的血管密度、灰度值、纹理等。这些特征对于病灶的恶性程度判断和预后预测具有重要意义。
脑部疾病的自动分割:脑部影像中包含了许多组织和器官,人工智能可以通过分割技术,自动将其分割成不同的区域,如脑组织、血管、脑脊液等,为医生提供更多的信息和参考,帮助他们更好地了解脑部结构和疾病特征。
脑部疾病的评估和预测:人工智能可以根据脑部影像的数据和其他临床数据,进行脑部疾病的评估和预测,如预测肿瘤生长的速度、预测病人的预后等,帮助医生更好地制定治疗计划,并且更好地预测疾病的进展情况。
脑部影像数据的可视化:人工智能可以将脑部影像数据转换为三维模型,提供更多的可视化信息,帮助医生更好地了解脑部结构和疾病特征。
综上所述,脑肿瘤图像的应用范围广泛,包括诊断、治疗、评估等多个方面,为脑部疾病的管理和治疗提供了强有力的支持。
三、数据集
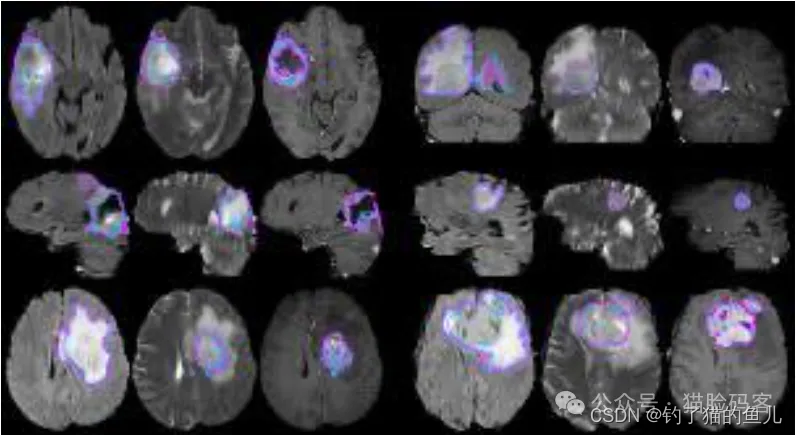
简介
BraTS 2015 数据集是一个用于脑肿瘤图像分割的数据集。它包含了220个高级别胶质瘤(HGG)和54个低级别胶质瘤(LGG)的MRI图像。这四种MRI模式是T1、T1c、T2和T2FLAIR。针对四种肿瘤内类别提供了分割的“真实标签”,即水肿、增强肿瘤、非增强肿瘤和坏死。
论文
https://ieeexplore.ieee.org/stamp/stamp.jsp?tp=&arnumber=6975210
论文摘要:本文报告了与MICCAI 2012和2013会议联合组织的多模态脑肿瘤图像分割基准(BRATS)的设置和结果。我们应用了20种最先进的肿瘤分割算法,对65例低级别和高级别胶质瘤患者的多对比度MR扫描图像进行了处理——这些图像由多达四名评分者手动标注,同时也对65例使用肿瘤图像模拟软件生成的类似扫描图像进行了处理。定量评估显示,不同的人类评分者在分割各种肿瘤亚区域时存在相当大的分歧(Dice系数在74%–85%之间),这说明了这项任务的难度。我们发现,不同的算法在不同的亚区域上表现最佳(达到了与人类评分者间变异性相当的性能),但没有一种算法在所有亚区域上同时排名第一。通过使用层次化多数投票融合几种好的算法,我们得到的分割结果始终优于所有单独的算法,这表明未来仍有进一步方法学改进的空间。BRATS图像数据和手动标注继续通过在线评估系统作为持续的基准资源公开提供。
地址
关注公众号,查看“第103期 语义分割——脑肿瘤图像分割数据集”,文章末尾



























 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










