简单实现和测试
## 参考 https://zhuanlan.zhihu.com/p/361357925
import math
import numpy as np
import sklearn
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import dendrogram, linkage
%matplotlib inline
def euler_distance(point1: np.ndarray, point2: list) -> float:
"""
计算两点之间的欧式距离,支持多维
"""
distance = 0.0
for a, b in zip(point1, point2):
distance += math.pow(a - b, 2)
return math.sqrt(distance)
class ClusterNode(object):
"""
:param vec: 保存两个数据聚类后形成新的中心
:param left: 左节点
:param right: 右节点
:param distance: 两个节点的距离
:param id: 用来标记哪些节点是计算过的
:param count: 这个节点的叶子节点个数
"""
def __init__(self, vec, left=None, right=None, distance=-1, id=None, count=1):
self.vec = vec
self.left = left
self.right = right
self.distance = distance
self.id = id
self.count = count
class Hierarchical(object):
def __init__(self, k = 1):
assert k > 0
self.k = k
self.labels = None
def fit(self, x):
nodes = [ClusterNode(vec=v, id=i) for i,v in enumerate(x)]
distances = {}
point_num, future_num = np.shape(x) # 特征的维度
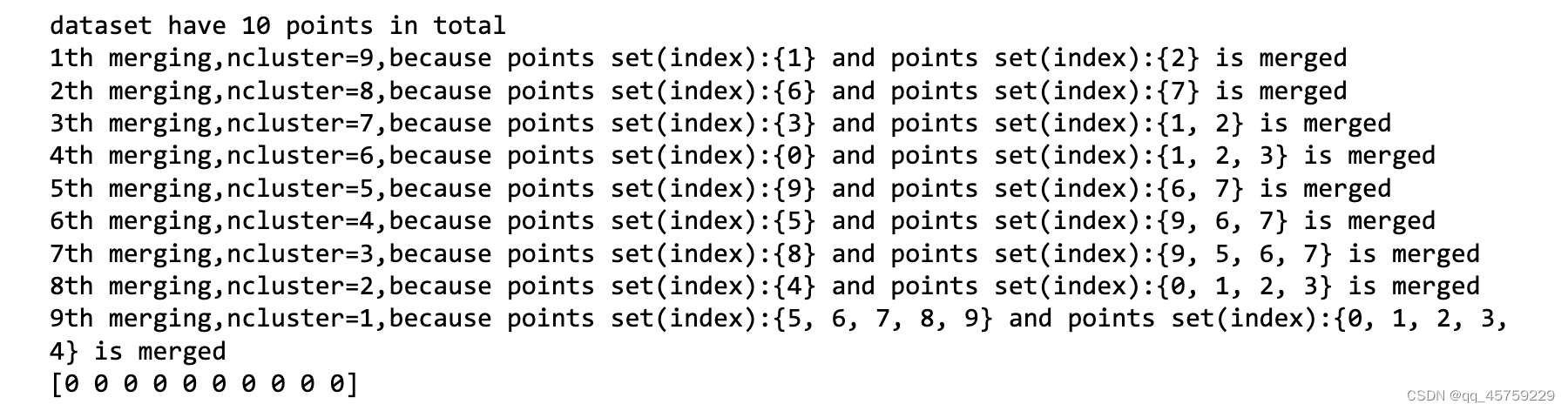
print("dataset have {} points in total".format(point_num))
merge_step=0
self.labels = [ -1 ] * point_num
currentclustid = -1
while len(nodes) > self.k:
merge_step+=1
min_dist = math.inf
nodes_len = len(nodes)
closest_part = None # 表示最相似的两个聚类
for i in range(nodes_len - 1):
for j in range(i + 1, nodes_len):
# 为了不重复计算距离,保存在字典内
d_key = (nodes[i].id, nodes[j].id)
if d_key not in distances:
distances[d_key] = euler_distance(nodes[i].vec, nodes[j].vec)
d = distances[d_key]
if d < min_dist:
min_dist = d
closest_part = (i, j)
# 合并两个聚类
part1, part2 = closest_part
node1, node2 = nodes[part1], nodes[part2]
###
set1=set()
set1=self.child_set(node1,set1)
#print(set1)
set2=set()
set2=self.child_set(node2,set2)
#print(set2)
print("{}th merging,ncluster={},because points set(index):{} and points set(index):{} is merged".format(merge_step,nodes_len-1,set1,set2))
new_vec = [ (node1.vec[i] * node1.count + node2.vec[i] * node2.count ) / (node1.count + node2.count) for i in range(future_num)]
new_node = ClusterNode(vec=new_vec,
left=node1,
right=node2,
distance=min_dist,
id=currentclustid,
count=node1.count + node2.count)
currentclustid -= 1
del nodes[part2], nodes[part1] # 一定要先del索引较大的
nodes.append(new_node)
self.nodes = nodes
self.calc_label()
def calc_label(self):
"""
调取聚类的结果
"""
for i, node in enumerate(self.nodes):
# 将节点的所有叶子节点都分类
self.leaf_traversal(node, i)
def leaf_traversal(self, node: ClusterNode, label):
if node.left == None and node.right == None:
self.labels[node.id] = label
if node.left:
self.leaf_traversal(node.left, label)
if node.right:
self.leaf_traversal(node.right, label)
def child_set(self,node:ClusterNode,s:set()):
if node.left == None and node.right == None:
s.add(node.id)
return s
else:
return self.child_set(node.left,s.copy()) | self.child_set(node.right,s.copy())
if __name__ == '__main__':
# iris = load_iris()
# my = Hierarchical(4)
# my.fit(iris.data)
# print(np.array(my.labels))
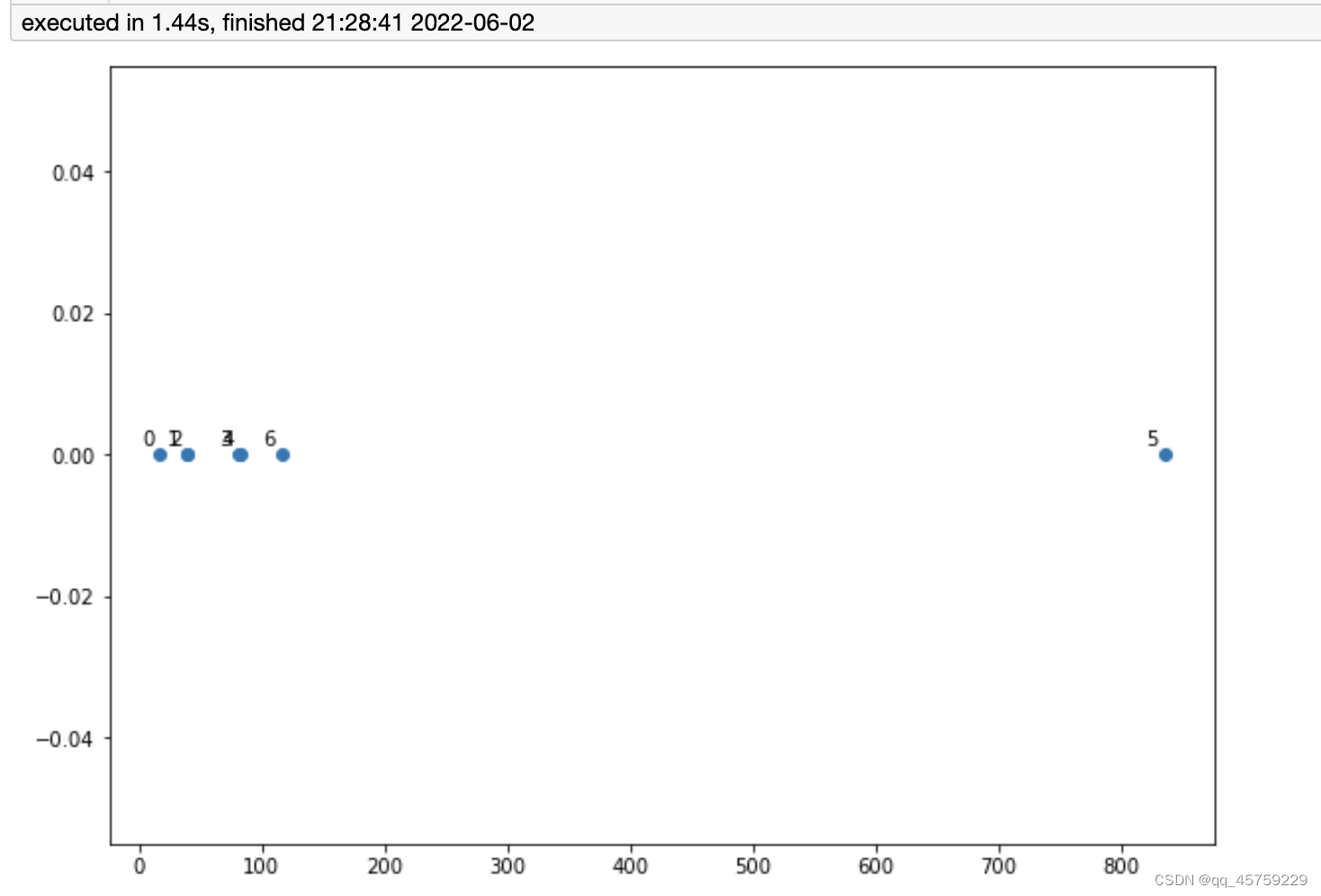
data = [[16.9,0],[38.5,0],[39.5,0],[80.8,0],[82,0],[834.6,0],[116.1,0]]
###################################################################
###################################################################
###################################################################
X=np.array(data) # list没有办法做切片,但是numpy array可以,所以需要转换数据类型
labels = range(0, X.shape[0]) # 都从0开始数起
plt.figure(figsize=(10, 7))
plt.subplots_adjust(bottom=0.1)
plt.scatter(X[:,0],X[:,1], label='True Position')
for label, x, y in zip(labels, X[:, 0], X[:, 1]):
plt.annotate(
label,
xy=(x, y), xytext=(-3, 3),
textcoords='offset points', ha='right', va='bottom')
plt.show()
###################################################################
###################################################################
###################################################################
my = Hierarchical(1)
my.fit(data)
print(np.array(my.labels))
## 我与sklearn画图对比一下下啦
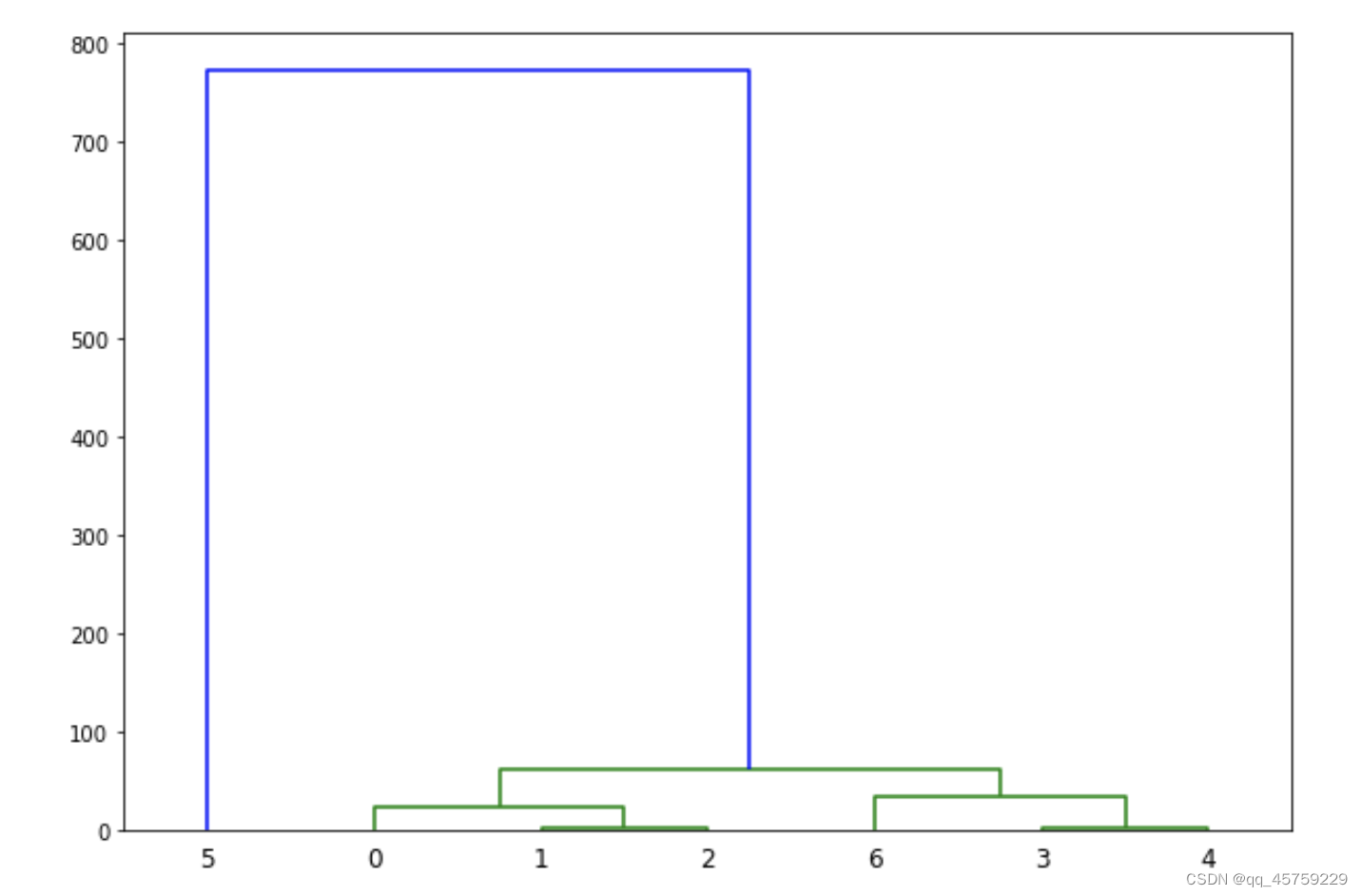
linked = linkage(X,method="centroid")
labelList = range(0, X.shape[0])
plt.figure(figsize=(10, 7))
dendrogram(linked,
orientation='top',
labels=labelList,
distance_sort='descending',
show_leaf_counts=True)
plt.show()
## 可以看到这个结果是一致的
# 注意树高表示距离,树越高,距离越远
结果如下

测试案例2
if __name__ == '__main__':
# iris = load_iris()
# my = Hierarchical(4)
# my.fit(iris.data)
# print(np.array(my.labels))
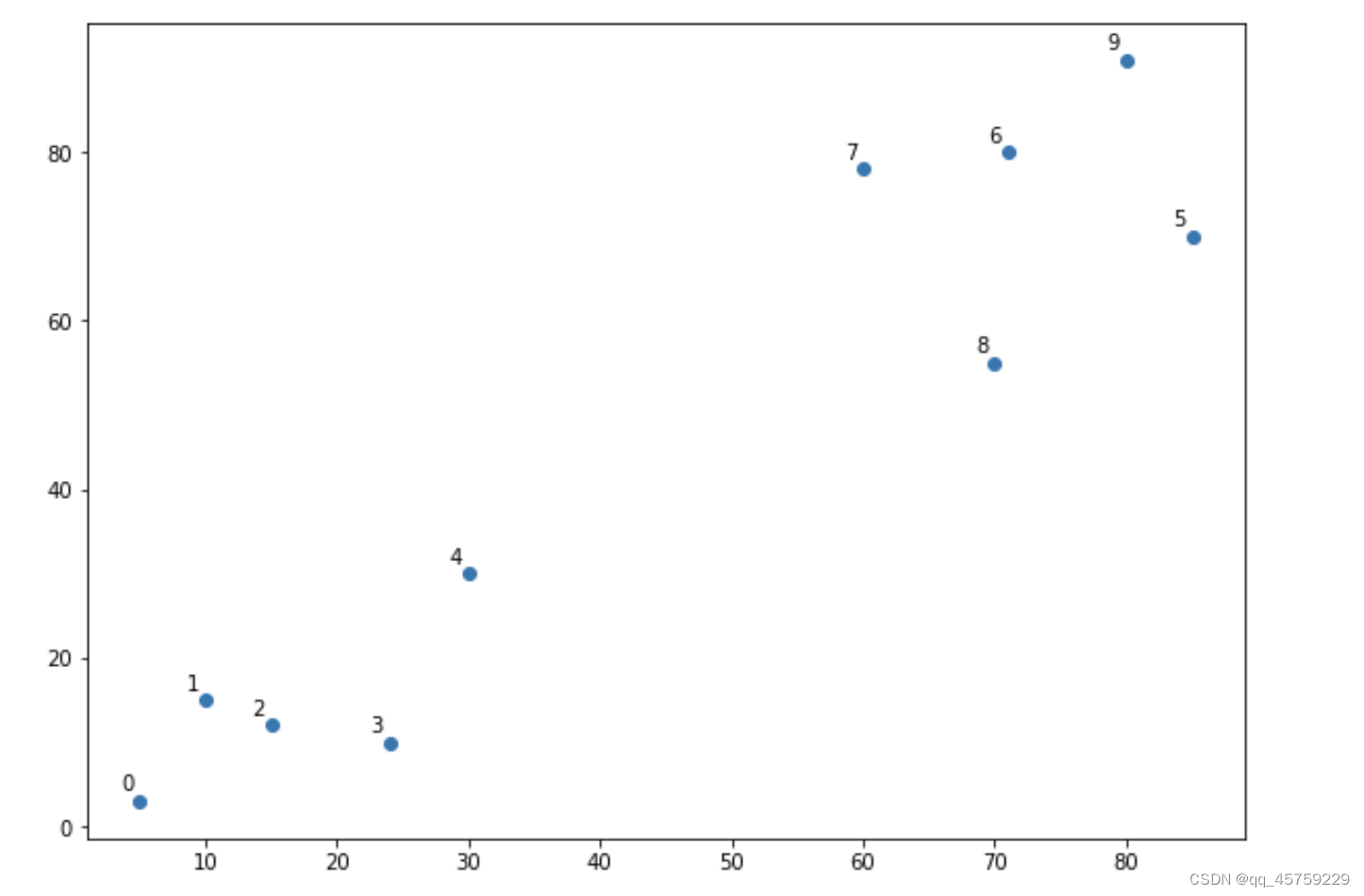
data = np.array([[5,3],
[10,15],
[15,12],
[24,10],
[30,30],
[85,70],
[71,80],
[60,78],
[70,55],
[80,91],])
###################################################################
###################################################################
###################################################################
X=np.array(data) # list没有办法做切片,但是numpy array可以,所以需要转换数据类型
labels = range(0, X.shape[0]) # 都从0开始数起
plt.figure(figsize=(10, 7))
plt.subplots_adjust(bottom=0.1)
plt.scatter(X[:,0],X[:,1], label='True Position')
for label, x, y in zip(labels, X[:, 0], X[:, 1]):
plt.annotate(
label,
xy=(x, y), xytext=(-3, 3),
textcoords='offset points', ha='right', va='bottom')
plt.show()
###################################################################
###################################################################
###################################################################
my = Hierarchical(1)
my.fit(data)
print(np.array(my.labels))
## 我与sklearn画图对比一下下啦
linked = linkage(X,method="centroid") ## 这个地方需要修改这个method连接部分
labelList = range(0, X.shape[0])
plt.figure(figsize=(10, 7))
dendrogram(linked,
orientation='top',
labels=labelList,
distance_sort='descending',
show_leaf_counts=True)
plt.show()
结果如下
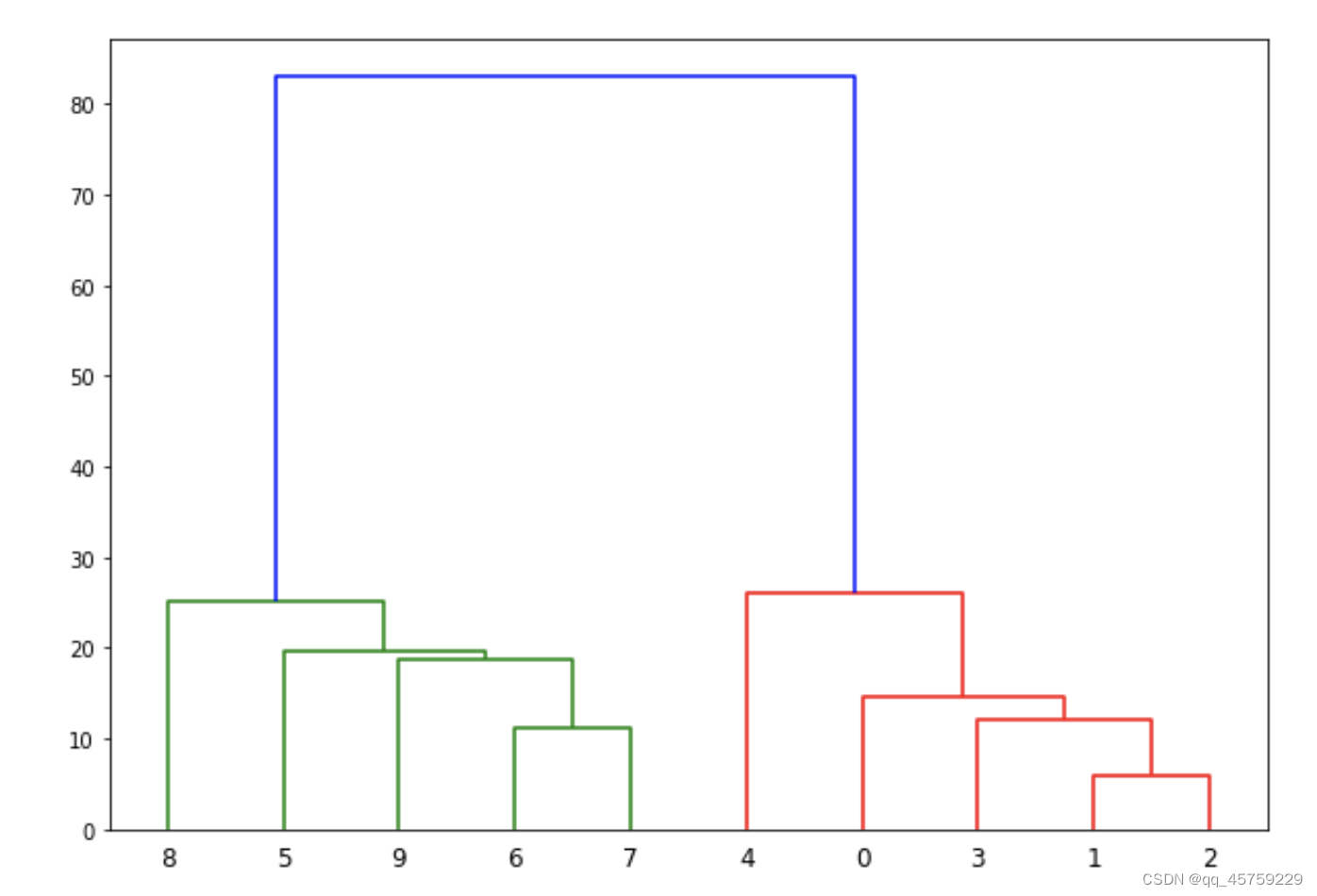
我之所要研究这个,其实就是说我想自定义这个分层聚类图,我今天终于算搞出来怎么自定义我自己的合并算法了
参考博客
https://blog.csdn.net/Tan_HandSome/article/details/79371076
当时我没仔细看这个返回值,就是这个Z的含义
我本来想重新实现R中hclust函数,而且我也已经找到了类似的实现
# 参考
https://github.com/dshonubi/Hierarchical-Agglomerative-Clustering
https://github.com/bwlewis/hclust_in_R/
https://github.com/Agu-Caleb/hierarchical-agglomerative-clustering
## 截
但是上天眷顾我,我在这里又看到了一个相关问题的解答
https://stackoverflow.com/questions/56259202/reordering-the-high-level-clusters-from-seaborn-clustermap-results

这种做法突然让我意识到我可以自定义Z(其实就是linkage),就可以达到我想要的结果,于是我就重新去看了之前翻的那篇博客,果然,我测试的结果大道了
自定义使用Z 案例1
from scipy.cluster.hierarchy import dendrogram, linkage,fcluster
from matplotlib import pyplot as plt
import numpy as np
Z=np.array([[0,1,0.1,2]]) ## 可以看到,
fig = plt.figure(figsize=(5, 3))
dn = dendrogram(Z)
plt.show()
结果如下
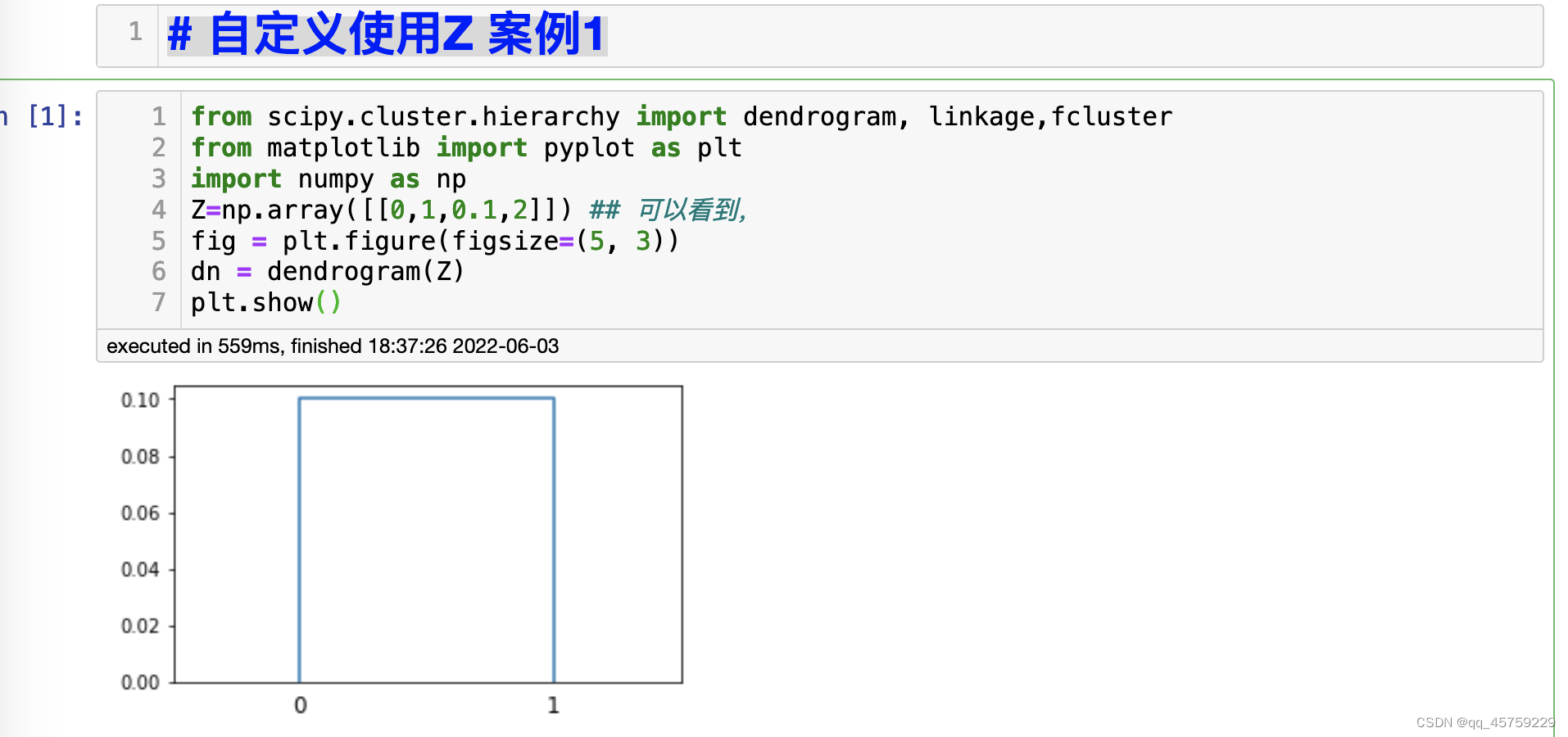 # 自定义使用Z 案例2
# 自定义使用Z 案例2
Z=np.array([[0,1,0.1,2],
[3,2,0.5,3]]) ## 可以看到
fig = plt.figure(figsize=(5, 3))
dn = dendrogram(Z)
plt.show() # 可以看这个图,
# 首先得搞明白这个逻辑,怎么说呢
# 这里我是自己定义好Z的,看起来好像我每次新增一个点进行画图
# 但是其实并不能这么理解,这个对新产生的类簇标号很重要
# 类簇标号总是这么产生的,假设一开始的数据点有n个
# 那么最开始饿类簇标号是0,1,2,...n-1
# 当第一次合并后,新的类簇标号就是n,
# 第二次合并后,新的类簇标号就是n+1,
# 依次类推
# 所以再回过头来看我新构造的Z,第一行显然就是0,1进行合并,这个没什么
# 那么第二行 3,2到底是什么意思呢
# 这里需要这么理解,就是说我首先对数据集中三个点进行层次聚类,那么第三个点的标号就是2
# 那么3是哪来的呢,因为首先数据集中有三个点,类的标号就已经占到了0,1,2,
# 由于第一步进行了合并,那么就会产生一个新类,他的标号自然就排到了3
# 所以第二行的意思就是第一行合并产生的新类和点2进行合并,图像也正如所料
结果如下
 # 自定义使用Z,案例3
# 自定义使用Z,案例3
# 现在这个图就 变成了这个样子
Z=np.array([[ 5. , 6. , 0.1 , 2. ],
[ 2. , 7. , 0.2 , 2. ],
[ 0. , 4. , 0.5 , 2. ],
[ 1. , 8. , 1.15470054, 3. ],
[ 9. , 10. , 2.12132034, 4. ],
[ 3. , 12. , 4.11096096, 5. ],
[11. , 13. , 14.07183949, 8. ]])
## 这个合并的关系
fig = plt.figure(figsize=(5, 3))
dn = dendrogram(Z)
plt.show() # 可以看这个图
结果如下
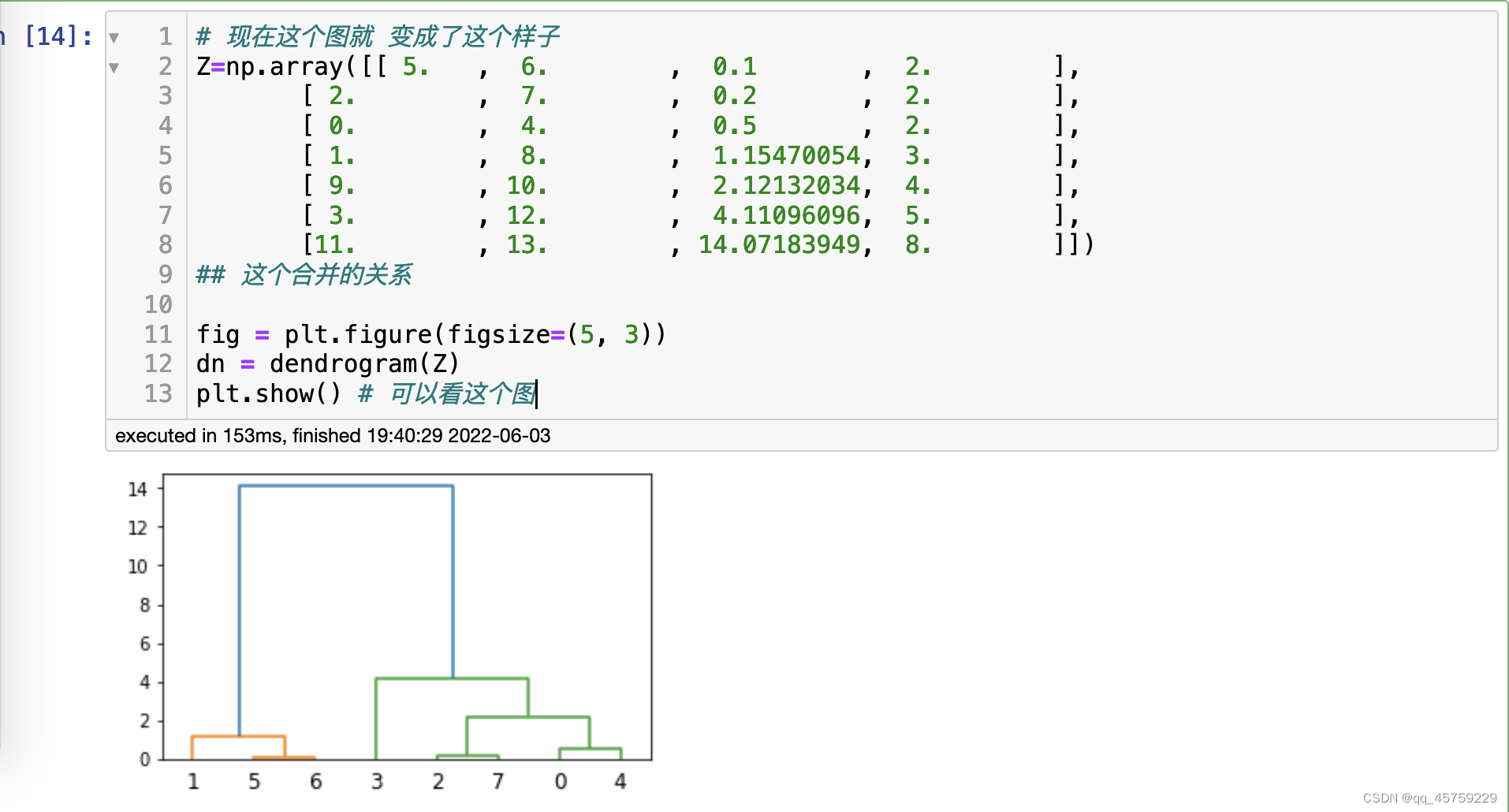
自定义使用Z 案例4
Z=np.array([[0,1,0.1,2],
[3,2,0.5,3],
[4,5,1.0,4]]) ## 注意这个写法,Z必须是一个连续的过程,也就是说从n类合并到1类
fig = plt.figure(figsize=(5, 3))
dn = dendrogram(Z)
plt.show() # 可以看这个图,
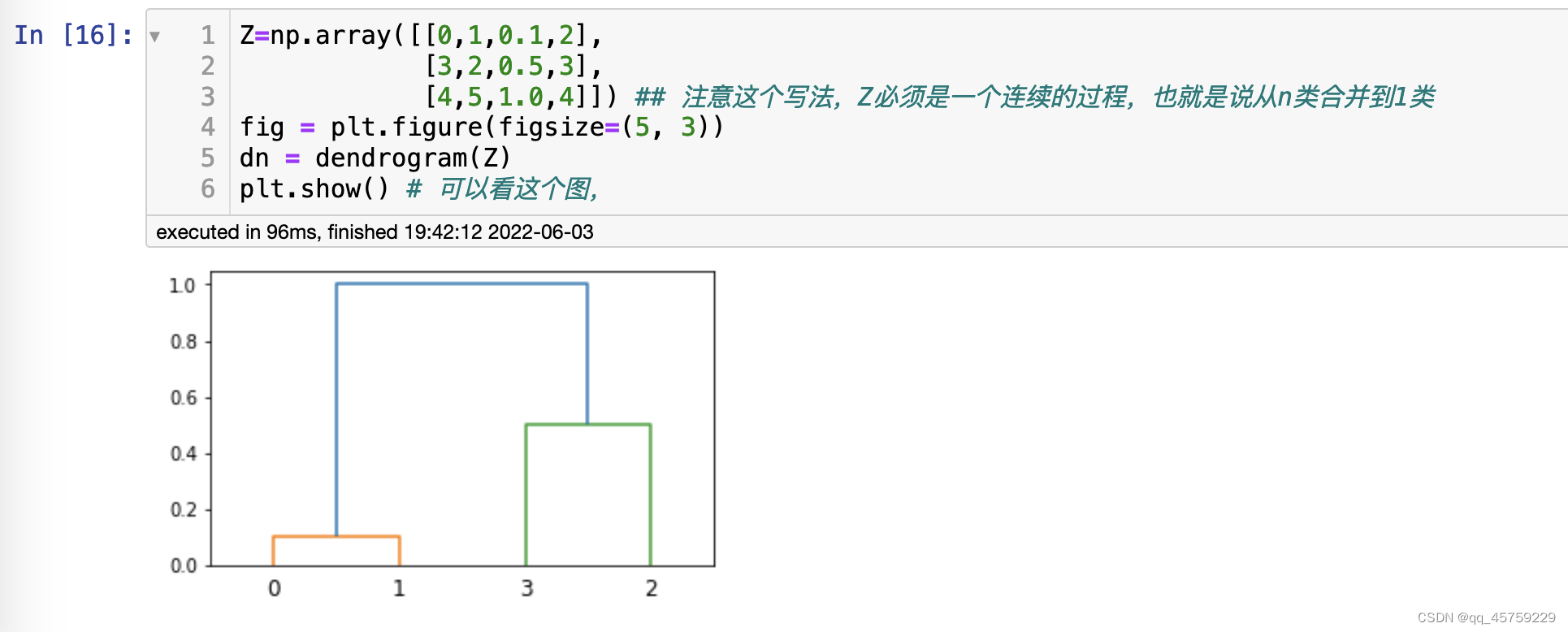
自定义使用Z 案例5
Z=np.array([[ 1. , 9. , 0.1 , 2. ], # (1,9)-12
[ 0. , 2. , 0.2 , 2. ], # (0,2)-13
[ 3. , 13. , 0.3 , 3. ], # (0,2,3)-14
[ 6. , 8. , 0.4 , 2. ], # (6,8)-15
[ 12. , 10. , 0.5 , 3. ], # (1,9,10)-16
[ 5. , 7. , 0.6 , 2. ], # (5,7)-17
[15. , 17. , 0.7 , 4. ], #(5,6,7,8)-18
[4. , 16. , 0.8 , 4. ], #(4,1,9,10)-19
[11. ,14. , 0.9 , 4. ], #(11,0,2,3)-20
[18. ,19. , 1.0 , 8. ], #(5,6,7,8,1,4,9,10)-21
[20. ,21. , 1.1 , 12. ]])
fig = plt.figure(figsize=(5, 3))
dn = dendrogram(Z)
plt.show() # 这个图是我想要的
结果如下

自定义Z结合clustermap
import pandas as pd
import seaborn as sns
cor_matrix=pd.read_csv("test.csv",index_col=0)
#cor_matrix
Z=np.array([[ 1. , 9. , 0.1 , 2. ], # (1,9)-12
[ 0. , 2. , 0.2 , 2. ], # (0,2)-13
[ 3. , 13. , 0.3 , 3. ], # (0,2,3)-14
[ 6. , 8. , 0.4 , 2. ], # (6,8)-15
[ 12. , 10. , 0.5 , 3. ], # (1,9,10)-16
[ 5. , 7. , 0.6 , 2. ], # (5,7)-17
[15. , 17. , 0.7 , 4. ], #(5,6,7,8)-18
[4. , 16. , 0.8 , 4. ], #(4,1,9,10)-19
[11. ,14. , 0.9 , 4. ], #(11,0,2,3)-20
[18. ,19. , 1.0 , 8. ], #(5,6,7,8,1,4,9,10)-21
[20. ,21. , 1.1 , 12. ]])
#fig = plt.figure(figsize=(5, 3))
#dn = dendrogram(Z)
#plt.show() # 可以看这个图
cp=sns.clustermap(cor_matrix,col_linkage=Z,row_linkage=Z,cmap="Blues")
cp.ax_row_dendrogram.set_visible(False)
plt.savefig("./treeplot.png")
plt.show()
最终的结果如下
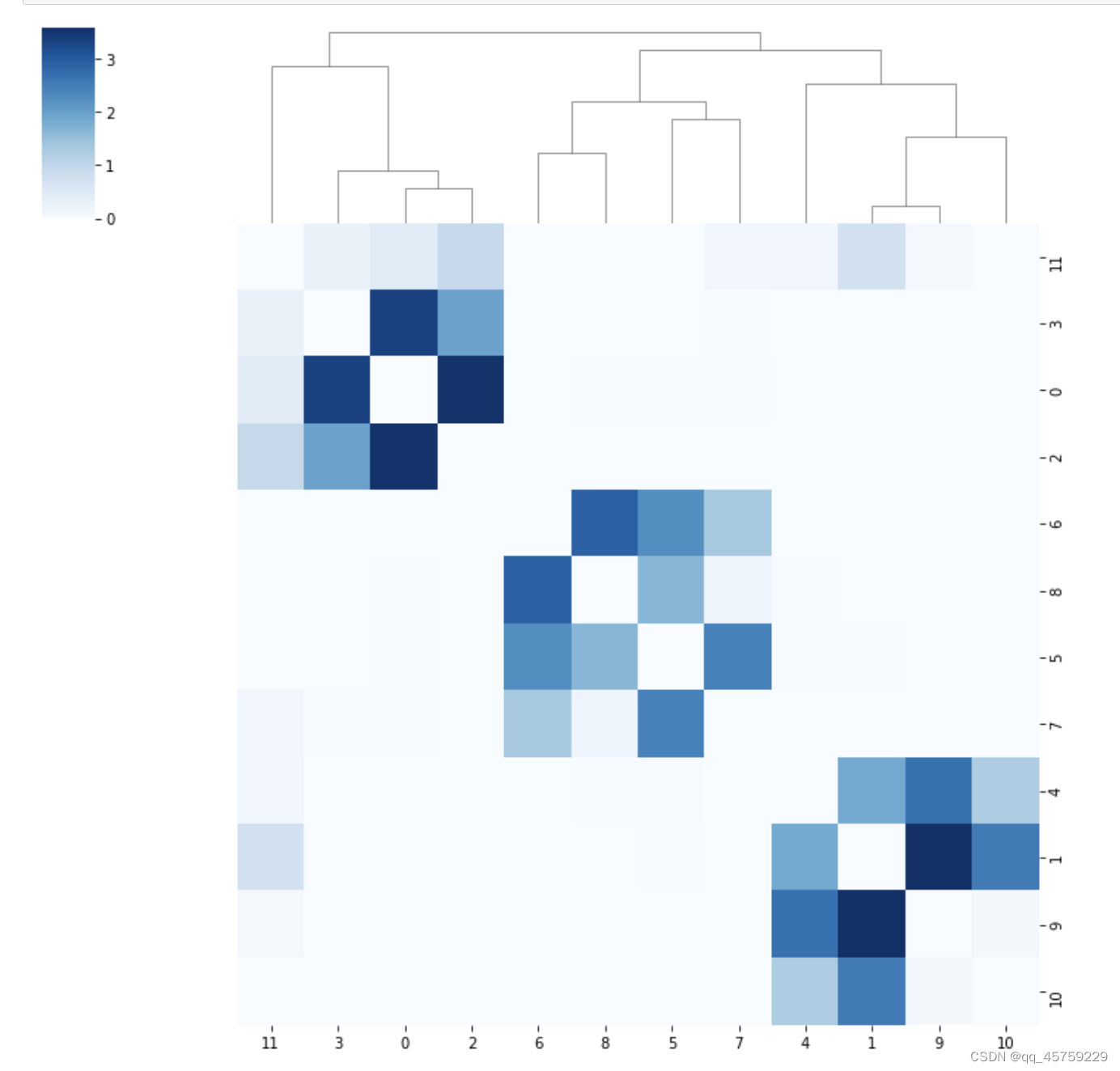 这个图就是我想要的,现在我的问题就变的很简单了,在我的算法的过程中,把合并的Z二维矩阵自动构造出来就可以了,这个是很简单的事情。嘿嘿
这个图就是我想要的,现在我的问题就变的很简单了,在我的算法的过程中,把合并的Z二维矩阵自动构造出来就可以了,这个是很简单的事情。嘿嘿























 4万+
4万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








