引言
蛋白质数据库(Protein Data Bank, PDB)文件是生物大分子结构研究中的核心工具之一。PDB 文件格式广泛用于存储蛋白质、核酸及其相关配体的三维结构信息,这些结构通过 X 射线晶体学、核磁共振(NMR)等实验方法解析并记录。研究人员可以通过这些文件,获取详细的原子坐标、化学组成以及生物大分子与配体之间的相互作用信息。
PDB 文件格式不仅在蛋白质结构研究中至关重要,还在药物设计、分子对接和大分子相互作用研究中得到了广泛应用。其详细的结构信息帮助科学家理解分子功能和相互作用的机制,并指导实验设计和计算模拟。
除了 PDB 格式,还有其他文件格式(如 CIF、SDF、SMILES)同样在生物大分子和小分子化合物的研究中扮演着重要角色。这些格式将在后续的文章中详细介绍。
1 PDB 文件结构概述
PDB 文件由一系列固定格式的文本条目组成,每一行都包含特定类型的信息。这些条目详细记录了蛋白质或其他大分子结构的所有关键信息,包括原子坐标、序列信息、实验数据和其他生物学相关的注释。PDB 文件的主要结构可以分为两部分:Header(头部)和主结构(主链与配体的原子坐标)。
-
Header 部分:这个部分记录了文件的元数据信息,包括结构的标题、解析方法、解析分辨率、作者信息、修饰和突变记录等。它为理解该分子结构的背景提供了重要的参考。
-
主结构部分:主要记录蛋白质、核酸或其他大分子的原子坐标信息,其中包括 ATOM 和 HETATM 两类条目。ATOM 主要记录蛋白质主链的原子坐标,而 HETATM 记录的是配体、辅因子、金属离子等分子的坐标。
PDB 文件中的数据结构清晰且标准化,这使得它能够被多种软件解析和使用,例如 PyMOL、Chimera 等三维可视化工具。
为了更好地理解 PDB 文件的格式与内容,本文将使用 PDB 代码为 7A0C 的结构作为示例。这是一个多链结构,包含多个蛋白质链和配体,能清晰展示 PDB 文件中关于链、配体和相互作用的描述方式。接下来我们将详细解析 7A0C.pdb 文件的 Header 部分以及其余内容。
RCSB PDB - 7A0C: X-ray structure of NikA from Escherichia coli in complex with Fe-6-Me2-BPMCN
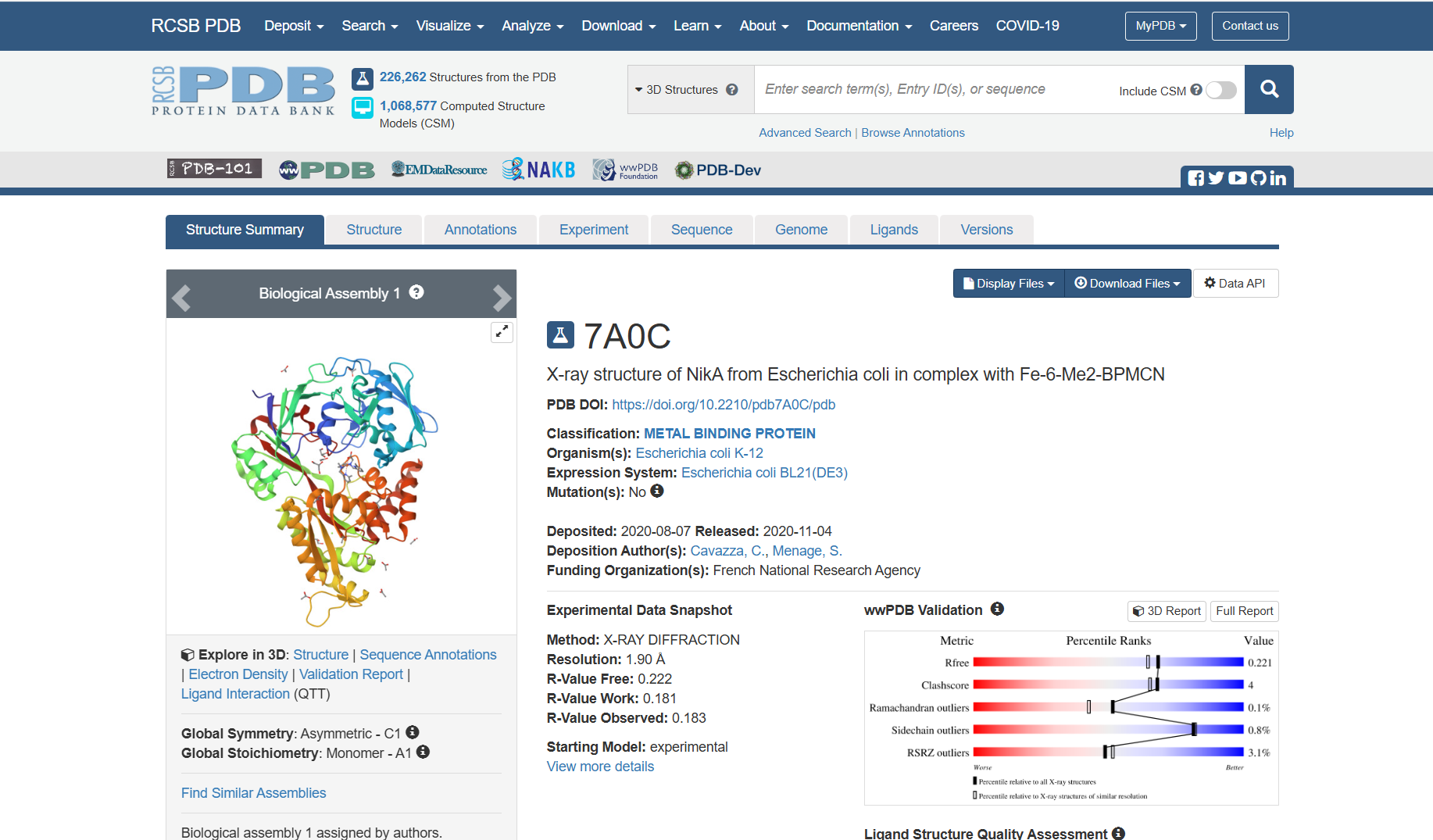
2 PDB Header 文件详解
要下载 PDB 文件的 header 信息,可以从 RCSB PDB 数据库网站找到目标 PDB 结构页面,并选择下载格式为 PDB Format (Header)。如图所示(见下方示例图),从下拉菜单中选择此选项,即可获取文件的头部信息。
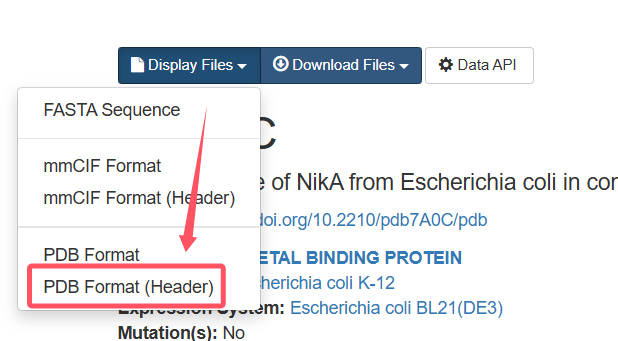
2.1 基础结构描述部分
以 7A0C.pdb 为例,其 Header 文件的基础结构描述部分如下:
HEADER METAL BINDING PROTEIN 07-AUG-20 7A0C
TITLE X-RAY STRUCTURE OF NIKA FROM ESCHERICHIA COLI IN COMPLEX WITH FE-6-
TITLE 2 ME2-BPMCN
COMPND MOL_ID: 1;
COMPND 2 MOLECULE: NICKEL-BINDING PERIPLASMIC PROTEIN;
COMPND 3 CHAIN: A, B;
COMPND 4 ENGINEERED: YES
SOURCE MOL_ID: 1;
SOURCE 2 ORGANISM_SCIENTIFIC: ESCHERICHIA COLI (STRAIN K12);
SOURCE 3 ORGANISM_TAXID: 83333;
SOURCE 4 STRAIN: K12;
SOURCE 5 GENE: NIKA, B3476, JW3441;
SOURCE 6 EXPRESSION_SYSTEM: ESCHERICHIA COLI BL21(DE3);
SOURCE 7 EXPRESSION_SYSTEM_TAXID: 469008
KEYWDS ARTIFICIAL METALLOENZYME, CROSS-LINKED ENZYME CRYSTAL, SULFOXIDATION,
KEYWDS 2 METAL BINDING PROTEIN
EXPDTA X-RAY DIFFRACTION
这个基础结构描述部分包含了以下几个关键字段:
- HEADER: 提供文件的基本信息,例如结构类型、解析日期和 PDB 代码。
- 在 7A0C 示例中:描述的是一个 METAL BINDING PROTEIN(金属结合蛋白),表明这个分子与金属有结合功能。解析日期是 2020年8月7日,PDB 代码是 7A0C。
- TITLE: 描述结构的具体名称和功能。
- 在 7A0C 示例中:标题是 X-RAY STRUCTURE OF NIKA FROM ESCHERICHIA COLI IN COMPLEX WITH FE-6-ME2-BPMCN,说明了这个结构是通过 X 射线晶体学 方法解析的,结构包含了 Nika 蛋白 和 FE-6-ME2-BPMCN 的复合物。
- COMPND: 说明结构中的分子组成,包括分子 ID、分子名称、链信息等。
- 在 7A0C 示例中:
- MOL_ID: 1 表示这是第一个分子。
- MOLECULE 是 Nickel-binding periplasmic protein,即镍结合周质蛋白。
- CHAIN 表明该分子有两个链,分别是 A 和 B 链。
- ENGINEERED: YES 表示这个蛋白是经过基因工程改造的。
- 在 7A0C 示例中:
- SOURCE: 描述分子的来源,例如生物物种、菌株、基因信息等。
- 在 7A0C 示例中:
- MOL_ID: 1 对应第一个分子。
- ORGANISM_SCIENTIFIC 是 Escherichia coli (strain K12),即大肠杆菌 K12 株。
- ORGANISM_TAXID 是大肠杆菌的 NCBI 分类号 83333。
- GENE 表示该蛋白由 NikA 基因(B3476 和 JW3441)编码。
- EXPRESSION_SYSTEM 表示该蛋白是在大肠杆菌 BL21(DE3) 中表达的。
- 在 7A0C 示例中:
- KEYWDS: 提供一些与结构相关的关键词,帮助检索。
- 在 7A0C 示例中:关键词包括 ARTIFICIAL METALLOENZYME(人工金属酶)、CROSS-LINKED ENZYME CRYSTAL(交联酶晶体)、SULFOXIDATION(硫氧化反应)和 METAL BINDING PROTEIN(金属结合蛋白)。
- EXPDTA: 描述用于解析该结构的实验方法。
- 在 7A0C 示例中:使用的实验方法是 X-RAY DIFFRACTION(X 射线衍射)。
2.2 出版与修订部分
AUTHOR C.CAVAZZA,S.MENAGE
REVDAT 4 31-JAN-24 7A0C 1 REMARK
REVDAT 3 23-DEC-20 7A0C 1 JRNL
REVDAT 2 25-NOV-20 7A0C 1 JRNL
REVDAT 1 04-NOV-20 7A0C 0
JRNL AUTH S.LOPEZ,C.MARCHI-DELAPIERRE,C.CAVAZZA,S.MENAGE
JRNL TITL A SELECTIVE SULFIDE OXIDATION CATALYZED BY HETEROGENEOUS
JRNL TITL 2 ARTIFICIAL METALLOENZYMES IRON@NIKA.
JRNL REF CHEMISTRY V. 26 16633 2020
JRNL REFN ISSN 0947-6539
JRNL PMID 33079395
JRNL DOI 10.1002/CHEM.202003746
- AUTHOR: 列出了参与解析结构的研究人员。
- 在 7A0C 示例中:作者包括 C. Cavazza 和 S. Menage,这些科学家参与了该蛋白质结构的研究和解析工作。
- REVDAT: (修订日期)记录了 PDB 文件的修订历史和变更细节。
- 在 7A0C 示例中:
- REVDAT 4 表示这是第 4 次修订,修订日期为 2024年1月31日,与文件的
REMARK部分有关。 - REVDAT 3 和 REVDAT 2 表示的是与该结构相关的文章的修订,分别是在 2020年12月23日 和 2020年11月25日。
- REVDAT 1 是最早的修订,日期为 2020年11月4日,是该结构的初始提交。
- REVDAT 4 表示这是第 4 次修订,修订日期为 2024年1月31日,与文件的
- 在 7A0C 示例中:
- JRNL: 列出了与该结构相关的出版物信息,包括作者、文章标题、期刊名称、卷号、页码等。此信息对于引用该结构的研究文献非常重要。
- 在 7A0C 示例中:
- AUTH 列出了文章的作者,包括 S. Lopez、C. Marchi-Delapierre、C. Cavazza 和 S. Menage。
- TITL 给出了文章的标题,标题是 A selective sulfide oxidation catalyzed by heterogeneous artificial metalloenzymes iron@Nika,描述了该结构的研究成果,重点在于铁@Nika 的人工金属酶的硫化物氧化催化功能。
- REF 表示这篇文章发表在期刊 Chemistry(化学),卷号 26,页码 16633,发表年份为 2020。
- REFN 列出了该期刊的 ISSN 编号为 0947-6539。
- PMID 是文章的 PubMed ID,33079395,可用于在 PubMed 数据库中查找这篇文章。
- DOI 是文章的数字对象标识符,10.1002/CHEM.202003746,可以用于直接在线查阅文章。
- 在 7A0C 示例中:
2.3 REMARK 部分简要说明
PDB 文件中的 REMARK 部分用于补充各种与结构相关的重要信息,这些信息往往涉及到实验方法、数据处理细节以及特殊的结构注释。不同的 REMARK 编号对应不同类型的注释,以下是一些常见的 REMARK 编号及其作用的简要说明:
REMARK 2: 分辨率信息
- 描述:
REMARK 2提供了结构的分辨率信息,常以 Ångström(Å)为单位,表示数据的精确度。 - 7A0C 中的例子:
REMARK 2 RESOLUTION. 1.90 ANGSTROMS表示该结构的分辨率为 1.90 Å。
REMARK 3: 精修信息
- 描述:
REMARK 3记录了结构精修过程中使用的程序和参数,包括使用的算法、模型的自由 R 值(Free R Value)、工作集的 R 值等。 - 7A0C 中的例子:精修过程使用了 PHENIX 程序,工作集的 R 值 为 0.182,自由 R 值 为 0.222。
REMARK 4: 格式合规性
- 描述:
REMARK 4表示该 PDB 文件符合特定的格式标准。一般用于确认文件是否符合最新的 PDB 格式规范。 - 7A0C 中的例子:
REMARK 4 7A0C COMPLIES WITH FORMAT V. 3.30, 13-JUL-11,表明该文件符合 3.30 版本的格式标准。
REMARK 200: 实验细节
- 描述:
REMARK 200提供了与实验相关的细节,如实验类型、数据收集日期、温度、PH值等信息。 - 7A0C 中的例子:实验类型为 X-RAY DIFFRACTION(X 射线衍射),数据收集的日期为 2017年7月13日,温度为 93K。
REMARK 280: 晶体信息
- 描述:
REMARK 280包含晶体的详细信息,如溶剂含量和 Matthews 系数等。 - 7A0C 中的例子:
REMARK 280 SOLVENT CONTENT, VS (%): 45.17表示晶体的溶剂含量为 45.17%。
REMARK 465: 缺失的残基
- 描述:
REMARK 465列出了在实验中无法解析的残基。 - 7A0C 中的例子:缺失的残基包括 ALA A 1 和 VAL A 500 等,表示这些残基在实验中未被解析。
REMARK 500: 几何和立体化学信息
- 描述:
REMARK 500记录了几何和立体化学相关的细节,包括原子之间的距离、二面角等信息。 - 7A0C 中的例子:列出了许多溶剂分子之间的距离,以及与其他原子间的相互作用距离。
需要注意的部分
在深度学习任务中,尤其是与蛋白质结构相关的研究中,REMARK 2、REMARK 3、REMARK 465 和 REMARK 500 是需要特别注意的部分。这些信息会影响数据的质量与模型的表现,建议在选择和处理数据时关注这些字段,以确保数据的可靠性和准确性:
- REMARK 2 和 REMARK 3(分辨率和精修信息):在药物设计和分子对接中,使用高质量的结构数据非常重要,低分辨率的结构可能导致实验偏差。
- REMARK 465(缺失的残基):在使用结构数据进行训练或推理前,缺失残基的信息可能需要处理或补齐,否则可能导致模型预测结果不准确。(注:不一定处理,视任务而定。)
- REMARK 500(几何信息):原子间的几何和立体化学关系在分子间相互作用中非常关键,尤其是在精确预测蛋白质与配体的结合时。几何信息通常涉及蛋白质中的二面角,这些角度描述了蛋白质主链上每个氨基酸残基的旋转,并在三维结构中定义了其折叠和构象。
2.4 序列和参考信息部分
在 PDB 文件中,序列和参考信息部分用于描述与数据库的关联、蛋白质序列的组成以及多肽链的详细信息。它包括一些重要字段,例如 DBREF、SEQRES等。
DBREF:
DBREF 7A0C A 1 502 UNP P33590 NIKA_ECOLI 23 524
DBREF 7A0C B 1 502 UNP P33590 NIKA_ECOLI 23 524
- 7A0C:PDB 结构的编号。
- A/B:这是对应的链,A 和 B 是两个链。
- 1 502:在 PDB 文件中链 A 和链 B 的序列号范围为 1 到 502。
- UNP:指的是蛋白质数据库 UniProt。
- P33590:这是 UniProt ID,即 Nika 蛋白在 UniProt 数据库中的唯一识别号。
- NIKA_ECOLI:该蛋白质的名称,来自大肠杆菌(Escherichia coli)。
- 23 524:这是在 UniProt 序列中的起始位置和结束位置。PDB 文件中这段序列对应的是 UniProt 数据库中的第 23 到 524 个氨基酸。
SEQRES: 提供了每条链的完整氨基酸序列信息,表示该蛋白质的一级序列。
SEQRES 1 A 502 ALA ALA PRO ASP GLU ILE THR THR ALA TRP PRO VAL ASN
SEQRES 2 A 502 VAL GLY PRO LEU ASN PRO HIS LEU TYR THR PRO ASN GLN
SEQRES 3 A 502 MET PHE ALA GLN SER MET VAL TYR GLU PRO LEU VAL LYS
SEQRES 4 A 502 TYR GLN ALA ASP GLY SER VAL ILE PRO TRP LEU ALA LYS
...
- SEQRES:固定的标识符,用于表示序列数据。
- 1 A 502:这是链 A 的序列,长度为 502 个氨基酸。
- ALA ALA PRO…:列出了每个氨基酸的三字母缩写,按顺序排列。
- 多个行:当链的序列过长时,会分成多行表示。在这个例子中,链 A 被分成了多行。
这些氨基酸序列是链 A 和链 B 的全部序列,而 SEQRES 部分列出了完整的一级结构信息,即氨基酸的线性顺序。这些序列在蛋白质折叠过程中会形成二级结构(如 α-螺旋、β-折叠片等),并在 PDB 文件中的 ATOM 和 HETATM 部分显示其三维坐标。

2.5 异质分子部分
在 PDB 文件中,异质分子部分记录了与蛋白质结构相关的非标准分子,通常包括配体、金属离子、辅因子、溶剂分子等。它由多个字段组成,包括 HET、HETNAM、HETSYN 和 FORMUL,共同描述了这些异质分子的化学组成、名称及位置。
此外,通过 FORMUL 和 HETNAM 等字段的化学信息,可以提取配体分子的分子式和化学结构。在后续的文章中,我们将详细讨论如何使用这些信息生成配体分子的 SMILES 表示法,这对于分子对接和药物设计等任务非常重要。
1. HET:
HET 部分列出了每个异质分子的基本信息,包括其名称、所在链、序列编号及原子数量。它为研究者提供了蛋白质结构中配体、金属离子等分子的具体位置信息。
HET FE A 601 1
HET QTT A 602 29
HET ACT A 603 4
HET ACT A 604 4
...
- FE 表示铁离子,位于链 A 的 601 号位置。
- QTT 是一个配体分子,位于链 A 的 602 号位置,包含 29 个原子。
- ACT 是乙酸根离子,位于链 A 的多个位置。
2.HETNAM:
HETNAM 部分记录了异质分子的化学名称,帮助研究者理解每个分子的具体化学成分。
HETNAM FE FE (III) ION
HETNAM QTT 2-[[(1~{S},2~{S})-2-[METHYL-[(6-METHYLPYRIDIN-2-YL)
HETNAM 2 QTT METHYL]AMINO]CYCLOHEXYL]-[(6-METHYLPYRIDIN-2-YL)
HETNAM 3 QTT METHYL]AMINO]ETHANOIC ACID
HETNAM ACT ACETATE ION
HETNAM GOL GLYCEROL
HETNAM MG MAGNESIUM ION
HETNAM CL CHLORIDE ION
HETSYN GOL GLYCERIN; PROPANE-1,2,3-TRIOL
- FE: 铁离子(Fe^3+)
- QTT: 化学名称较为复杂,是一个包含多个部分的配体,完整化学名称在多行中展示。
- ACT: 乙酸根离子。
- GOL: 甘油。
- MG: 镁离子。
- CL: 氯离子。
3.HETSYN:
HETSYN 提供了异质原子的别名或其他化学名称。
HETSYN GOL GLYCERIN; PROPANE-1,2,3-TRIOL
- GOL:异质原子甘油(GOL)的其他化学名称,包括 Glycerin 和 Propane-1,2,3-triol。
4.FORMUL:
FORMUL 提供了异质原子的化学分子式,表示它们的化学组成。
FORMUL 3 FE 2(FE 3+)
FORMUL 4 QTT 2(C23 H32 N4 O2)
FORMUL 5 ACT 9(C2 H3 O2 1-)
FORMUL 14 GOL 9(C3 H8 O3)
FORMUL 19 MG 5(MG 2+)
FORMUL 30 CL CL 1-
FORMUL 31 HOH *795(H2 O)
- FE: 表示结构中有 2 个三价铁离子 (Fe 3+)。
- QTT: 配体 QTT 的化学式为 C23 H32 N4 O2。
- ACT: 乙酸根离子的化学式为 C2 H3 O2 1-,其中有 9 个乙酸根离子。
- GOL: 甘油的化学式为 C3 H8 O3。
- MG: 表示有 5 个二价镁离子 (Mg 2+)。
- CL: 氯离子 (Cl 1-)。
- HOH: 表示 795 个水分子 (H2O)。
数据清洗与任务相关性
在实际任务中,可以根据研究目标决定哪些异质分子需要保留或清洗。例如,在分子对接任务中,配体分子和金属离子可能非常关键,需要保留以确保分子间相互作用的准确预测。而诸如盐离子(如 CL)和大部分水分子(HOH)在多数情况下是晶体化的产物,通常可以清洗掉,除非它们直接参与蛋白质的功能性区域。详细的清洗规则和策略将在后续文章中详细讨论。
2.6 蛋白质二级结构与分子链接信息
这一部分记录了蛋白质的二级结构和蛋白质与其他分子或金属离子的链接信息。具体包括螺旋结构 (HELIX)、片层结构 (SHEET)、原子链接 (LINK) 以及顺式肽键 (CISPEP) 的信息。
1. HELIX
HELIX 描述了蛋白质中的 α-螺旋结构。每一条记录提供了螺旋的起始和终止残基编号,以及螺旋的长度。
HELIX 1 AA1 GLN A 26 TYR A 34 1 9
HELIX 2 AA2 ASP A 80 ASP A 93 1 14
HELIX 3 AA3 ASN A 94 ALA A 99 5 6
HELIX 4 AA4 LEU A 101 GLN A 106 1 6
- HELIX 1:从链 A 的 26 号残基 GLN(谷氨酰胺) 到 34 号残基 TYR(酪氨酸) 形成一个 α-螺旋,长度为 9 个残基。
- HELIX 2:从链 A 的 80 号残基 ASP(天冬氨酸) 到 93 号残基形成一个 α-螺旋,长度为 14 个残基。
2. SHEET
SHEET 描述了蛋白质中的 β-折叠片结构。每一条记录提供了 β-折叠片的起始和终止残基,以及链之间的氢键信息。
SHEET 1 AA1 4 GLU A 5 TRP A 10 0
SHEET 2 AA1 4 LYS A 193 VAL A 198 1 O THR A 195 N THR A 8
SHEET 3 AA1 4 TYR A 175 ARG A 180 -1 N ASP A 176 O PHE A 196
SHEET 4 AA1 4 TRP A 165 LYS A 171 -1 N GLN A 168 O VAL A 177
- SHEET 1:链 A 从 GLU(谷氨酸)5 到 TRP(色氨酸)10 形成 β-折叠片。
- SHEET 2:链 A 从 LYS(赖氨酸)193 到 VAL(缬氨酸)198 形成 β-折叠片。
3. LINK
LINK 描述了蛋白质结构中不同原子或分子之间的键,例如金属离子和蛋白质残基之间的键。
LINK OD1 ASP A 291 MG MG A 617 1555 1555 2.24
LINK O GLU A 320 MG MG B 607 1555 4455 2.66
LINK FE FE A 601 N3 QTT A 602 1555 1555 2.26
LINK FE FE A 601 N2 QTT A 602 1555 1555 2.71
- LINK 1:链 A 的 天冬氨酸 (ASP) 291 的 OD1 原子与 镁离子 (Mg) 形成一个键,键长为 2.24Å。
- LINK 3:链 A 的 铁离子 (FE 601) 与配体 QTT 602 的 N3 原子之间形成一个键,键长为 2.26Å。
4. CISPEP
CISPEP 描述了蛋白质中存在的顺式肽键,通常发生在某些特殊的残基之间,如脯氨酸。
CISPEP 1 THR A 23 PRO A 24 0 5.39
CISPEP 2 THR A 23 PRO A 24 0 -1.42
CISPEP 3 ARG A 137 PRO A 138 0 7.00
CISPEP 4 ALA A 258 PRO A 259 0 -1.69
- CISPEP 1:链 A 的 THR 23 和 PRO 24 之间形成顺式肽键,二面角为 5.39°。
- CISPEP 3:链 A 的 ARG 137 和 PRO 138 之间形成顺式肽键,二面角为 7.00°。
2.7 晶体结构与坐标信息
这一部分记录了蛋白质晶体的几何信息和坐标变换矩阵。具体包括晶体的晶格参数 (CRYST1) 和坐标变换矩阵 (ORIGX, SCALE)。
1. CRYST1
CRYST1 提供了晶体的晶格参数和空间群信息。这些参数用于描述晶体的三维结构。
CRYST1 86.809 93.796 124.222 90.00 90.00 90.00 P 21 21 21 8
- 晶格参数:a = 86.809 Å, b = 93.796 Å, c = 124.222 Å,晶体角度为 90°。
- 空间群:晶体的空间群为 P 21 21 21。
2. ORIGX 和 SCALE
ORIGX 和 SCALE 提供了用于从晶体学坐标系转换到实际三维坐标系的转换矩阵。
ORIGX1 1.000000 0.000000 0.000000 0.00000
ORIGX2 0.000000 1.000000 0.000000 0.00000
ORIGX3 0.000000 0.000000 1.000000 0.00000
SCALE1 0.011520 0.000000 0.000000 0.00000
SCALE2 0.000000 0.010661 0.000000 0.00000
SCALE3 0.000000 0.000000 0.008050 0.00000
- ORIGX:用于描述原始坐标的变换矩阵,通常用于恢复蛋白质的实际晶体学坐标。
- SCALE:用于将晶体学坐标系缩放到实际三维坐标的矩阵。
3 PDB 整体文件详解
要下载 PDB 文件的整体信息,可以从 RCSB PDB 数据库网站找到目标 PDB 结构页面,并选择下载格式为 PDB Format。如图所示(见下方示例图),从下拉菜单中选择此选项,即可获取文件的所有原子坐标信息、配体信息、二级结构等完整数据。
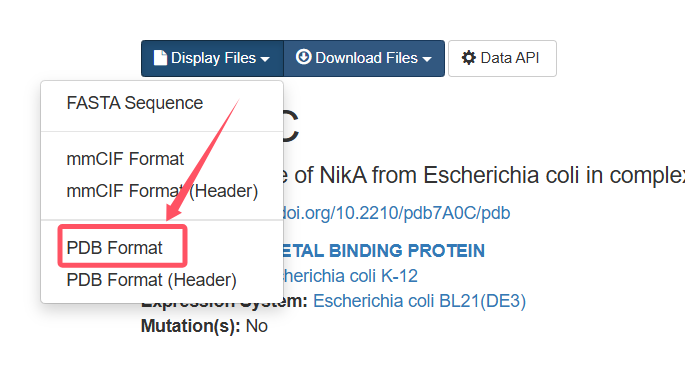
3.1 ATOM 条目格式解释
ATOM 条目是 PDB 文件中最核心的信息之一,它描述了每个标准氨基酸的原子坐标。每一列都包含了与该原子相关的重要数据。以7A0C为例,前几行的数据解释如下:
| 序号 | 原子名称 | 残基名称 | 链标识符 | 残基序号 | X 坐标 | Y 坐标 | Z 坐标 | 占有率 | B 因子 | 元素符号 |
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | N | ALA | A | 2 | 17.406 | 18.438 | 57.995 | 1.00 | 74.03 | N |
| 2 | CA | ALA | A | 2 | 18.345 | 17.403 | 57.577 | 1.00 | 68.42 | C |
| 3 | C | ALA | A | 2 | 18.325 | 17.235 | 56.060 | 1.00 | 60.96 | C |
| 4 | O | ALA | A | 2 | 17.346 | 16.740 | 55.499 | 1.00 | 42.05 | O |
| 5 | CB | ALA | A | 2 | 18.022 | 16.085 | 58.263 | 1.00 | 57.36 | C |
| 6 | N | PRO | A | 3 | 19.413 | 17.639 | 55.398 | 1.00 | 67.14 | N |
| 7 | CA | PRO | A | 3 | 19.459 | 17.584 | 53.931 | 1.00 | 59.38 | C |
列表头说明:
- 序号: 每个原子的唯一标识符。
- 原子名称: 该原子的名称,如氮(N)、碳(C)等。
- 残基名称: 该原子所在的氨基酸残基名称,如丙氨酸 (ALA)、脯氨酸 (PRO)。
- 链标识符: 所在的链的标识符,通常为 A、B 等。
- 残基序号: 该原子所在的残基在链中的序号。
- X 坐标, Y 坐标, Z 坐标: 原子的三维空间坐标。
- 占有率: 该原子在该位置的占有率,通常为 1.00。
- B 因子: 该原子的 B 因子(温度因子),表示该原子的热运动程度。
- 元素符号: 原子的元素类型。
TER 条目解释
TER 是 终止标识符(Terminator),用于标识一个链的结束。在 PDB 文件中,每条链的最后一个残基后面通常会有一个 TER 条目,表示该链的结束。
TER 4029 PRO A 499
- TER:表示链的终止。
- 4029:这是一个序号,表示这个
TER条目在整个文件中的位置。 - PRO A 499:
PRO表示脯氨酸,A表示链 A,499表示链 A 中的最后一个残基的编号是 499。
3.2 HETATM 条目解释
在 PDB 文件 中,HETATM 条目用于描述非标准的原子或分子,它们通常不属于标准氨基酸或核苷酸序列。这些分子包括配体、金属离子、溶剂分子、辅因子等。以下是一些常见的 HETATM 分子类型及其在 PDB 文件中的描述:
1.金属离子:
金属离子是非常常见的 HETATM 类型,它们经常作为蛋白质或酶的辅因子,参与催化反应或结构稳定性。常见的金属离子包括:
- FE(铁离子):通常是参与氧化还原反应的辅因子,例如在血红蛋白或细胞色素中。
- MG(镁离子):广泛参与多种生物过程,如在 ATP 的水解反应中。
- ZN(锌离子):常见于锌指蛋白,参与 DNA 结合或酶的催化。
- CA(钙离子):在钙信号传导和结构稳定性中发挥重要作用。
示例:
| 原子类型 | 原子编号 | 残基名称 | 元素 | 链ID | 残基编号 | X 坐标 | Y 坐标 | Z 坐标 | 占有率 | 温度因子 | 元素符号 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| HETATM | 8160 | MG | MG | B | 607 | 22.538 | 40.509 | 0.534 | 1.00 | 51.75 | MG |
| HETATM | 8009 | FE | FE | A | 601 | 1.378 | -4.520 | 34.830 | 0.56 | 55.59 | FE |
2.配体分子:
配体是与蛋白质结合的小分子,它们可能是药物、抑制剂、底物或天然产物。在分子对接或药物设计研究中,配体是非常重要的分子类型。
示例:
HETATM 8106 N1 QTT B 601 34.781 20.935 19.575 1.00 37.90 N
HETATM 8107 C1 QTT B 601 35.482 19.762 19.090 1.00 35.53 C
...
HETATM 8133 C01 QTT B 601 39.029 23.525 21.162 1.00 45.98 C
HETATM 8134 C02 QTT B 601 36.395 26.037 19.839 1.00 57.72 C
3.辅因子:
辅因子是帮助酶发挥功能的非蛋白质分子,通常是小分子或金属离子,它们有助于催化过程的完成。常见的辅因子包括:
- FAD(黄素腺嘌呤二核苷酸):常见于氧化还原反应。
- FMN(黄素单核苷酸):与氧化还原酶结合的辅因子。
- COA(辅酶 A):参与脂肪酸和能量代谢。
许多金属离子可以作为辅因子。例如,铁(Fe²⁺/Fe³⁺)、镁(Mg²⁺)、锌(Zn²⁺)、钙(Ca²⁺)等金属离子常常作为辅因子帮助酶进行催化反应。它们在许多生物反应中发挥重要作用。但辅因子并不仅限于金属离子。有机分子如辅酶 A、NAD⁺ 也可以是辅因子,而这些分子并不是金属离子。
示例(非7A0C示例):
HETATM 7001 FAD A 601 3.672 5.021 45.982 1.00 45.39 C
4.水分子:
水分子以 HOH 代号表示,通常是在蛋白质结晶过程中形成的水分子。在某些情况下,水分子可以与蛋白质活性位点或配体相互作用,但大多数水分子在分析过程中是可以去除的。
示例:
HETATM 8165 O HOH A 701 0.857 18.984 52.768 1.00 46.72 O
HETATM 8166 O HOH A 702 4.227 -12.126 25.934 1.00 32.04 O
5.常见小分子:
一些常见的小分子,虽然不是标准氨基酸或核苷酸,但在研究蛋白质相互作用或代谢途径时很重要。
- GOL(甘油):常用于蛋白质结晶时的溶剂。
- ACT(乙酸根):通常与酶活性位点结合。
- SO4(硫酸根离子):常见于生物学实验中的溶剂或缓冲液。
示例:
HETATM 8157 O2 GOL B 606 27.252 22.337 11.422 1.00 55.90 O
HETATM 8164 CL CL B 611 13.329 32.596 33.670 1.00 46.00 CL
数据清洗建议
在处理生物分子数据(特别是 PDB 文件)时,数据清洗的目标通常是移除冗余或不相关的分子,以简化结构,便于后续分析。水分子和小溶剂分子通常是最常被移除的部分,而金属离子、辅因子和配体分子则根据研究的具体需求和目标来决定是否保留。
典型数据清洗策略:
-
移除水分子:水分子常作为晶体结构中存在的背景分子,除非它们与研究相关(如蛋白质-水分子相互作用或水在蛋白质功能中的特殊作用),否则通常会被移除。
-
移除小溶剂分子:小分子溶剂(如乙醇、乙酸等)通常用于结晶过程中,但在大多数蛋白质功能研究中无关紧要,因此通常会被移除,除非它们对蛋白质的稳定性或活性有特殊作用。
-
保留金属离子:金属离子如镁(Mg²⁺)、锌(Zn²⁺)、铁(Fe²⁺/Fe³⁺)等,通常作为关键辅因子参与蛋白质的催化反应或维持其结构稳定,因此在数据清洗时一般会被保留。
-
保留辅因子:辅因子如铁硫簇、血红素等,往往在蛋白质的生物化学功能中扮演重要角色,因此在分析中应予保留。
-
保留配体分子:配体是与蛋白质结合的分子,通常是药物设计、分子对接等研究的核心。因此,在研究蛋白质-配体相互作用时,配体是非常重要的部分,应予以保留。
注意:不同研究的清洗策略会根据需求有所不同。例如,如果研究的重点是蛋白质主链,可以采取更严格的清洗策略,移除所有与主链无关的分子或原子。这意味着你可以清除水分子、小溶剂分子、金属离子、辅因子和配体分子,仅保留与蛋白质主链相关的部分。需要注意的是,即使在 PDB 文件的
ATOM记录中,也可能包含不属于主链的原子(如氨基酸的侧链原子)。这些细节可以在后续文章中进一步探讨。
3.3 CONECT 条目解释
CONECT 条目用于描述分子中的原子之间的连接关系,通常是共价键或金属离子与其他分子之间的键。在 CONECT 记录中,原子通过编号(对应于 ATOM 或 HETATM 条目中的原子序号)进行连接。这些条目对于定义配体、金属离子、辅因子等分子与蛋白质的相互作用至关重要,尤其在小分子与蛋白质对接的场景下,定义了非标准氨基酸和蛋白质主链之间的连接关系。
示例:
CONECT 2359 8105
CONECT 5844 8162
CONECT 8009 8010 8013 8029 8030
CONECT 8009 8036 8414
CONECT 8010 8009 8011 8027 8034
- CONECT 2359 8105:表示 原子 2359 和 原子 8105 之间存在一个键(例如,金属离子与配体之间的连接)。
- CONECT 8009 8010 8013 8029 8030:表示 原子 8009 与 原子 8010, 8013, 8029, 8030 之间分别存在连接。这可以用于定义配体或金属离子如何与蛋白质或其他小分子相互作用。
使用场景:
- 配体与蛋白质:如果分析配体与蛋白质的结合方式,CONECT 条目可以帮助你明确配体与哪些蛋白质残基或金属离子有化学键或相互作用。
- 金属离子:在金属离子与蛋白质或配体之间的相互作用中,CONECT 条目明确表示了金属离子与哪些原子或分子有连接。
3.4 MASTER 条目
MASTER 是 PDB 文件的摘要条目,用于提供文件的概述信息。这些信息包含了文件中不同条目的计数,帮助检查文件是否完整和正确。这个条目不包含具体的分子结构信息,而是一个统计汇总。
总结
通过本文对 PDB 文件的详细解析,我们深入探讨了 PDB 文件结构的各个组成部分,包括 Header 部分的元数据信息、主结构中记录的原子坐标,以及与实验方法和数据相关的 REMARK 部分等。这些信息不仅为研究人员提供了丰富的结构数据,还能帮助我们在蛋白质研究、药物设计和分子对接任务中做出更加准确的判断和决策。PDB 文件作为三维结构数据的载体,其标准化和广泛应用使其成为生物大分子研究中的重要工具。在下一篇文章中,我们将继续探讨其他常见的文件格式,如 SDF、SMILES,以及它们在小分子化合物研究中的应用。

























 782
782

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










