1. 文库复杂性
ChIPseq 中的一个潜在噪声源是 ChIPseq 库在 PCR 步骤中的过度放大。这可能会导致大量重复读取,从而混淆峰值调用。

2. 重复
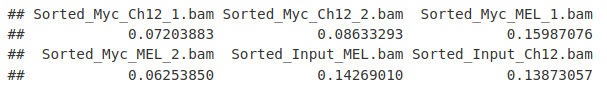
我们应该比较样本之间的重复率,以确定任何经历过度放大的样本,从而确定复杂性较低的可能性。
flattagcounts() 函数报告可以报告重复的数量和总映射读取,因此我们可以从那里计算我们的重复率。
myFlags <- flagtagcounts(myQC)
myFlags["DuplicateByChIPQC", ]/myFlags["Mapped", ]
3. 跨基因 reads 富集
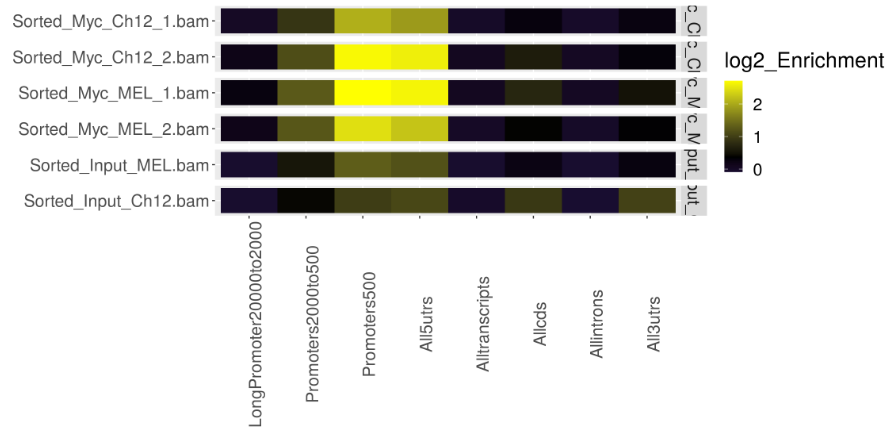
我们还可以使用 ChIPQC 使用 plotRegi() 函数来查看我们在基因特征上的 reads 分布。
在这里,与输入样本相比,我们预计 ChIPseq 信号在 5'UTR 和启动子中更强。
p <- plotRegi(myQC)
本文由 mdnice 多平台发布




















 2883
2883











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








