Multiobjective Evolutionary Algorithms: A Comparative Case Study and the Strength Pareto Approach
摘要
该算法有以下四个特点
a. 在第二个连续更新的种群中存储非支配解。(精英保留机制)
b. 根据支配其的非支配点的数量来评估个体的固定性
c. 使用帕累托优势关系来保留人口多样性。
d. 结合聚类程序以减少非支配解的集合而不破坏其特征。
简单总结一下,该算法主要在两个方便有所研究,(1) 每个个体的适应度求解方式,(2)使用聚类来减少多余点。
该文的主要研究在第五部分,如果想对其他算法有所了解请参考前面的章节,如果想直接了解该算法,可直接跳到第五部分,这里也是对该算法进行分析。
算法
该算法保留了其他多目标算法的特点:
存储非支配解。
用帕累托支配的概念来分配适应度。
用聚类来减少非支配解的数目。
该算法与其他算法不同点:
包含了上面三点(。。。)。
适应度的分配仅依靠非支配解集,其他支配解互不相关。
外部非支配解集合中的所有解都参与选择。
为了保持多样性采用了一种新的小生境方式,这种方式不需要距离计算,是基于帕累托的。
算法步骤:
step1. 生成初始解P,建立一个空集合(精英解集合)P’。
step2. 复制P->P’。
step3. 从P’删除被其他个体支配的解。
step4. 如果P’ 中的解超过给定的最大值N’ ,用聚类方式修剪P’ 。
step5. 计算P’ 和P 中个体的适应值。
step6. 从P+P’中选出个体放到交配池(这里应该为P)中。
step7. 对交配池里面的元素进行杂交变异。
step8. 判断循环。
细节
是适应度分配方式:
step1. 对P’ 中的元素i给定一个Si值(强度),Si与该点支配P集合里点的数目有关,设定n代表该点(P’中的非支配点)支配的点(在P中的点)的数目,N代表P集合中点的总数目。则Si= n/(N+1)。
step2. P集合里的点的适应度等于P’中支配该点的非支配解的Si值相加+1。
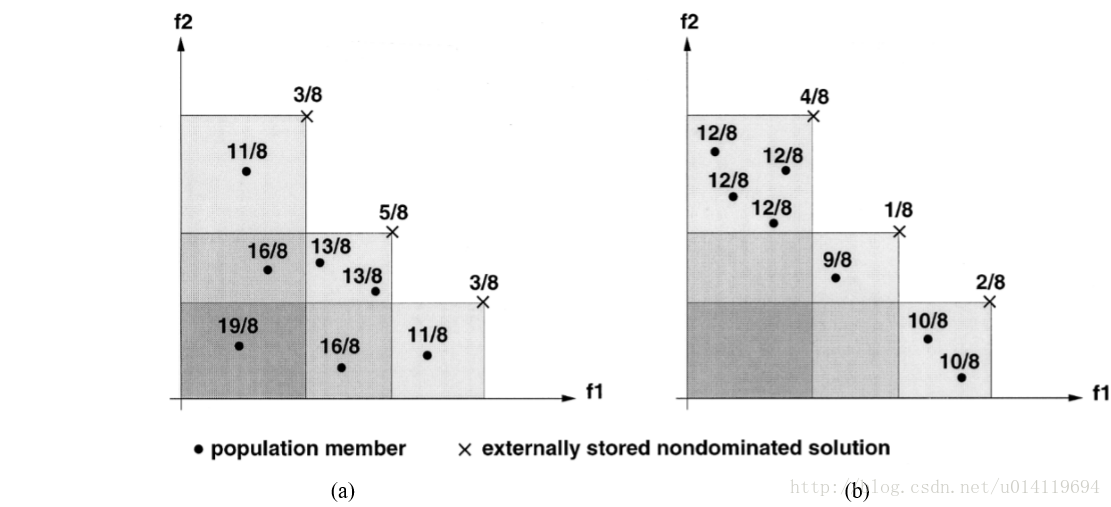
用图来分析,先看a图,每个x点表示P’里的非支配解,实心原点表示P里面的支配解。最上面的支配解3/8是因为该点支配3个点,P’中的点个数是8。11/8是因为该点被右上角的x点支配,x点的Si值是3/8,3/8+1=11/8。其他值同样如此。
聚类算法
step1. 初始化聚类集合C,每一个P’中的点构建一个聚类集合。
step2. 如果C里聚类的个数少于N’(给定的最大值),go step5。
step3. 计算两个聚类c1,c2没对点间的距离。
step4. 选择最小的距离,合并这两个聚类。
step5. 选取聚类中心(该聚类中聚其他点的平均距离最小)作为代表解。








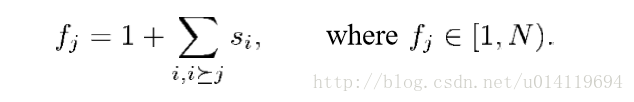
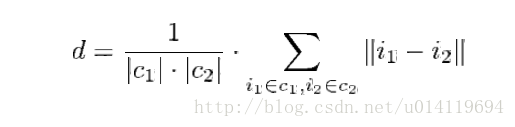














 3232
3232

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








