什么是环状RNA?
环状RNA(circular RNA,circRNA)是一类没有5’帽子和3'poly (A)尾巴的环状闭合非编码RNA分子。

如上图所示,基因组上的外显子1,2,3,4,5,6可以转录形成线性RNA分子,例如右侧中间的线性RNA分子由1,2,3,4,5,6号外显子形成,右侧下边的线性RNA分子由1,2,4,5,6号外显子形成(不同的转录本)。这些线性RNA带有5’帽子和3’polyA尾巴。
而大部分由外显子组成的环状RNA(exonic circRNA)是通过外显子的反向剪切(back splicing)形成的。例如左侧的4个circRNAs:2号外显子自身环化形成(上);3号和2号外显子环化形成(左);5号和4号外显子环化形成(右);4号、2号和3号外显子环化形成。这些环状RNA没有polyA尾巴,不受核酸外切酶影响,不易降解,表达更稳定。
注:还有内含子等来源的环状,但是绝大部分还是外显子来源的。
CeRNA机制
CircRNA的作用机制之一就是吸附miRNA,影响miRNA的表达,进而影响基因的表达,即所谓的ceRNA(竞争性内源RNA)假说。
图2. ceRNA机制
如图所示,circRNA以圆形表示其中包含磁铁图标,暗示它可以吸附周围的miRNAs分子(miRNA分子以梳子形式表示);而miRNA又可以靶向mRNA,从而抑制mRNA的表达。这样就形成了3分子间的相互作用轴(circRNA-miRNA-mRNA)。当circRNA表达升高时,它吸附的miRNA就多,导致靶向mRNA的miRNA减少,mRNA被抑制的程度降低,从而mRNA的表达量也升高。这种低-高-低,或者高-低-高的现象就是ceRNA假说。
环状RNA吸附miRNA的预测
现在一般以常规的miRNA靶基因预测软件,例如targetscan,miRanda等来预测circRNA与miRNA的结合。我们微生信也推出了在线版的miRanda预测模块,可以预测miRNA与circRNA的结合。预测结果一般是miRNA在circRNA上的结合坐标。我们可以形象化地用圆形结合图来展示环状RNA与所吸附的miRNA的定性(哪些miRNA结合)和定位(结合位置)关系。
环状RNA吸附miRNA结合图
例如这篇:CircNT5E Acts as a Sponge of miR-422a to Promote Glioblastoma Tumorigenesis文章的fig6A。
图中以棕色圆形表示circNT5E,以红色小矩形表示miRNA结合。今天我们使用微生信平台来在线模仿下这个图。当然,这个图中用的miRNA是用小矩形表示的,不够圆滑。
1,打开circRNA-miRNA吸附圆形图绘图页面
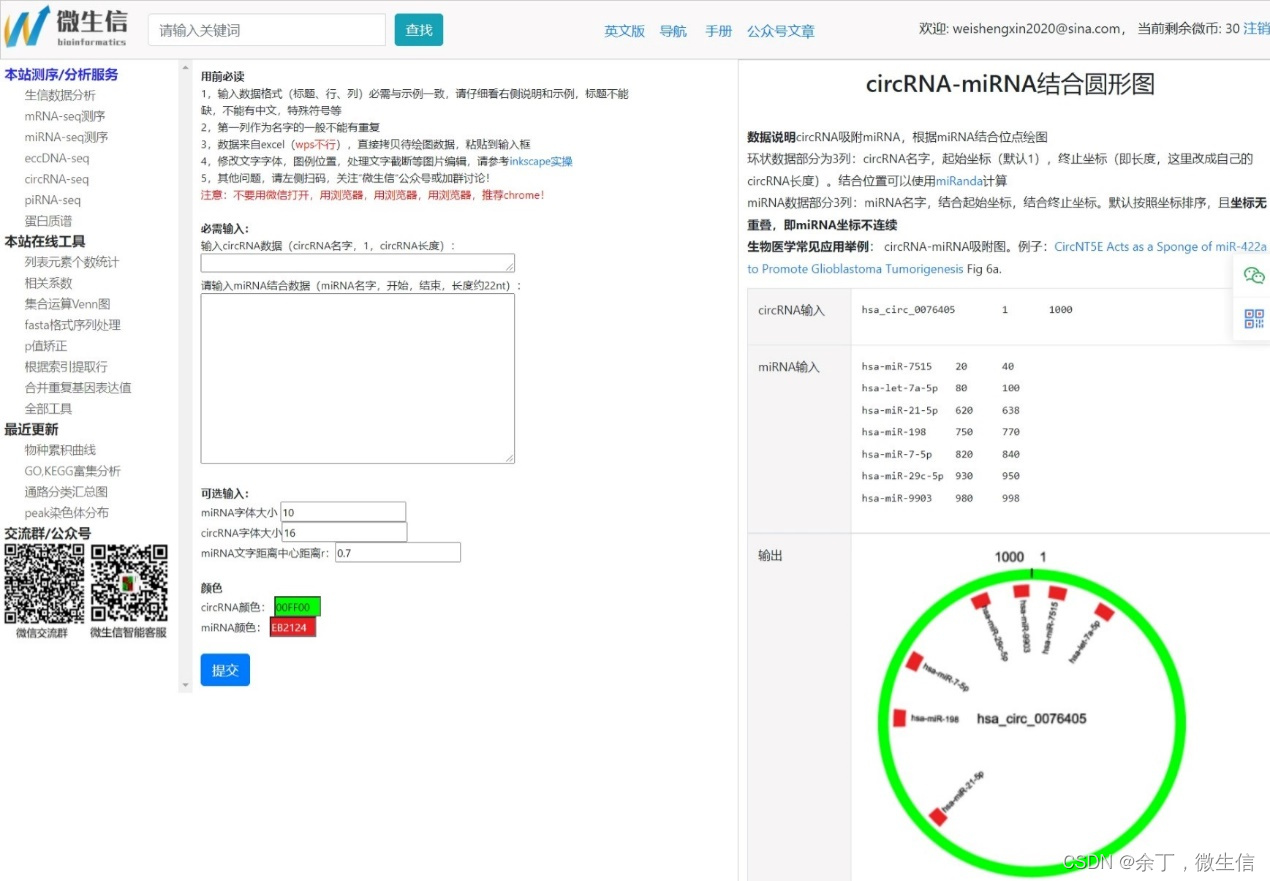
2,查看示例数据
输入数据包括两个:
第一个是circRNA名字及坐标。这里默认起始坐标是1,终止坐标为你的circRNA的长度。
第二个是miRNA的名字及坐标。第一列是名字,第二列是结合的起始坐标,第三列是结合的终止坐标。
注意1:部分预测软件,例如miranda预测结果中只有一个坐标,因此,我们要加上miRNA自身的长度,构成第三列。三列齐全才能绘图,因为后台程序没有判断miRNA长度的功能。
注意2:miRNA的结合位置要从小到大排列下才能作图。
注意3:本模块仅处理一个miRNA轨道的情况,对于miRNA吸附位点坐标有重复的本模块也不能处理。例如,若两个miRNA的结合位置是:20-40和25-45,由于中间25-40都是重复的,所有需要两个轨道才能绘图。
3,拷贝并粘贴数据
4,修改参数,并提交
该模块可以修改颜色,字体大小等。基本可以满足90%的需求了。如果你有另外10%的需求,请直接联系我们进行定制!

没有预览就是没有出图,这时请参考前面的注意事项,检查输入数据格式!
若存在文字截断,需要修改字体等,可以使用inkscape软件进行操作。
微生信简介circRNA
微生信云平台(微生信-您身边的数据分析、可视化专家)以140多款在线作图、在线数据分析为基本方式,致力于0代码分析科研数据,0代码展示数据结果,帮助生命科学、医学等领域的科研工作者更便捷地分析数据,了解数据,挖掘数据背后的生物医学意义,辅助科研,促进知识传播。
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








