作者简介:taoyan:伪码农,R语言爱好者,爱开源。
文章来源:R语言可视化学习笔记之添加p-value和显著性标记(已与黄老师协商过转载)
在R语言在生物信息学的实际应用中,常常需要添加p-value和显著性标记。很多同学对于添加P-value这个问题一直一知半解,殊不知,不同的类型添加P-value选择的方法也是不同的。
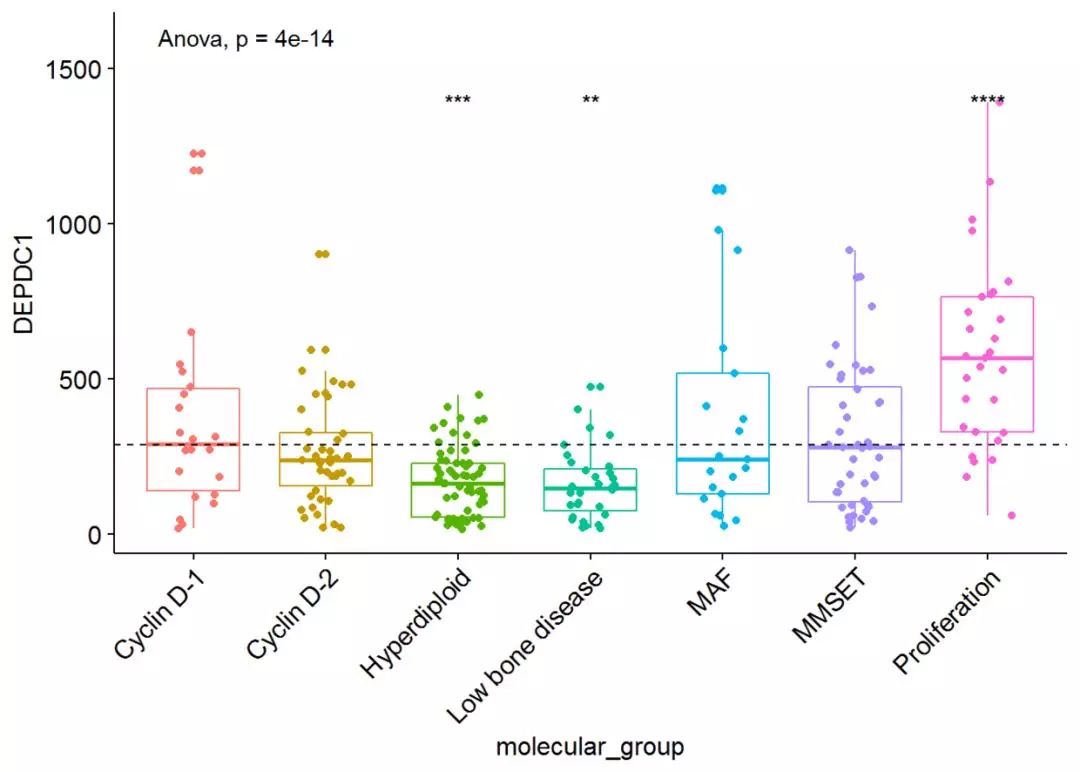
本文将详细介绍如何通过ggpubr包为ggplot图添加p-value以及显著性标记。利用数据集ToothGrowth进行演示。
#先加载包
library(ggpubr)
#加载数据集ToothGrowth
data("ToothGrowth")
head(ToothGrowth)
## len supp dose
## 1 4.2 VC 0.5
## 2 11.5 VC 0.5
## 3 7.3 VC 0.5
## 4 5.8 VC 0.5
## 5 6.4 VC 0.5
## 6 10.0 VC 0.5比较方法
R中常用的比较方法主要有下面几种:
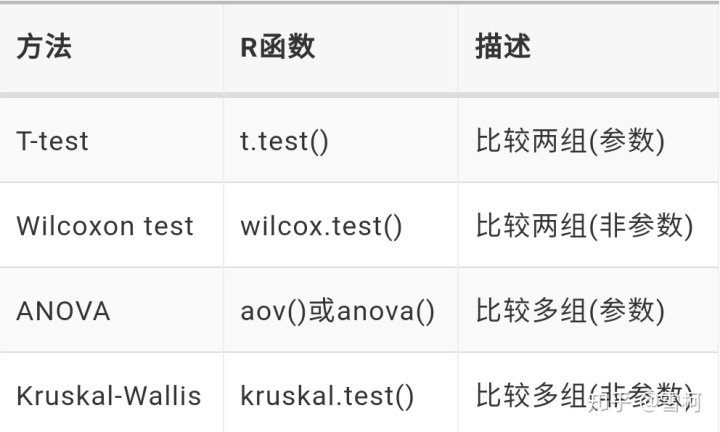
各种比较方法后续有时间一一讲解。
添加p-value
主要利用ggpubr包中的两个函数:
compare_means():可以进行一组或多组间的比较stat_compare_mean():自动添加p-value、显著性标记到ggplot图中
##compare_means()函数
该函数主要用用法如下:
compare_means(formula, data, method = "wilcox.test", paired = FALSE,
group.by = NULL, ref.group = NULL, ...)注释:
- formula:形如x~group,其中x是数值型变量,group是因子,可以是一个或者多个
- data:数据集
- method:比较的方法,默认为"wilcox.test", 其他可选方法为:"t.test"、"anova"、"kruskal.test"
- paired:是否要进行paired test(TRUE or FALSE)
- group_by: 比较时是否要进行分组
- ref.group: 是否需要指定参考组
##stat_compare_means()函数
主要用法:
stat_compare_means(mapping = NULL, comparisons = NULL hide.ns = FALSE,
label = NULL, label.x = NULL, label.y = NULL, ...)注释:
- mapping:由aes()创建的一套美学映射
- comparisons:指定需要进行比较以及添加p-value、显著性标记的组
- hide.ns:是否要显示显著性标记ns
- label:显著性标记的类型,可选项为:p.signif(显著性标记)、p.format(显示p-value)
- label.x、label.y:显著性标签调整
- …:其他参数
比较独立的两组
compare_means(len~supp, data=ToothGrowth)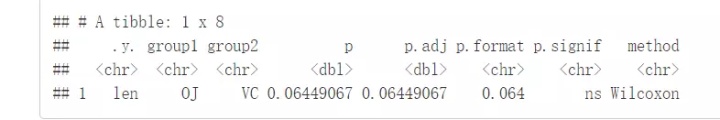
结果解释:
- .y:测试中使用的y变量
- p:p-value
- p.adj:调整后的p-value。默认为p.adjust.method="holm"
- p.format:四舍五入后的p-value
- p.signif:显著性水平
- method:用于统计检验的方法
##绘制箱线图
p <- ggboxplot(ToothGrowth, x="supp", y="len", color = "supp",
palette = "jco", add = "jitter")#添加p-valuep+stat_compare_means()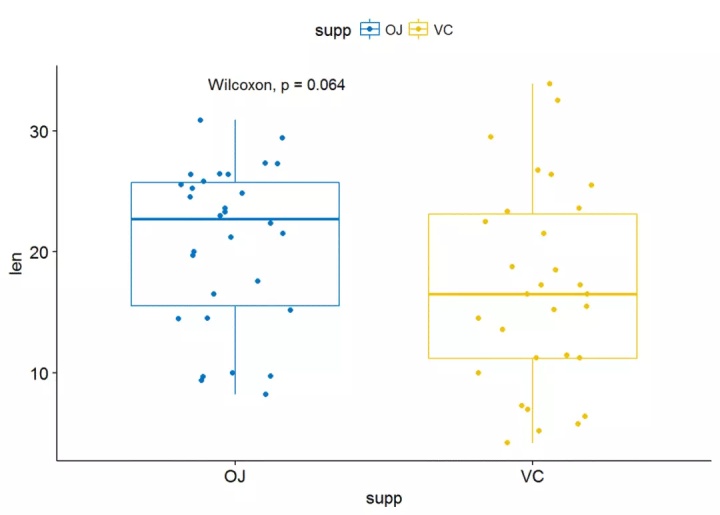
#使用其他统计检验方法
p+stat_compare_means(method = "t.test")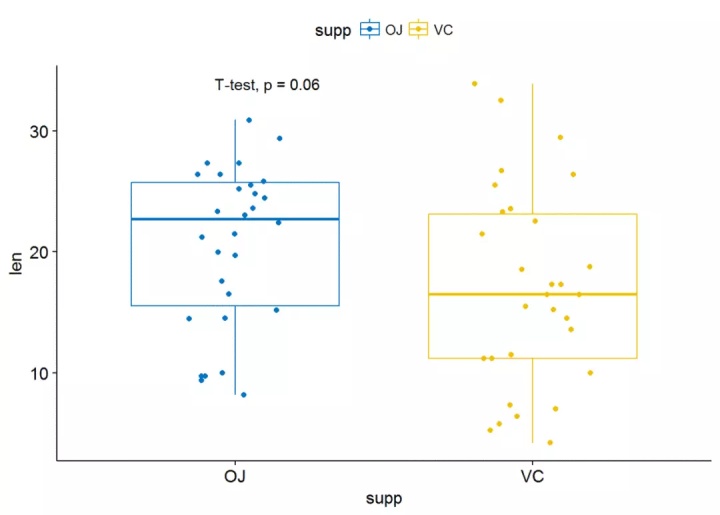
上述显著性标记可以通过label.x、label.y、hjust及vjust来调整
显著性标记可以通过aes()映射来更改:
aes(label=..p.format..)或aes(lebel=paste0("p=",..p.format..)):只显示p-value,不显示统计检验方法aes(label=..p.signif..):仅显示显著性水平aes(labe








 本文详细介绍了如何在R语言中使用ggpubr包为图形添加p值和显著性标记,包括比较独立两组、配对样本、多组比较的方法,并通过实例演示了箱线图、ggpaired()函数的应用,以及如何处理多个分组变量的情况。
本文详细介绍了如何在R语言中使用ggpubr包为图形添加p值和显著性标记,包括比较独立两组、配对样本、多组比较的方法,并通过实例演示了箱线图、ggpaired()函数的应用,以及如何处理多个分组变量的情况。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1788
1788

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








