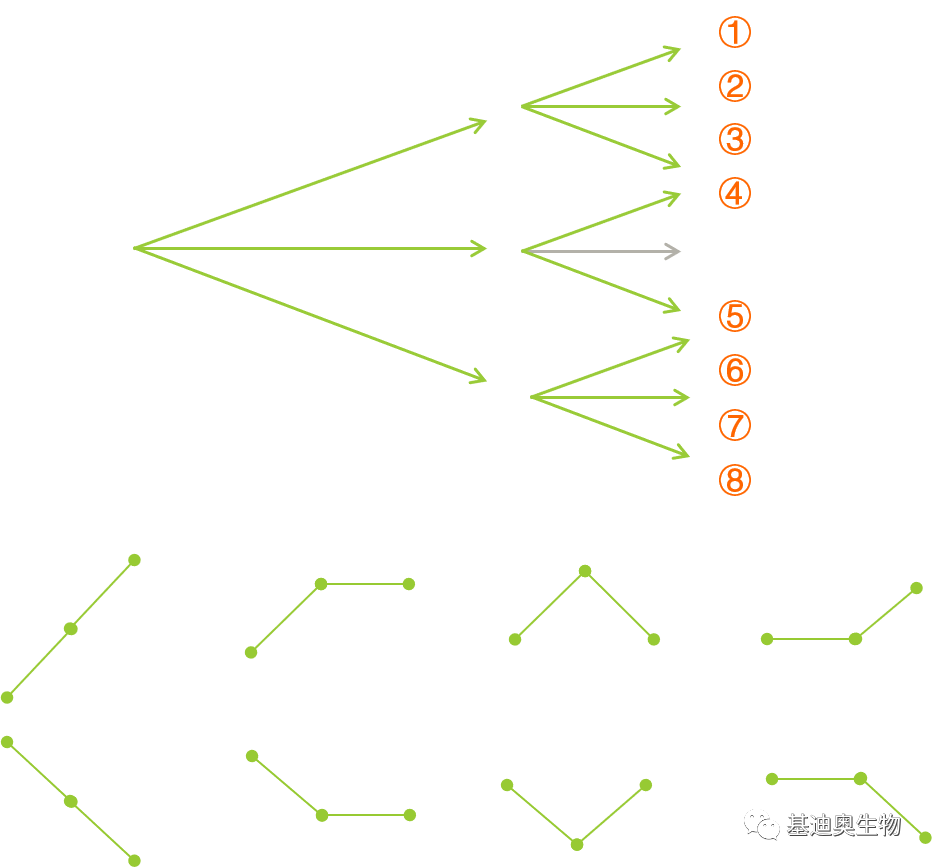
趋势分析(Series Test of Cluster)是基因表达分析研究中的一种重要方法,可用来挖掘具有相同表达模式的基因。简单来说,趋势分析实质就是将表达趋势相同的基因进行归类。例如,假设你的数据来自是3个时间点的梯度处理,当相邻两个时间点的趋势变化的最大单位设为1时,可将基因归类到8种趋势中,如下。
 如果对趋势分析的算法原理感兴趣,可以查看周老师的帖子,这里不再赘述。
链接:
http://www.omicshare.com/forum/thread-446-1-1.html
那么,有没有简单的做趋势分析的工具呢?当然有!OmicShare 目前有两个趋势分析工具作可轻松完成趋势分析。
工具链接:
https://omicshare.com/tools/
如果对趋势分析的算法原理感兴趣,可以查看周老师的帖子,这里不再赘述。
链接:
http://www.omicshare.com/forum/thread-446-1-1.html
那么,有没有简单的做趋势分析的工具呢?当然有!OmicShare 目前有两个趋势分析工具作可轻松完成趋势分析。
工具链接:
https://omicshare.com/tools/
 OS用户可以在
软件中心
的
会员专属
选
项下找到“
动态趋势分析
”工具,如上图(黑色图标),然后进入工具页面。虽然是OmicShare VIP会员专享工具,但
从今日18:00到下周一18:00前,即使你是普通用户也可以使用!
OS用户可以在
软件中心
的
会员专属
选
项下找到“
动态趋势分析
”工具,如上图(黑色图标),然后进入工具页面。虽然是OmicShare VIP会员专享工具,但
从今日18:00到下周一18:00前,即使你是普通用户也可以使用!
 此外,由于趋势分析通常对表达量有变化的基因进行分析(表达没有变化的基因可以认为与处理无关,无需分析),所以我们可直接使用组间差异基因的并集,用于趋势分析。至于如何合并表格,你可能你会需要OmicShare 的表格合并工具。
注意,把Excel中整理好的数据文件另存为
“制表符分隔”的txt文件
,如下, OmicShare和几乎所有的生信工具一样不支持Excel格式。另外,表格中避免出现中文、空格、特殊符号如()、?、*、[ ]、$ 等。
此外,由于趋势分析通常对表达量有变化的基因进行分析(表达没有变化的基因可以认为与处理无关,无需分析),所以我们可直接使用组间差异基因的并集,用于趋势分析。至于如何合并表格,你可能你会需要OmicShare 的表格合并工具。
注意,把Excel中整理好的数据文件另存为
“制表符分隔”的txt文件
,如下, OmicShare和几乎所有的生信工具一样不支持Excel格式。另外,表格中避免出现中文、空格、特殊符号如()、?、*、[ ]、$ 等。
 如果想做练习,可直接使用工具页面提供的示例文件。对于OmicShare所有的工具,大家都可以点击
示例文件
,进入范例数据页面,通过
右键/另存为
,将数据保存为文本文件(默认是制表符分隔的txt文件),然后可上传示例文件用于作图练习。
如果想做练习,可直接使用工具页面提供的示例文件。对于OmicShare所有的工具,大家都可以点击
示例文件
,进入范例数据页面,通过
右键/另存为
,将数据保存为文本文件(默认是制表符分隔的txt文件),然后可上传示例文件用于作图练习。
 需要重点理解的是“
基因筛选的最小变化倍数
”这个参数。在趋势分析前,需要过滤掉那些表达量变化不大的基因,默认过滤掉表达量差异倍数小于2的基因。当然,如果使用差异基因集做趋势分析可不用在意这个参数。
趋势的数量
设置为20也足够了,不建议超出50个。
数据预处理
这里使用默认的“Log2标准化”。
关于
数据预处理
,提供了三种标准化方法:“
Log2标准化
”、“
标准化
”和“
不做标准化/加0
” 。对于这三种方法,原始数据选第1个,已经取过对数的数据选第2个,如果标准化后的数据缺少零时间点的数据,选第3个;数据为0或负数会当成缺失值处理。
需要重点理解的是“
基因筛选的最小变化倍数
”这个参数。在趋势分析前,需要过滤掉那些表达量变化不大的基因,默认过滤掉表达量差异倍数小于2的基因。当然,如果使用差异基因集做趋势分析可不用在意这个参数。
趋势的数量
设置为20也足够了,不建议超出50个。
数据预处理
这里使用默认的“Log2标准化”。
关于
数据预处理
,提供了三种标准化方法:“
Log2标准化
”、“
标准化
”和“
不做标准化/加0
” 。对于这三种方法,原始数据选第1个,已经取过对数的数据选第2个,如果标准化后的数据缺少零时间点的数据,选第3个;数据为0或负数会当成缺失值处理。

 当然,你也可以选择用基因数目对模块进行排序,如下。
当然,你也可以选择用基因数目对模块进行排序,如下。
 接着,在
背景修改
选项下,可以对趋势分析图的配色进行自定义调整,如下。
接着,在
背景修改
选项下,可以对趋势分析图的配色进行自定义调整,如下。
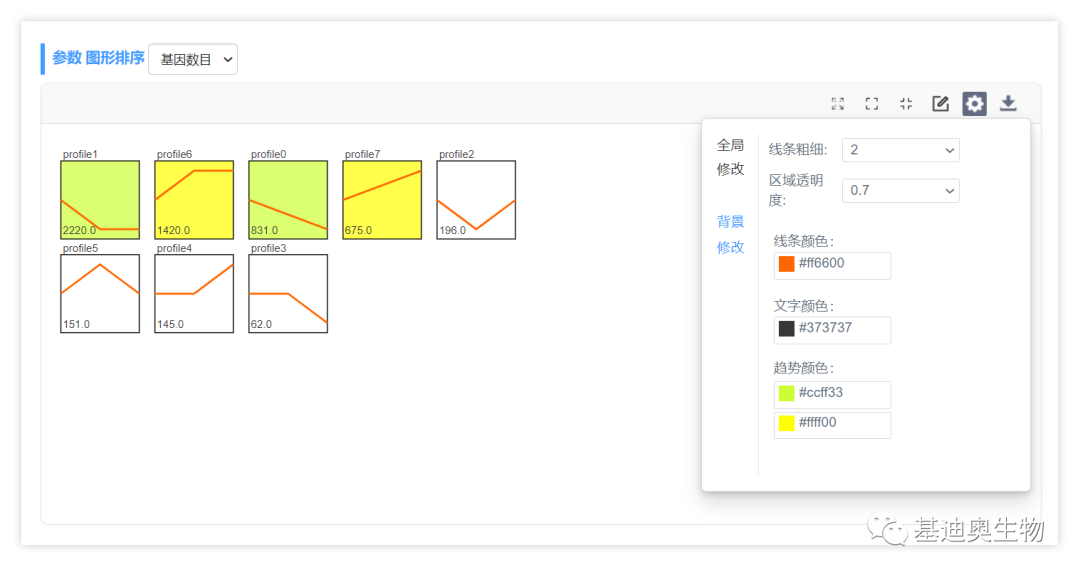 点击编辑趋势按钮,可通过拖拽改变模块位置、单击修改模块名称等。
点击编辑趋势按钮,可通过拖拽改变模块位置、单击修改模块名称等。
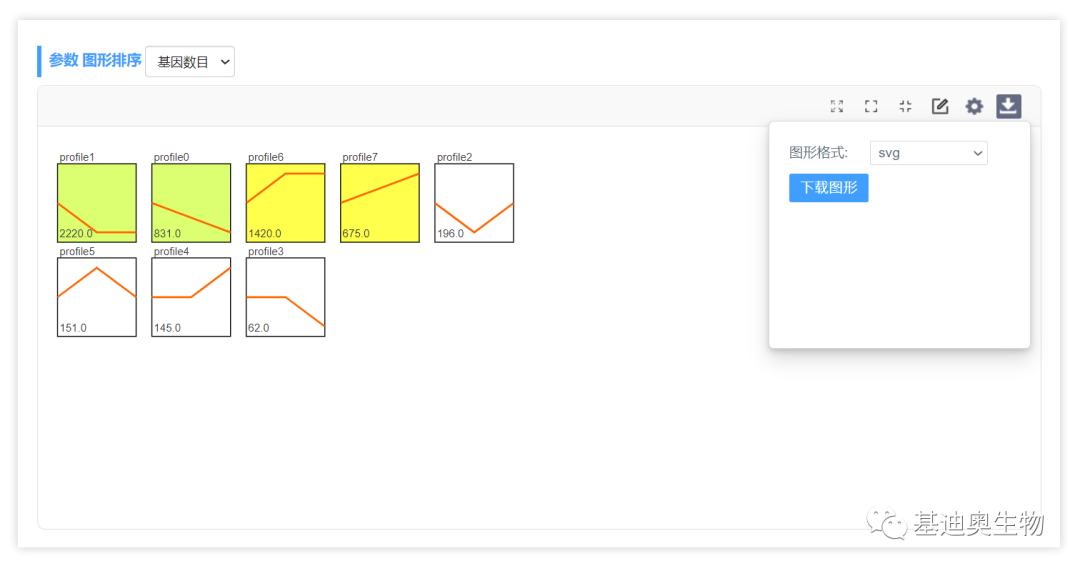
 调整满意后,点下载按钮下载图片即可,OmicShare tools支持下载svg格式的矢量图,便于后期在Ai(Adobe illustrator)中调整或直接在word、PPT中使用。
调整满意后,点下载按钮下载图片即可,OmicShare tools支持下载svg格式的矢量图,便于后期在Ai(Adobe illustrator)中调整或直接在word、PPT中使用。
 此外,在我的任务中可下载静态分析报告,解压后得到下图的结果。
此外,在我的任务中可下载静态分析报告,解压后得到下图的结果。
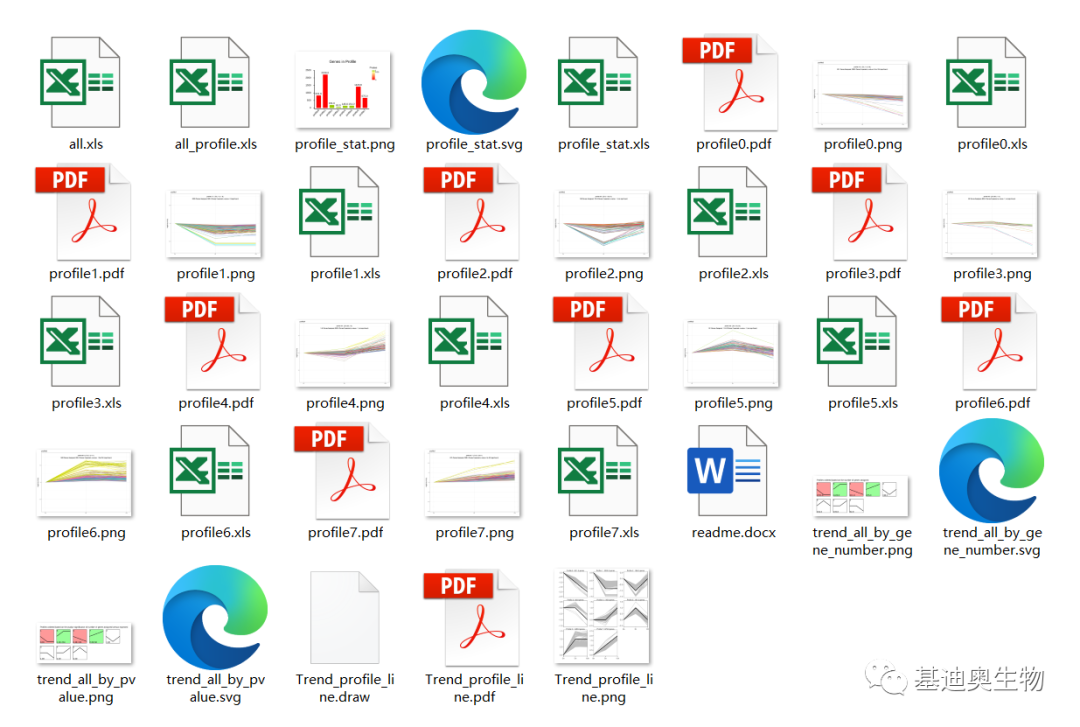 我们所需的数据主要在Trend_analysis这个文件夹中,“
all.xls
”为这次分析用到的gene集,也就是过滤后的基因表达量表;“
All-Profile.xls
”为这些基因对应的“趋势”统计表(可用于个性化作图);然后是每个趋势(profile 0~8)对应的基因集的表达量表(标准化后的)和折线图;以及按基因数和p值排序的所有趋势图。
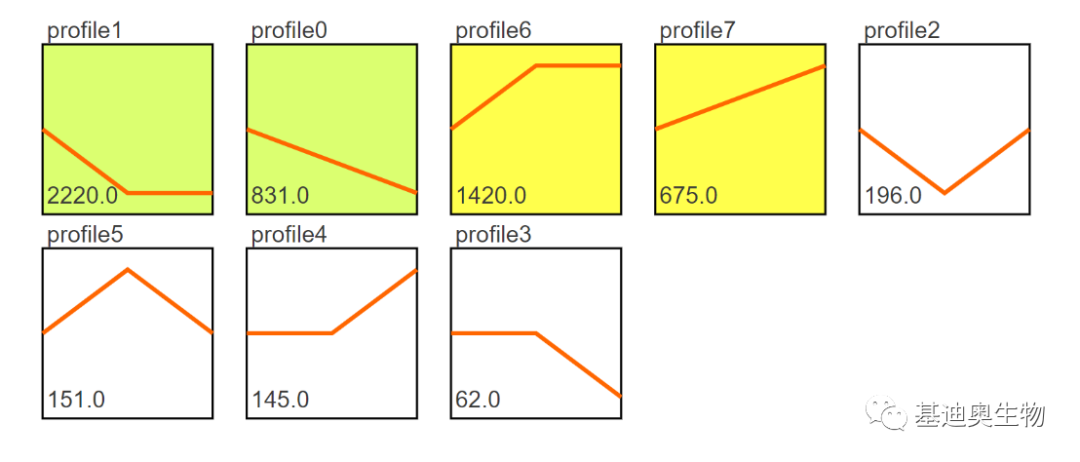
其中,trend_all_by_gene_number(如下图,模块顺序稍作调整)和trend_all_by_pvalue这两种图表比较漂亮,如下,可直接用于文章发表。这两类趋势图在OmicShare用户发表的SCI文章中比较常见。
我们所需的数据主要在Trend_analysis这个文件夹中,“
all.xls
”为这次分析用到的gene集,也就是过滤后的基因表达量表;“
All-Profile.xls
”为这些基因对应的“趋势”统计表(可用于个性化作图);然后是每个趋势(profile 0~8)对应的基因集的表达量表(标准化后的)和折线图;以及按基因数和p值排序的所有趋势图。
其中,trend_all_by_gene_number(如下图,模块顺序稍作调整)和trend_all_by_pvalue这两种图表比较漂亮,如下,可直接用于文章发表。这两类趋势图在OmicShare用户发表的SCI文章中比较常见。
 好啦,趋势分析就完成啦,接下来就可以对感兴趣的模块做进一步的挖掘啦~
好啦,趋势分析就完成啦,接下来就可以对感兴趣的模块做进一步的挖掘啦~


 如果对趋势分析的算法原理感兴趣,可以查看周老师的帖子,这里不再赘述。
链接:
http://www.omicshare.com/forum/thread-446-1-1.html
那么,有没有简单的做趋势分析的工具呢?当然有!OmicShare 目前有两个趋势分析工具作可轻松完成趋势分析。
工具链接:
https://omicshare.com/tools/
如果对趋势分析的算法原理感兴趣,可以查看周老师的帖子,这里不再赘述。
链接:
http://www.omicshare.com/forum/thread-446-1-1.html
那么,有没有简单的做趋势分析的工具呢?当然有!OmicShare 目前有两个趋势分析工具作可轻松完成趋势分析。
工具链接:
https://omicshare.com/tools/
 OS用户可以在
软件中心
的
会员专属
选
项下找到“
动态趋势分析
”工具,如上图(黑色图标),然后进入工具页面。虽然是OmicShare VIP会员专享工具,但
从今日18:00到下周一18:00前,即使你是普通用户也可以使用!
OS用户可以在
软件中心
的
会员专属
选
项下找到“
动态趋势分析
”工具,如上图(黑色图标),然后进入工具页面。虽然是OmicShare VIP会员专享工具,但
从今日18:00到下周一18:00前,即使你是普通用户也可以使用!
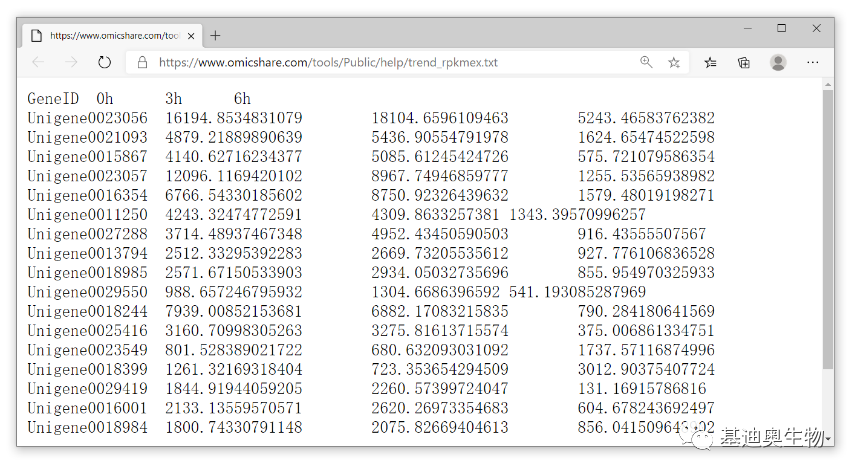
数据准备
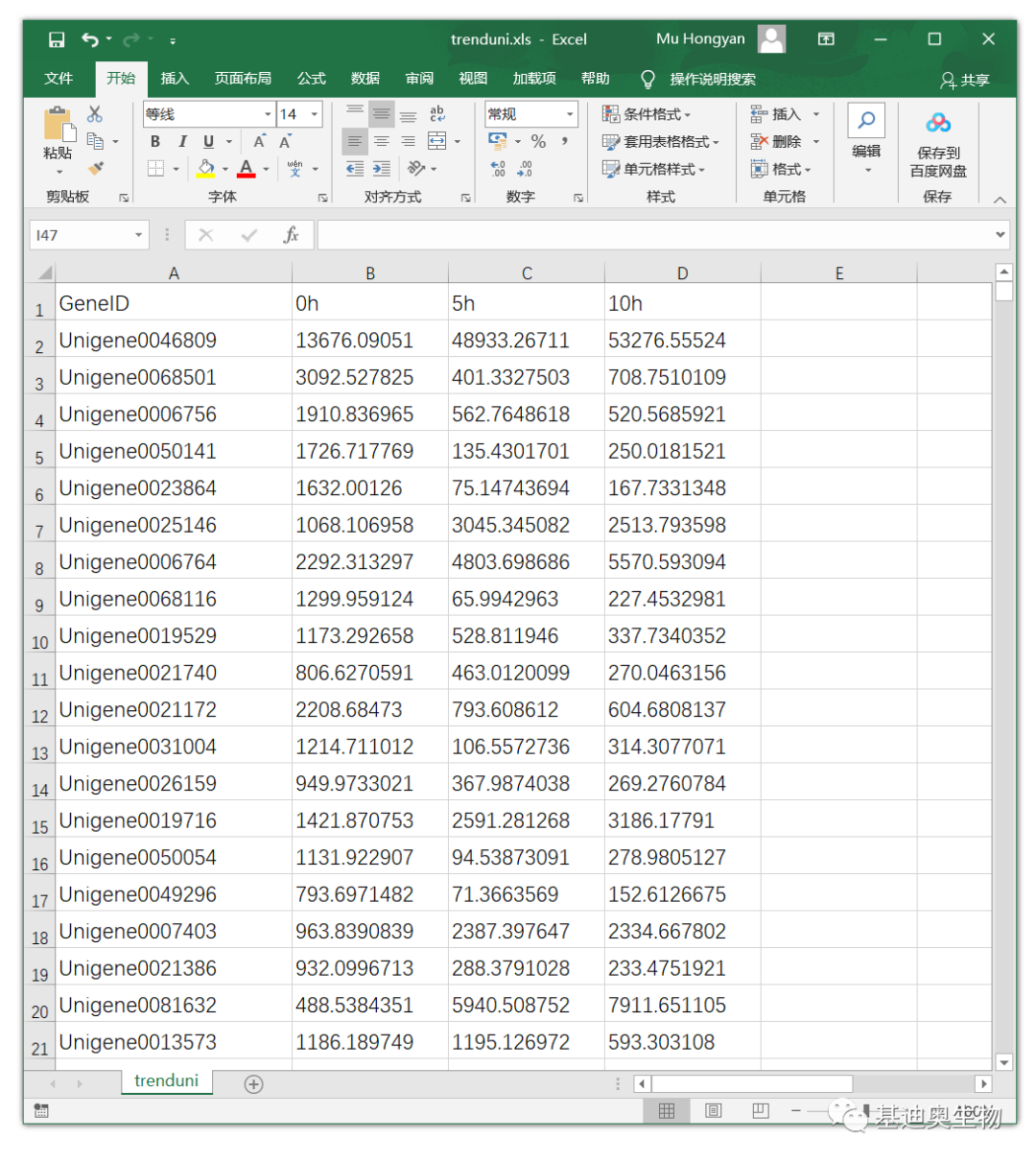
数据的样式如下图,第1列为gene id,之后的列为相应时间点的基因表达量(FPKM)数值。如果每个时间点有3次重复,可以取中值(或均值)用于趋势分析,建议“时间点”分组限制到3~5个。 此外,由于趋势分析通常对表达量有变化的基因进行分析(表达没有变化的基因可以认为与处理无关,无需分析),所以我们可直接使用组间差异基因的并集,用于趋势分析。至于如何合并表格,你可能你会需要OmicShare 的表格合并工具。
注意,把Excel中整理好的数据文件另存为
“制表符分隔”的txt文件
,如下, OmicShare和几乎所有的生信工具一样不支持Excel格式。另外,表格中避免出现中文、空格、特殊符号如()、?、*、[ ]、$ 等。
此外,由于趋势分析通常对表达量有变化的基因进行分析(表达没有变化的基因可以认为与处理无关,无需分析),所以我们可直接使用组间差异基因的并集,用于趋势分析。至于如何合并表格,你可能你会需要OmicShare 的表格合并工具。
注意,把Excel中整理好的数据文件另存为
“制表符分隔”的txt文件
,如下, OmicShare和几乎所有的生信工具一样不支持Excel格式。另外,表格中避免出现中文、空格、特殊符号如()、?、*、[ ]、$ 等。
 如果想做练习,可直接使用工具页面提供的示例文件。对于OmicShare所有的工具,大家都可以点击
示例文件
,进入范例数据页面,通过
右键/另存为
,将数据保存为文本文件(默认是制表符分隔的txt文件),然后可上传示例文件用于作图练习。
如果想做练习,可直接使用工具页面提供的示例文件。对于OmicShare所有的工具,大家都可以点击
示例文件
,进入范例数据页面,通过
右键/另存为
,将数据保存为文本文件(默认是制表符分隔的txt文件),然后可上传示例文件用于作图练习。

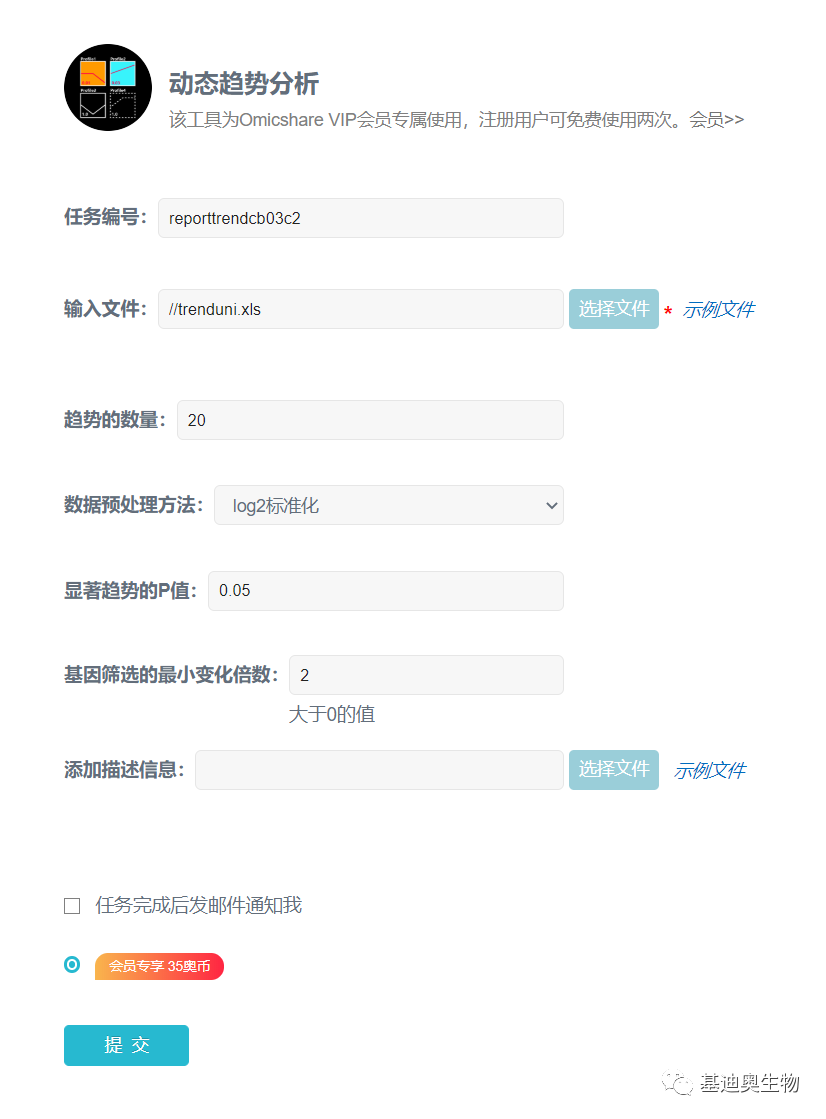
参数设置
参数设置非常简单: 1. 点选择文件按钮上传数据文件; 2. 提交,如下图,其他参数保持默认即可。 需要重点理解的是“
基因筛选的最小变化倍数
”这个参数。在趋势分析前,需要过滤掉那些表达量变化不大的基因,默认过滤掉表达量差异倍数小于2的基因。当然,如果使用差异基因集做趋势分析可不用在意这个参数。
趋势的数量
设置为20也足够了,不建议超出50个。
数据预处理
这里使用默认的“Log2标准化”。
关于
数据预处理
,提供了三种标准化方法:“
Log2标准化
”、“
标准化
”和“
不做标准化/加0
” 。对于这三种方法,原始数据选第1个,已经取过对数的数据选第2个,如果标准化后的数据缺少零时间点的数据,选第3个;数据为0或负数会当成缺失值处理。
需要重点理解的是“
基因筛选的最小变化倍数
”这个参数。在趋势分析前,需要过滤掉那些表达量变化不大的基因,默认过滤掉表达量差异倍数小于2的基因。当然,如果使用差异基因集做趋势分析可不用在意这个参数。
趋势的数量
设置为20也足够了,不建议超出50个。
数据预处理
这里使用默认的“Log2标准化”。
关于
数据预处理
,提供了三种标准化方法:“
Log2标准化
”、“
标准化
”和“
不做标准化/加0
” 。对于这三种方法,原始数据选第1个,已经取过对数的数据选第2个,如果标准化后的数据缺少零时间点的数据,选第3个;数据为0或负数会当成缺失值处理。

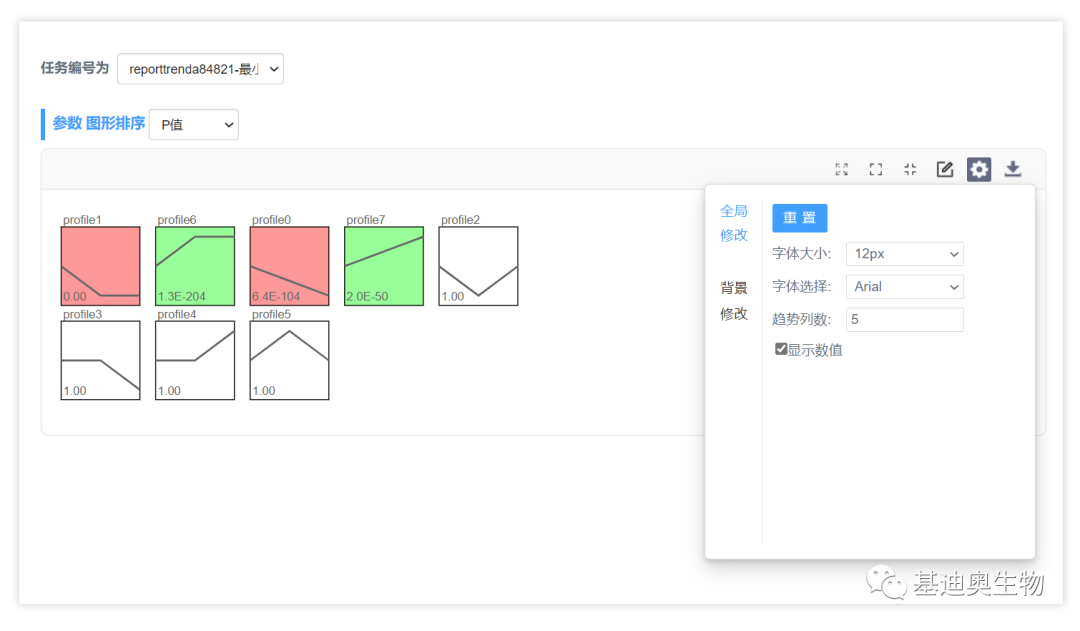
分析结果
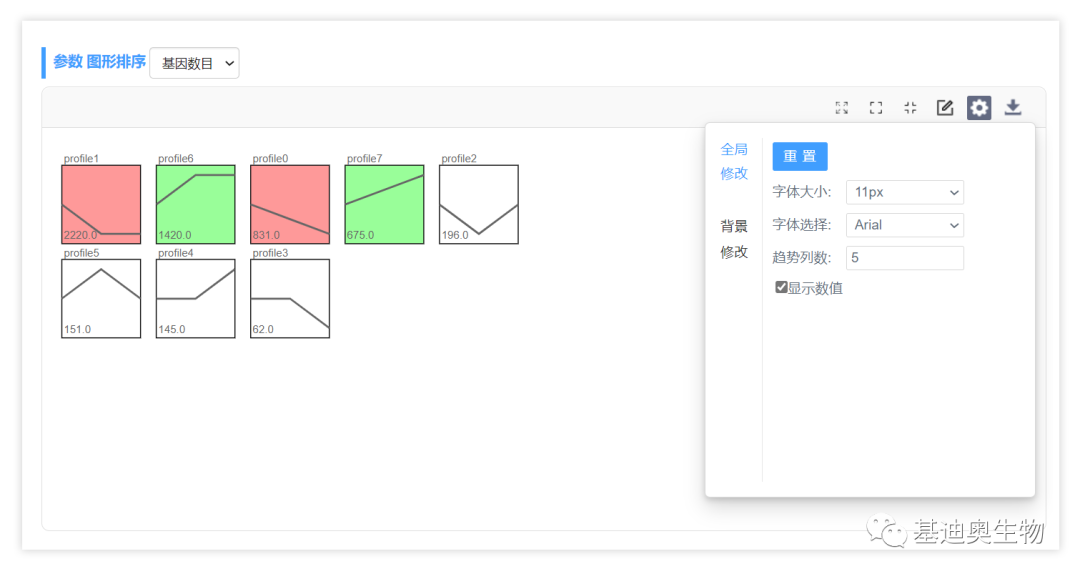
任务编号会随机生成,点击提交按钮,到 我的任务 页面可以查看任务状态,大约十几秒后刷新任务状态即可,然后点跳转查看初始绘制效果,如下图。初始的结果默认是按照p值对模块排序的,彩色的趋势代表显著富集的趋势,颜色相同表示趋势相似(相关系数大于0.7),可进一步合并。也可点设置按钮,在 全局修改 选项下勾选显示数值,设置字体,每行显示的模块数(趋势列数)等。 当然,你也可以选择用基因数目对模块进行排序,如下。
当然,你也可以选择用基因数目对模块进行排序,如下。
 接着,在
背景修改
选项下,可以对趋势分析图的配色进行自定义调整,如下。
接着,在
背景修改
选项下,可以对趋势分析图的配色进行自定义调整,如下。
 点击编辑趋势按钮,可通过拖拽改变模块位置、单击修改模块名称等。
点击编辑趋势按钮,可通过拖拽改变模块位置、单击修改模块名称等。
 调整满意后,点下载按钮下载图片即可,OmicShare tools支持下载svg格式的矢量图,便于后期在Ai(Adobe illustrator)中调整或直接在word、PPT中使用。
调整满意后,点下载按钮下载图片即可,OmicShare tools支持下载svg格式的矢量图,便于后期在Ai(Adobe illustrator)中调整或直接在word、PPT中使用。
 此外,在我的任务中可下载静态分析报告,解压后得到下图的结果。
此外,在我的任务中可下载静态分析报告,解压后得到下图的结果。
 我们所需的数据主要在Trend_analysis这个文件夹中,“
all.xls
”为这次分析用到的gene集,也就是过滤后的基因表达量表;“
All-Profile.xls
”为这些基因对应的“趋势”统计表(可用于个性化作图);然后是每个趋势(profile 0~8)对应的基因集的表达量表(标准化后的)和折线图;以及按基因数和p值排序的所有趋势图。
其中,trend_all_by_gene_number(如下图,模块顺序稍作调整)和trend_all_by_pvalue这两种图表比较漂亮,如下,可直接用于文章发表。这两类趋势图在OmicShare用户发表的SCI文章中比较常见。
我们所需的数据主要在Trend_analysis这个文件夹中,“
all.xls
”为这次分析用到的gene集,也就是过滤后的基因表达量表;“
All-Profile.xls
”为这些基因对应的“趋势”统计表(可用于个性化作图);然后是每个趋势(profile 0~8)对应的基因集的表达量表(标准化后的)和折线图;以及按基因数和p值排序的所有趋势图。
其中,trend_all_by_gene_number(如下图,模块顺序稍作调整)和trend_all_by_pvalue这两种图表比较漂亮,如下,可直接用于文章发表。这两类趋势图在OmicShare用户发表的SCI文章中比较常见。
 好啦,趋势分析就完成啦,接下来就可以对感兴趣的模块做进一步的挖掘啦~
好啦,趋势分析就完成啦,接下来就可以对感兴趣的模块做进一步的挖掘啦~

实用科研工具推荐
详实生信软件教程分享
前沿创新组学文章解读
独家生信视频教程发布








 本文介绍了如何进行PPT中的SWOT分析图制作,包括数据准备和参数设置步骤,并分享了实用的科研工具以及生信软件教程,同时探讨了科研领域的前沿趋势和文章解读。
本文介绍了如何进行PPT中的SWOT分析图制作,包括数据准备和参数设置步骤,并分享了实用的科研工具以及生信软件教程,同时探讨了科研领域的前沿趋势和文章解读。














 3万+
3万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








