高斯分布在自然界非常常见,中心极限定理很好的说明了它,但事情往往不是那么地纯粹,很多时候我们得到的结果里面会混入两个截然不同的样本数据集,虽然它们各自都是高斯分布,但是它们的均值和方差都不一样,如果拿到的是它们的混合数据,就不能简单的使用一个高斯拟合来处理它了。
如果我们有比较强的背景知识,或者看了如下分布的条形图,会下意识的猜想出是两个高斯分布的混合,但是想从数据的角度来探索,两个独立的高斯分布各自独立的均值和方差该如何推测出来呢?
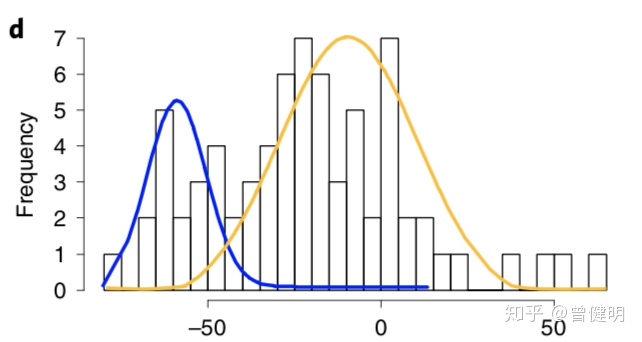
这个难题早在四年前我处理免疫组库数据就遇到了,那个时候功力很浅,全网搜索各自资料也没有解决,后来换工作了,这个项目也就不了了之,最近看文献比较多,其中一个文章的描述了一个R包可以做:
The bimodal distribution was approximated by the ‘normalmixEM’ function in the ‘mixtools’ package of R.
生成测试数据
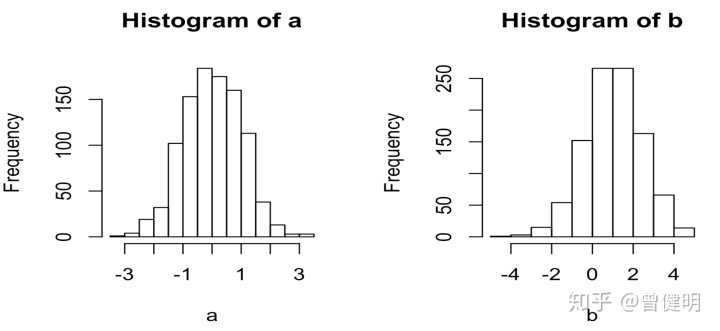
生成两个高斯分布,它们有各自独立的均值和方差
a=rnorm(1000)
b=rnorm(1000,mean = 1,sd=1.4)
par(mfrow=c(1,2))
hist(a);hist(b)可视化如下:
使用mixtools包的normalmixEM函数
首先加载安装好的包:
> library(mixtools)
mixtools package, version 1.1.0, Released 2017-03-10
This package is based upon work supported by the National Science Foundation under Grant No. SES-0518772.先看帮助文档的代码,
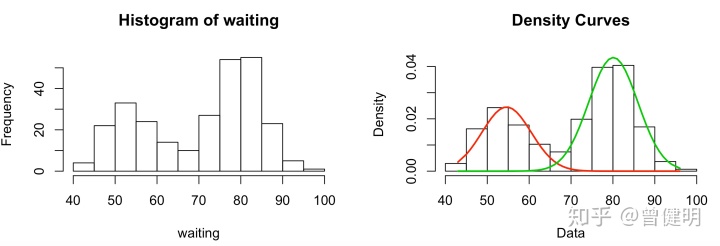
data(faithful)
attach(faithful)
set.seed(100)
system.time(out<-normalmixEM(waiting, arbvar = FALSE, epsilon = 1e-03))
out
hist(waiting)
plot(out,2)可以看到,很简单一个函数,就可以把faithful这个数据框里面的waiting列的数据进行双重高斯分布拟合
在我们的数据上面是使用
前面我们根据R包说明书进行了示例数据分析,那么理所当然就学会了它,然后就应该是应用于自己的数据,我们测试数据是两个高斯分布的向量,如果要使用mixtools包的normalmixEM函数处理它,就应该是需要把两个向量合并.
d=c(a,b)
out<-normalmixEM(d, arbvar = FALSE, epsilon = 1e-03)
hist(d);plot(out,2)可以看到,其实这个函数并不是把我们生成的两个高斯分布完成拆解开来,前面我们模拟的是平均值分别是0和1的两个分布,但是函数拟合后是0和2的两个高斯分布,如下:
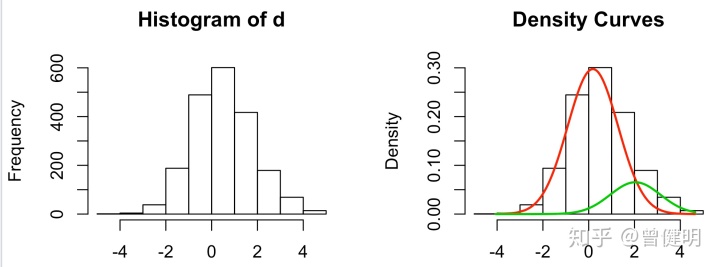
这个时候,有两种解决方案,首先你可以继续花时间去攻克这个R包的详细文档,看看有没有什么参数可以调整,其次你可以去看看其它R包或者算法。
因为这个不是我的课题了,仅仅是想分享给生信技能树的粉丝,符合你们的需求,所以接下来靠你们自己花费时间去摸索哈,比如 http://exploringdatablog.blogspot.com/2011/08/fitting-mixture-distributions-with-r.html

后记
其实这个R包早在2009就发表了,不知道为什么我四年前没有搜索到它,那个时候还没有生信技能树公众号呢
Benaglia, T., Chauveau, D., Hunter, D. R., and Young, D. mixtools: An R package for analyzing finite mixture models. Journal of Statistical Software, 32(6):1-29, 2009.
最后,跟大家互动一下,说到双重高斯分布,大家首先想到的是什么呢?





















 2874
2874











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








