| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
文章目录
前言
分子对接是采用计算模拟的方式,预测受体与配体之间的结合模式,即Pose,以便于后续的Pose评估(打分)。传统对接基于构象搜索,深度学习将分子对接抽象为回归问题,但都没有很好的解决对接准确性问题。
DiffDock来自MIT CSAIL的Regina教授和Tommi教授课题组的工作,他们将分子对接视为一种生成任务,并采用了时下在图像生成等领域相当热门的生成扩散模型(DGM)。
已有的测试显示:DiffDock在已知的对接任务中取得了38%的成功率,对比最先进的深度学习模型(20%)和基于搜索的方法(23%)成功率有较大提升,同时速度也有3-12倍提升。对于被折叠的复合物结构,此前各种方法最大仅达到10.4%的对接成功率,而DiffDock仍达到了21.7%的成功率水平。
本文介绍DiffDock在Linux Ubuntu系统的安装及使用。
一、DiffDock是什么?

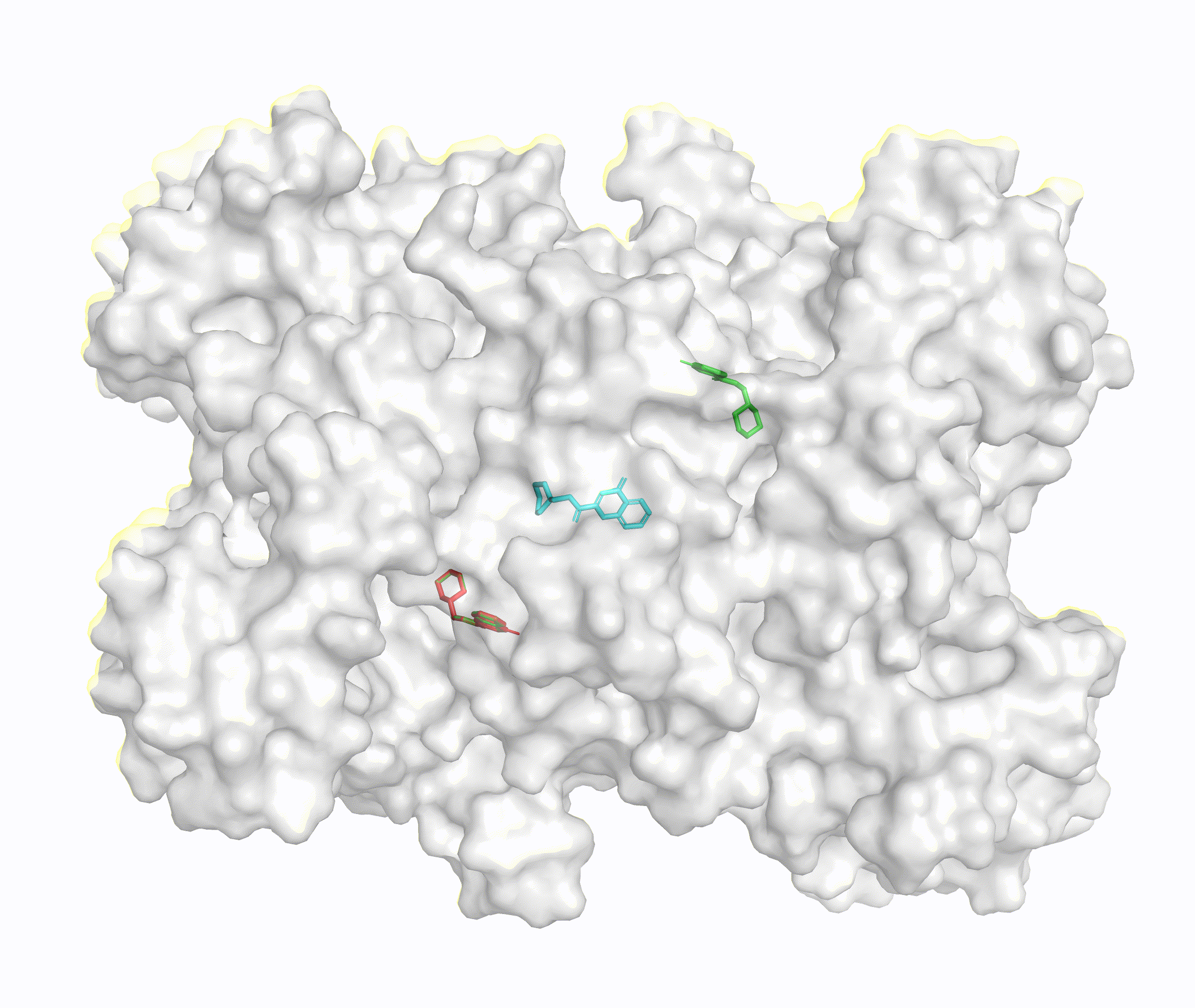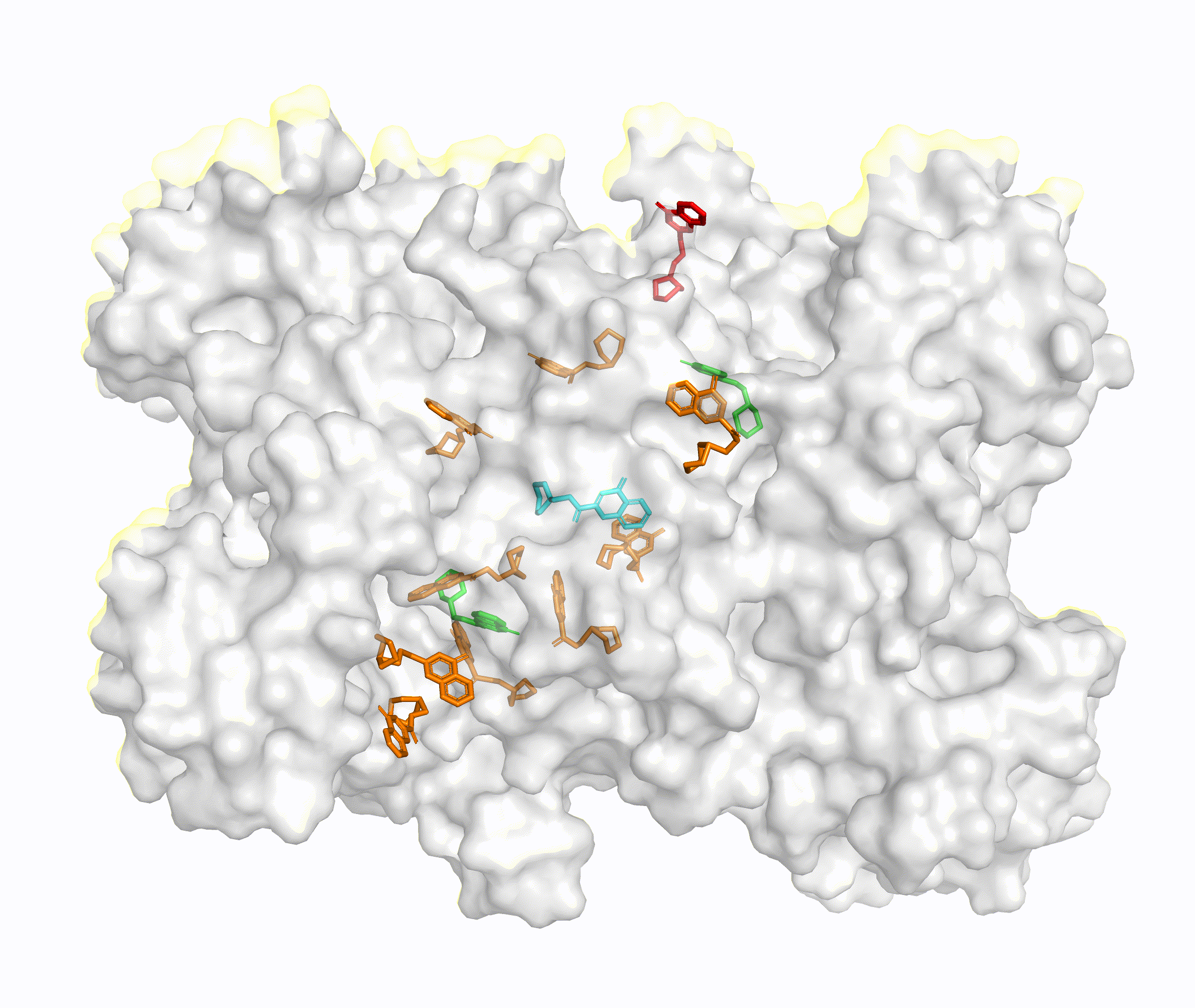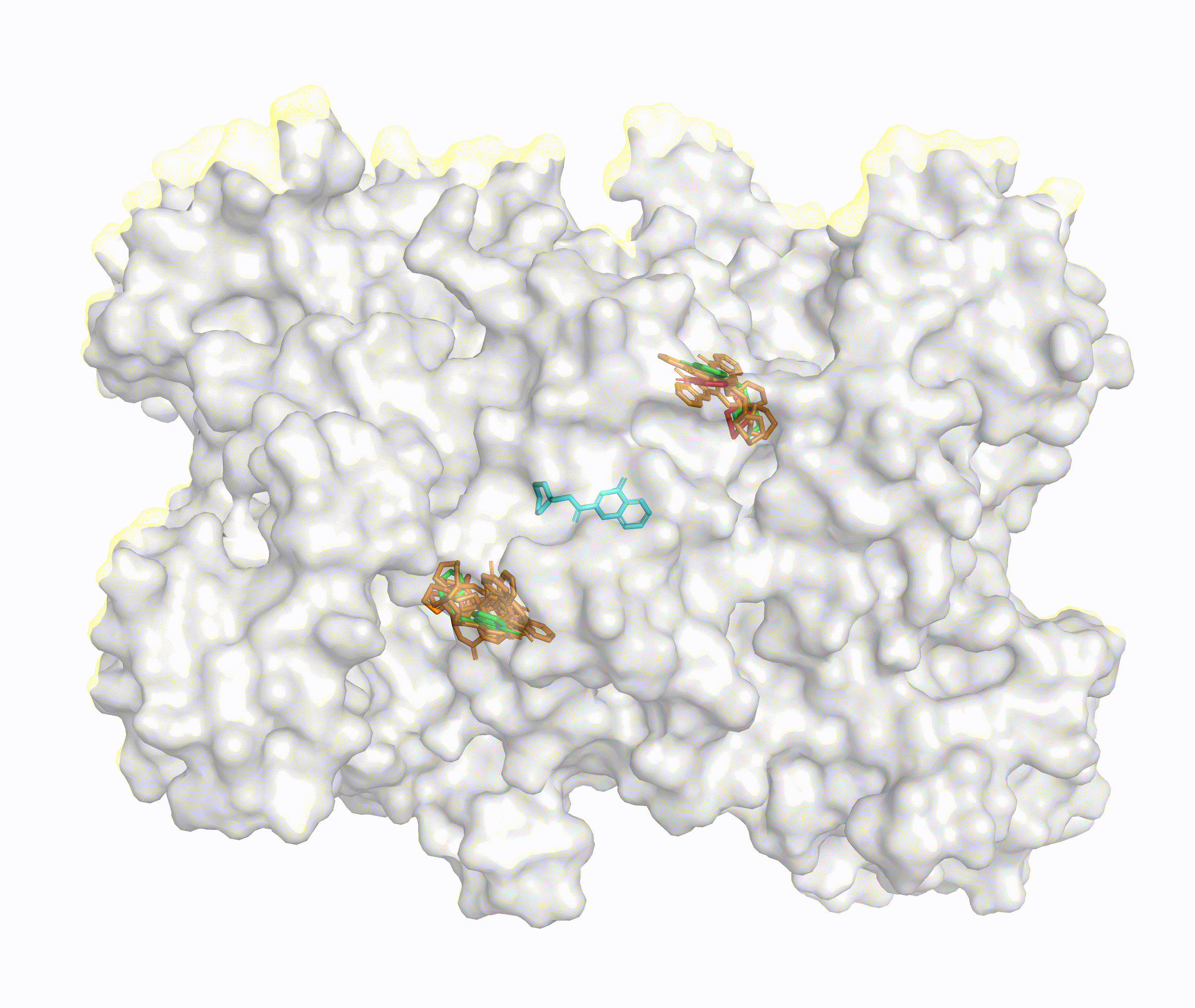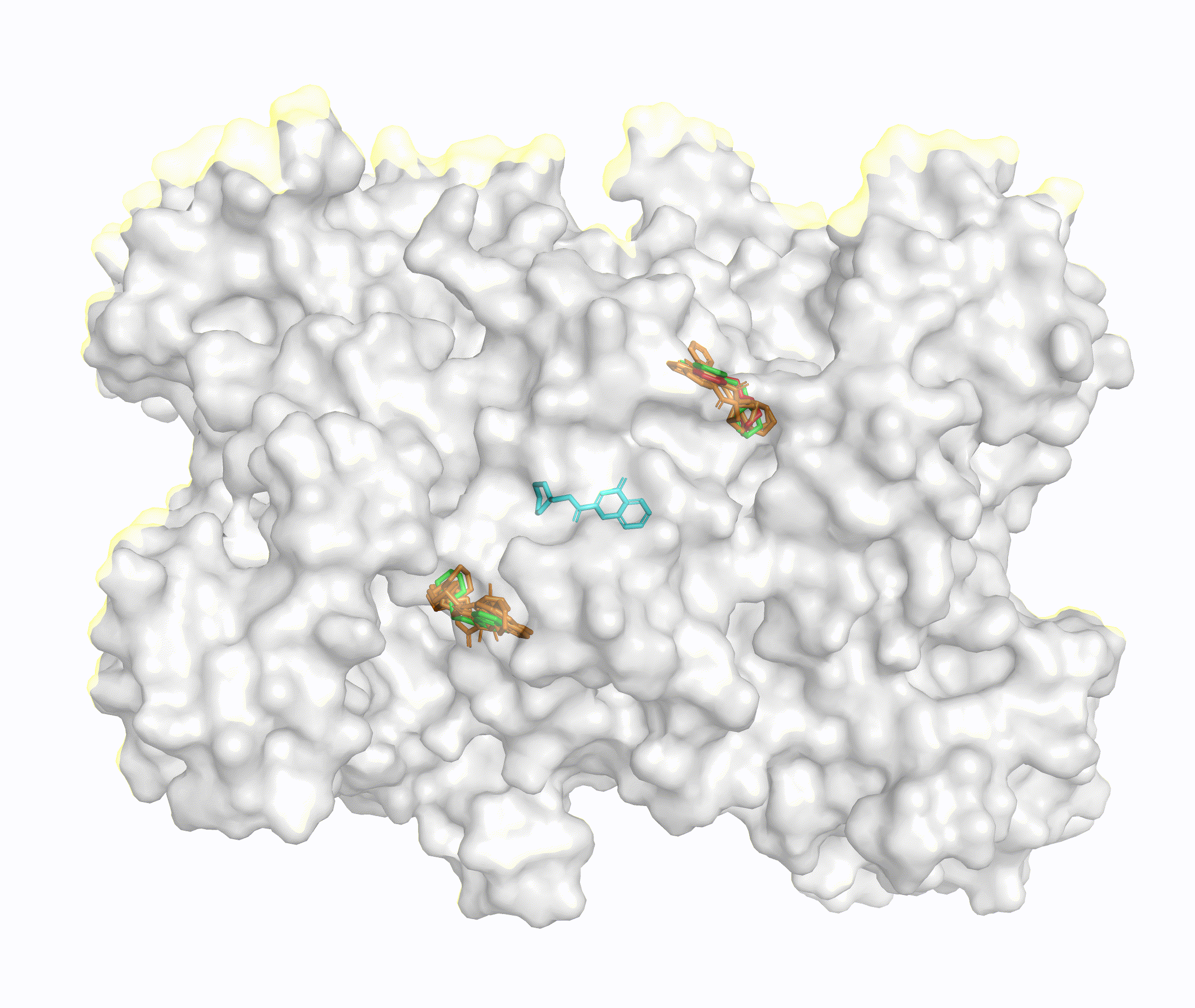
论文来源:DiffDock: Diffusion Steps, Twists, and Turns for Molecular Docking,arxiv.org/abs/2210.01776
代码来源:https://github.com/gcorso/DiffDock
DiffDock的工作流程:将单独的配体和蛋白质结构作为输入,通过平移、旋转和扭转自由度上的反向扩散,对随机采样的初始姿态进行降噪,正确的通过置信度模型对采样的Pose进行排序,以产生最终预测和置信度得分。
DiffDock的主要特点:
- 将分子对接任务定义为一个生成问题,并强调了以前深度学习方法的问题。
- 在配体姿态上建立了一个新的扩散过程,该过程对应于分子对接所涉及的自由度。
- 在PDBBind对接基准上实现了最优的Top1 准确率(RMSD<2A)达到38%,远超此前的最佳搜索算法(23%)和深度学习方法(~20%)。
- 使用ESMFold生成近似的蛋白质apo结构,Top1 准确率(RMSD<2A)达到28%,几乎是最准确基线的准确性的3倍。
与传统对接方法及其他机器学习分子对接方法相比,在PDB共晶结构redock及无监督语言模型预测apo蛋白结构上实现准确度的SOTA:
 Diffdock在PDB共晶结构与ESMFold预测结构上对接,与gnina的比较:
Diffdock在PDB共晶结构与ESMFold预测结构上对接,与gnina的比较:
(RMSD<1.5A时,正确率优于gnina的表现;在ESMFold预测结构上整体优势更加明显。)

在实际场景中(未见受体),DiffDock优势明显,计算效率更高:
 对于apo受体的柔性侧链对接,DiffDock整体最优:
对于apo受体的柔性侧链对接,DiffDock整体最优:

二、DiffDock安装步骤
1. 下载
不建议使用environment.yml直接创建diffdock环境,后续的ESM安装会导致torch_geometric不可用,出现Segmentation fault (core dumped)。可以参考environment.yml安装包的数量和名称。
git clone https://kkgithub.com/gcorso/DiffDock.git
2.创建conda环境并安装
由conda创建虚拟环境并安装必要的包,最后配置ESM及pyg相关包。
STEP 1. 创建conda环境并配置
conda create -n diffdock_env python=3.9
conda activate diffdock_env
conda install pytorch==1.11.0 pytorch-cuda=11.7 -c pytorch -c nvidia
conda install pyarrow joblib
STEP 2. 配置ESM和OpenFold
python -m pip install PyYAML scipy "networkx[default]" biopython rdkit-pypi e3nn spyrmsd pandas biopandas
pip install "fair-esm[esmfold]"
pip install 'dllogger @ git+https://github.com/NVIDIA/dllogger.git'
pip install 'openfold @ git+https://github.com/aqlaboratory/openfold.git'
STEP 3. 检查cuda和pytorch geometric安装
输入以下,查看torch安装版本版本:
python -c "import torch; print(torch.__version__)"
输出:
1.13.1+cu117
输入以下,查看cuda安装版本版本:
python -c "import torch; print(torch.version.cuda)"
输出:
11.7
通过pyg 官网安装pytorch geometric及附件,torch-1.13.1+cu117.html部分需要与上面输出一致:
pip install pyg_lib \
torch_scatter==2.0.9 \
torch_sparse==0.6.15 \
torch_cluster==1.6.0 \
torch_spline_conv==1.2.2 \
torch_geometric==2.0.4
-f https://data.pyg.org/whl/torch-1.13.1+cu117.html
STEP 4. 检查pytorch geometric library
保证torch_geometric能正常工作,检查安装:
python -c "from torch_geometric.loader import DataLoader"
如果没有返回信息,即成功。
三、 DiffDock使用
1. 单一配体+单一受体的复合物对接
简单分子对接,DiffDock支持多种输入方式,用 --protein_path定义受体pdb结构 或者 --protein_sequence定义字符串作为受体序列,用 --ligand 定义配体sdf文件或者smile名称。
运行实例如下:
cd Diffdock
python -m inference \
--protein_path data/1a0q/1a0q_protein_processed.pdb \
--ligand "COc(cc1)ccc1C#N" \
--out_dir results/user_predictions_small_pdb-smi \
--inference_steps 20 \
--samples_per_complex 40 \
--batch_size 10 \
--actual_steps 18 \
--no_final_step_noise
结果保存在–out_dir指定的位置。
python -m inference \
--protein_path data/1a0q/1a0q_protein_processed.pdb \
--ligand data/1a0q/1a0q_ligand.sdf \
--out_dir results/user_predictions_small_pdb-sdf \
--inference_steps 20 \
--samples_per_complex 40 \
--batch_size 10 \
--actual_steps 18 \
--no_final_step_noise
结果保存在–out_dir指定的位置。
2. 多个配体+多个受体对接
需要由--protein_ligand_csv定义一个csv文件,按格式注明配体,受体的名称或者对应文件路径,示例文件位于data/protein_ligand_example_csv.csv,如下:

complex_name:用于保存预测的名称,可以留空
protein_path:.pdb文件的路径,如果为空则使用sequence
ligand_description:SMILE或文件路径
protein_sequence:如果protein_path为空,则使用ESMFold
运行命令如下:
python -m inference \
--protein_ligand_csv data/protein_ligand_example_csv.csv \
--out_dir results/user_predictions_small \
--inference_steps 20 \
--samples_per_complex 40 \
--batch_size 10 \
--actual_steps 18 \
--no_final_step_noise
3. 查看对接Pose
pymol results/user_predictions_small/complex_0/rank1.sdf \
data/1a0q/1a0q_protein_processed.pdb

4. 重新训练模型
对于受体结构已知的分子对接,可以直接使用Diffdock已训练好的模型参数。
如果有需要,可以使用Diffdock提供的工具重新训练模型,需要安装esm获得ESM2 embeddings,然后获得model weights,再训练score model 和confidence model,用于结构推理。
这部分可参阅README文件,此处不再描述。
总结
DiffDock可借助ESMFold实现靶点3D结构未知,从序列开始的分子对接,对接准确度优于已知方法。
本文介绍DiffDock在Linux Ubuntu系统的安装及使用,为分子对接研究提供一种区别于传统对接软件的新方法。
参考资料
- https://github.com/gcorso/DiffDock
- https://blog.csdn.net/weixin_42486623/article/details/130617496
- https://zhuanlan.zhihu.com/p/665191390
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|







 DiffDock是一种利用生成扩散模型改进分子对接的先进技术,它在LinuxUbuntu系统中展示了显著的性能提升。本文详细介绍了DiffDock的原理、安装步骤和使用方法,以及其在结构预测和高准确度对接中的优势。
DiffDock是一种利用生成扩散模型改进分子对接的先进技术,它在LinuxUbuntu系统中展示了显著的性能提升。本文详细介绍了DiffDock的原理、安装步骤和使用方法,以及其在结构预测和高准确度对接中的优势。

















 3586
3586

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










