IDNA-ABF:multi‑scale deep biological language learning model for the interpretable prediction of DNA methylations
期刊:Genome Biology
影响因子:17.906
中科院分区:1区
代码链接:https://github.com/FakeEnd/iDNA_ABF
Abstract
在本研究中,提出了IDNA-ABF,这是一种多尺度深度学习生物语言学习模型,能够仅基于基因组序列对DNA甲基化进行解释性预测。基准比较表明,IDNA-ABF在不同甲基化预测方面优于最先进的方法,重要的是,展示了深度语言学习在从背景基因组中获取顺序和功能语义信息方面的威力。此外,通过可解释的分析机制,我们从很好地解释了模型所学东西,帮助我们建立了从发现重要序列决定因素到深入分析其生物功能的映射。
question
现有的深度学习预测工具尚未充分探索特征表示学习的能力,特别是在发现关键序列模式方面,这些模式对于阐明DNA甲基化机制非常重要。
- 背景基因组序列是否包含可指导DNA甲基化发展的额外可区分信息
- DNA甲基化是否存在,从计算角度来看,跨物种或细胞系的序列模式的保守性和特异性也是一个关键问题
Datasets
Different species datasets
本文选择了IDNA-MS最初提出的相同基准数据集,数据集有三种主要的DNA甲基化类型(4mC、6mA, 5hmC)组成,总共包括17个数据集,此外还收集了三种人类细胞系(K562、GM12878、hepG2)的5mC甲基化数据
Model architecture

Multi‑scale information processing module (多尺度信息处理模块)
本文用k-mer来标记DNA序列,这样每个标记都有k个碱基表示,从而为每个核苷酸整合了更丰富的上下文信息。本文将k设置为3或6。
BERT‑based encoder module(基于BERT的编码器模块)
这里我们使用一个预训练的BERT模型,即DNABERT,使用与BERT基础相同的架构,它由12个Transformer层组成,每个层有768个隐藏单元和12个注意头。值得注意的是,由于DNA序列之间没有直接的语义逻辑,该域预训练调整序列长度,并使模型能够预测适应DNA序列的连续k标记。此外,它使用了与原始BERT相似的屏蔽语言模型技术。
Feature fusion module (特征融合模块)
为了获得两个BERT部分的最终输出,我们通过维度融合门F将两种不同尺度的输出进行组合。

然后通过F生成特定分子的最终矢量表示输出:

Classification module(分类模块)
对抗训练是分类器的一种新的正则化方法,用于提高对小的、近似最坏情况扰动的鲁棒性。
FGM算法
NLP几种常用的对抗训练方法_华师数据学院·王嘉宁的博客-CSDN博客_fgm算法

在FGM方法中,我们将对抗性扰动应用于提取的序列嵌入,而不是直接应用于输入。为了在单词嵌入上定义对抗性扰动,我们将k-mer的相关嵌入表示为s。然后我们将s上的对抗性扰动定义为

为了训练鲁棒模型,我们基于等式(6)中定义的对抗性扰动定义了新的对抗性损失,公式如下:
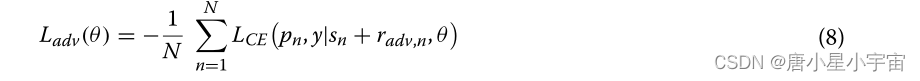
Results
compare with the state‑of‑the‑art methods(独立测试)

如图A和B所示,我们的模型在17个数据集中的15个数据集上优于四个现有模型,只有两个例外——5hmC_M.musulus和6mA_A.thaliana,其中我们的模型实际上也与最佳预测因子相当。具体而言,我们的模型在所有数据集上的平均ACC分别高于两个亚军预测因子iDNA ABT的1.34%和BERT6mA的3.73%。

为了验证模型的稳健性,我们进一步说明了四个数据集上预测因子的ROC和PR曲线。
如图C–F所示。我们可以看到,我们的iDNA ABF在所有四个数据集中具有最高的AUC和AP。具体而言,与其他预测因子相比,我们的模型在四个数据集上的平均AUC和AP值分别增加了约1.39–2.81%和0.1–13.8%。结果进一步证明了我们的模型在DNA甲基化预测任务中的稳健性能。
Adversarial training enhances the predictive performance and the robustness of iDNA‑ABF(对抗训练增强iDNA-ABF的预测性能和鲁棒性)
对抗训练是iDNA ABF的重要组成部分,为了调查对抗训练的有效性,我们将我们的原始iDNA- ABF与未使用对抗训练的模型进行了比较。

图J显示了从数据集中随机选择的两个数据集上进行对抗训练和不进行对抗训练的模型曲线。从图1J中,我们可以看到,尽管损失减少率比没有对抗性训练的模型下降得慢,但与没有对抗性培训的模型相比,有对抗性训练模型的测试损失更低。此外,使用对抗训练,模型在训练过程的后期保持较低的测试损失,而没有对抗训练的模型逐渐开始过度匹配,这表明对抗训练增强了我们模型在DNA甲基化预测中的鲁棒性。

iDNA‑ABF reveals the methylation conservation across species at sequential level
为了研究不同物种的甲基化序列模式是否保守,我们首先使用Lifemap为相同甲基化类型的不同物种构建了进化树。关于4mC甲基化,图2A说明了四个物种的进化关系。

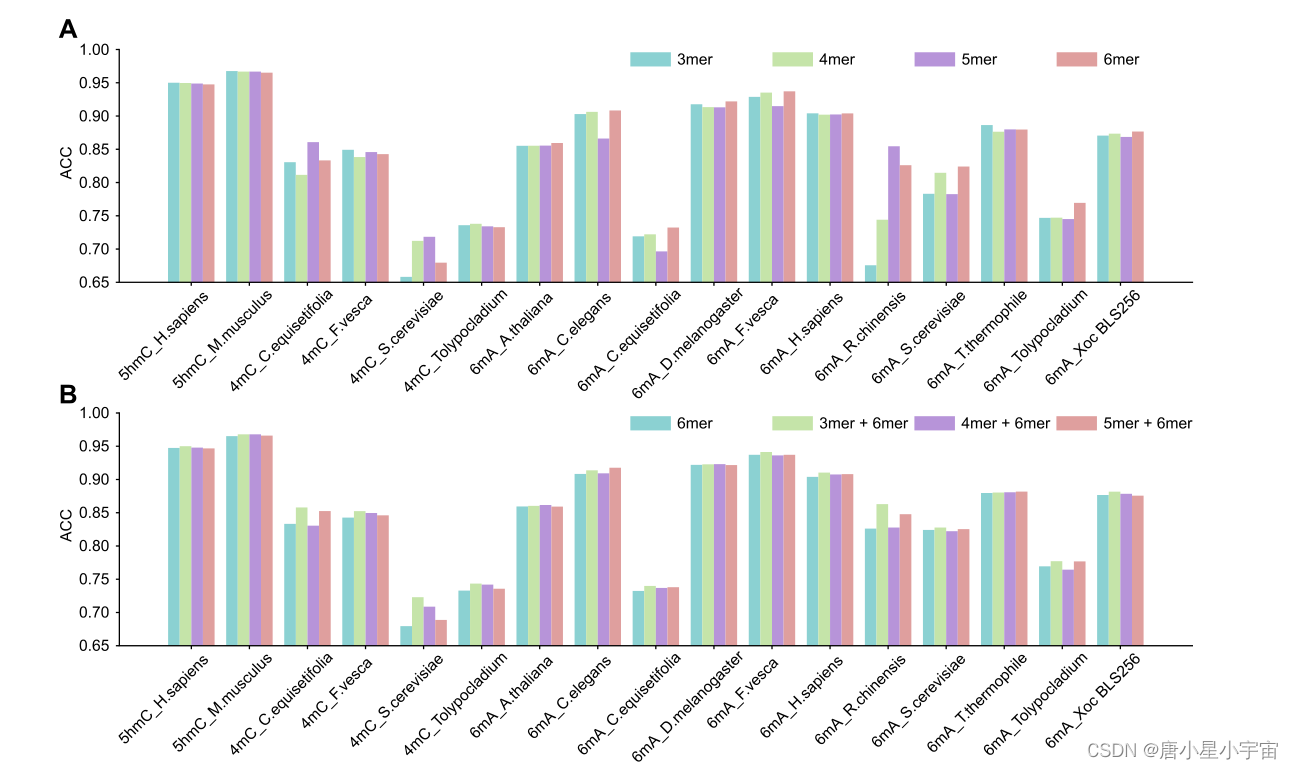
Multi‑scale sequential design choice is more appropriate to elucidate methylation mechanisms
在我们的模型中,我们提出了一种多尺度信息处理策略,通过使用不同的k-mers来表示用于特征表示学习的不同“生物单词”。我们可以看到不同的k-mers确实分别在不同的数据集上具有优势。没有观察到一致的结果。这可能是因为甲基化序列区域的长度因物种和甲基化类型而异。因此,使用单尺度序列模式进行特征表示不能自适应地和充分地捕捉甲基化的固有特征。为了解决这个问题,我们整合了不同尺度的k-mers作为我们的模型输入。
我们进一步研究了为什么使用多尺度k-mer更适合于识别信息捕获。为此,我们利用注意力机制直观地解释我们的模型从两个尺度(3-mer和6-mer)学到的信息。我们分别在图C和D中可视化了两个尺度的注意力热图。

为了清楚地证明哪个序列区域对甲基化预测最重要,我们从三个不同DNA甲基化类型的物种中随机选择了三个序列,并应用注意机制从这些序列中识别关键区域。

iDNA‑ABF sufficiently explores genomic information in 5mC prediction across human cell lines
本节分析了iDNA -ABF在人类细胞系中进行甲基化预测的效果。由于5mC是人类基因组中研究最广泛的甲基化类型之一,我们选择了5mC甲基化来执行我们的方法。我们构建了三个新的5mC数据集,分别对应于三种人类细胞系,包括GM12878、K562和HepG2。
首先讨论了甲基化序列区长度对5mC甲基化预测的影响。因此,对于每个细胞系,我们构建了四个5mC数据集,其中每个5mC序列分别长11、41、71和101 bp(碱基对)。



随着序列长度的增加,模型性能显著提高,这表明更长的序列会给模型带来额外的基因组上下文信息。当长度为71bp时达到峰值。之后,模型性能逐渐下降。
一个有趣的问题是,将NGS数据与序列数据相结合是否有助于更准确的预测。为此,我们选择了两个组蛋白修饰(HM)数据,H3k4me3和H3k36me3

与使用ChIP seq数据训练的模型相比,使用序列数据训练的模型获得了显著更好的性能,当将ChIP seq数据与序列数据相结合用于模型训练时,所有性能指标都得到了进一步改进,这表明ChIP seq数据和序列数据在改进的5mC预测中是相互补充的。
iDNA‑ABF has robust performance in 5mC prediction on unseen human cell lines
为了分析iDNA -ABF在未发现细胞系中的预测性能,我们进行了跨细胞系验证。具体来说,我们在一个细胞系上训练模型,并在另一个细胞上进行评估。

为了更好的解释,我们引入了三种人类细胞系甲基化中心区的概率分布分析。图5B和C分别显示了三种细胞系中阳性和阴性样品的概率分布。

iDNA‑ABF has good transfer learning ability to capture the specificity of methylated sequential patterns
5mC甲基化主要发生在人类基因组中具有CpG模式的序列中;实际上,在少数情况下,在CHH和CHG模式中也检测到甲基化(其中H=A、C或T)。为了找出不同的甲基化序列模式是否相互关联,我们分别为三种细胞系构建了额外的CHG和CHH数据集。为了观察我们的模型在检测不同甲基化模式方面是否具有良好的迁移学习能力,我们首先在CpG数据集上预训练了一个模型,并在CHG或CHH数据集上对其进行了微调,产生了另一个称为“迁移学习模型”的模型,然后用CHG或CHH数据集的相同测试数据集对两个模型进行评估。


结果表明,我们的模型具有良好的迁移学习能力;预训练机制可以从一个特定模式中带来额外的鉴别信息,从而有利于目标模式的预测,从而提高预测性能。
为了深入解释使用迁移学习提高性能的可能原因,我们进一步分析和比较了从三个模型中学习的基序,包括基线模型、迁移学习模型和仅在CpG数据集上训练的预训练模型。HepG2细胞系中的基序比较结果如图所示。

我们观察到迁移学习模型不仅保留了从预训练模型继承的一些CpG模式,而且捕获了基线模型学习的CHG模式的特异性。
Discussions
iDNA-ABF,这是一种仅基于基因组序列通过生物语言学习鉴定DNA甲基化的新方法。我们的iDNA-ABF不仅能够实现跨物种和跨细胞系的相对准确的甲基化预测,而且还利用可解释的注意机制建立了从序列水平到功能水平的映射,以研究深入的DNA甲基化机制。
- 我们研究了模型的预测性能, 在17个基准数据集中的实验结果表明,与现有的基于序列的方法相比,我们的模型表现出一贯优越和稳健的性能,这些数据集涵盖了多个物种中的三种甲基化类型(4mC、5hmC和6mA)。
- 消融分析揭示了对抗训练在模型性能中的重要性。特别是,我们训练过程中的对抗训练减轻了大规模参数的影响,特别是对一些小数据集的影响,并提高了我们模型在不同物种和甲基化类型之间的泛化能力。
- 此外,我们还研究了序列长度对甲基化预测的影响。结果表明,模型性能通常随着输入序列长度的增加而提高,表明甲基化序列区域周围的上游和下游对识别DNA甲基化位点至关重要。它们可能包含从序列角度来看的某种程度的特异性信息,以帮助我们的模型区分甲基化位点和非甲基化位点;
- 另一方面,我们采用DNABERT相同的模型架构进行模型构建,这是一种经过预训练的强大的自然语言学习模型,拥有百万级基因组序列数据。它使我们的模型能够从背景基因组中捕获更多顺序语义。
- 特征空间可视化分析结果证明,与现有的预测因子相比,我们的模型学习了更多可区分的特征表示。有趣的是,通过将组蛋白修饰数据(例如,H3K4me3和H3K36me3)等ChIP-seq数据整合到我们的模型中,我们观察到性能进一步提高,表明生物信号和序列数据对于改进的预测是互补的。
为了提高模型的可解释性,我们对模型构建进行了两项主要改进。一种是提出用于模型训练的多尺度序列处理策略,另一种是引入用于模型分析的注意力机制。我们从不同尺度学习的基序与传统基序查找工具STREME的基序高度一致,表明我们的模型能够发现保守的序列模式。
为了回答模型所学习的序列模式是否具有生物学意义或与甲基化相关这个问题,将模型应用于人类细胞系中5mC甲基化的预测。我们研究了模型的迁移学习能力,实验结果表明,从CpG甲基化模式中学习知识可以帮助改进对其他两种甲基化模式(即CHH和CHG)的预测。这表明我们的模型在学习不同甲基化模式的特异性方面有很强的能力。结果也暗示了模型在发现其他罕见甲基化模式方面的潜力。
Conclusion
本文提出的深度生物语言学习模型在DNA甲基化预测中取得了令人满意的性能。重要的是,我们展示了深度语言学习在从背景基因组中捕获顺序和功能语义信息方面的能力。此外,通过整合可解释的分析机制,我们很好地解释了我们所学到的东西,帮助我们构建了从发现重要序列决定因素到深入分析其生物学功能的映射。然而,仍有很大的改进空间。例如,在构建甲基化预测模型时,我们只考虑了甲基化位点周围的局部序列区域,其中的鉴别信息可能在一定程度上受到限制。研究表明,基因组中的基因调控具有长期的相互影响,如增强子-启动子相互作用。因此,探索长序列整合信息如何影响DNA甲基化水平可能是未来工作的一个重要方向。







 提出IDNA-ABF模型,一种多尺度深度生物语言学习模型,用于基因组序列的DNA甲基化预测。该模型在多种甲基化类型及跨物种预测中表现优异,并通过可解释性分析揭示甲基化机制。
提出IDNA-ABF模型,一种多尺度深度生物语言学习模型,用于基因组序列的DNA甲基化预测。该模型在多种甲基化类型及跨物种预测中表现优异,并通过可解释性分析揭示甲基化机制。
















 4633
4633

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








