由于本人专业纯理科,真的对计算机代码很小白,有幸接触到研究方向计算生物,这一周在尝试输出蛋白质序列的嵌入表示代码(ESM2)时出现了很多问题,因此来此地记录自己的学习过程。
ESM:https://github.com/facebookresearch/esm/tree/main?tab=readme-ov-file
一.关于环境
建立环境并激活
在VScode终端输入:conda activate esmfold #esmfold可以是任意名字
第一条命令创建了一个名为 esmfold 的新环境,并安装 Python 3.8。
conda create --name new_esm python=3.8
conda activate new_esm
在创建环境中可能会看到 Proceed ([y]/n)? 这样的提示表示 Conda 正在询问你是否确认继续操作。输入 y 并按回车键,以继续创建环境或安装软件包。

二.关于pytorch
要确定 PyTorch 和 CUDA 版本兼容性,可以参考 PyTorch 官方网站提供的兼容性表。
访问 PyTorch 官方网站的 Get Started 页面:
Previous PyTorch Versions | PyTorch
在安装之前,需要确保系统已经安装了对应版本的 CUDA。可以使用以下命令来检查当前安装的 CUDA 版本:
nvcc --version


比如我的 CUDA 版本 11.8,使用以下命令安装与 CUDA 11.8 兼容的 PyTorch 版本:
conda install pytorch torchvision torchaudio pytorch-cuda=11.8 -c pytorch -c nvidia
直接用上面的代码下,很慢,一直卡在下图所示的位置,看了大佬的博客后才知道需要用镜像(安装指令都是从国外的通道下载,下载速度会非常慢。)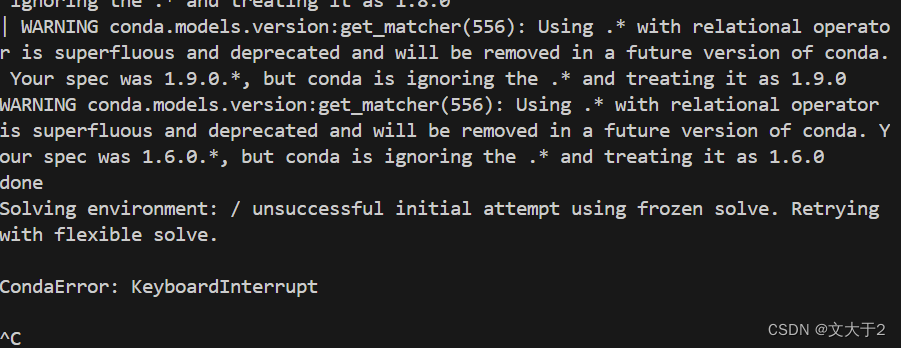
大佬博客:2023最新pytorch安装(超详细版)-CSDN博客
加上清华镜像
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main
conda config --get根据大佬博客推荐,使用pip安装

有惊无险地装完(耗时大概一个半小时?午觉睡醒就装好了),最后提示有ERROR

把缺失的包都装完
pip install bitarray cffi cython "omegaconf<2.1" regex sacrebleu scikit-learn tqdm
继续有ERROR
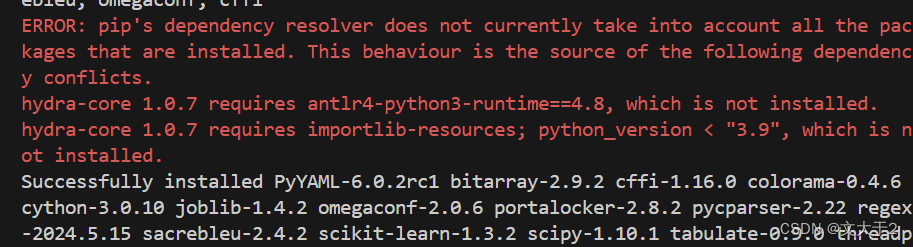
pip install antlr4-python3-runtime==4.7.2 importlib-resources==5.2.2(emmmmm, 为嘛还有错误,但是本人表示不想管了,直接再试试装fairseq)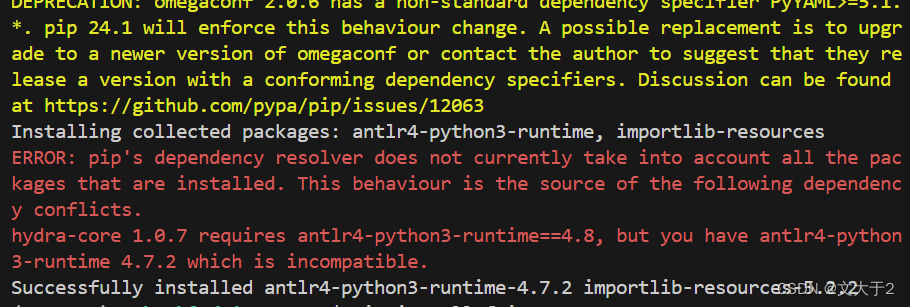
pip install fairseq成功。(没有红色的ERROR了 哈哈哈哈哈哈哈哈哈)

2.2使用torch
事情果然没有这么简单......import torch时,又来了新问题。
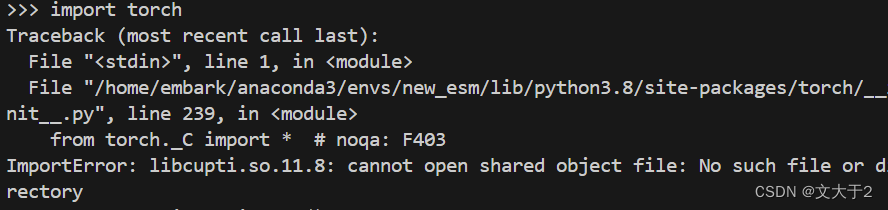
输入exit(),退出python
虽然 torch 已经安装成功,但是仍然会出现 libcupti.so.11.8 相关的错误,说明 libcupti.so.11.8 这个共享库文件可能没有正确配置或没有被系统找到。
检查 CUDA 安装路径
假设你的 CUDA 安装在 /usr/local/cuda,检查 libcupti.so 是否存在:
ls /usr/local/cuda/extras/CUPTI/lib64/libcupti.so*
出现的结果表明libcupti.so 文件确实存在于 /usr/local/cuda/extras/CUPTI/lib64/ 目录中。这表示库文件是存在的,但可能没有正确配置到系统的库路径中。
设置环境变量以包含 libcupti 的路径
确保系统可以找到 libcupti.so 文件,我们需要将其路径添加到 LD_LIBRARY_PATH 环境变量中。
在终端中运行以下命令来设置环境变量:
export CUPTI_PATH=/usr/local/cuda/extras/CUPTI/lib64
export LD_LIBRARY_PATH=$CUPTI_PATH:$LD_LIBRARY_PATH为了使这些设置在每次打开终端时生效,可以将这些命令添加到 ~/.bashrc 或 ~/.zshrc 文件中:
echo 'export CUPTI_PATH=/usr/local/cuda/extras/CUPTI/lib64' >> ~/.bashrc
echo 'export LD_LIBRARY_PATH=$CUPTI_PATH:$LD_LIBRARY_PATH' >> ~/.bashrc
source ~/.bashrc验证环境变量设置
echo $LD_LIBRARY_PATH看到包含 /usr/local/cuda/extras/CUPTI/lib64 的路径。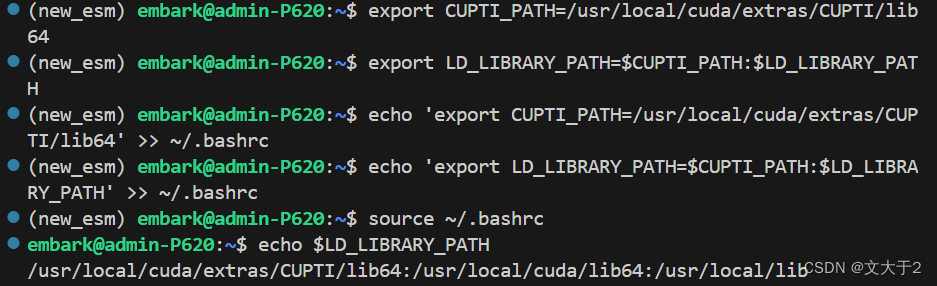
再重新import torch,不会再出现错误啦~
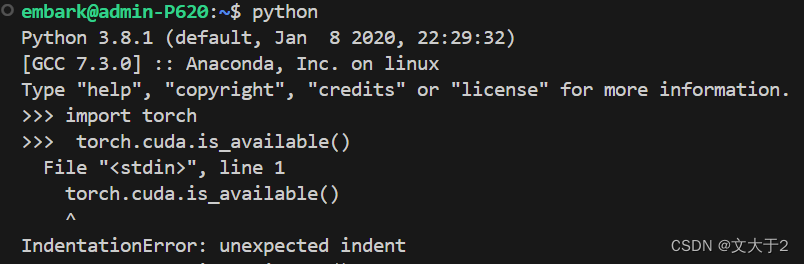
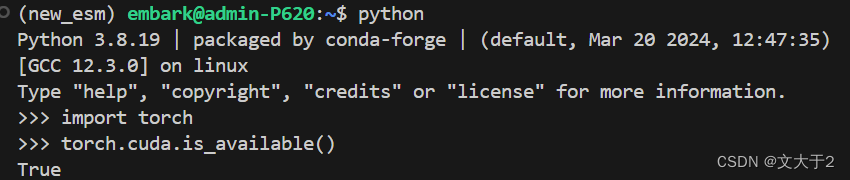
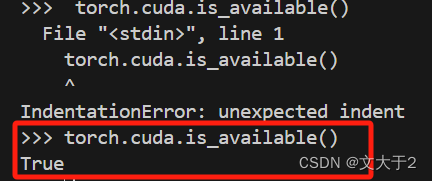
上面的代码:torch.cuda.is_available()之所以有问题是因为前面多打了一个空格
正常输入就可以啦~
三.正式跑代码(ESM)
3.1怎么老卡在import torch?
果不其然,又来了问题,在 Jupyter Notebook 中运行 PyTorch 时,仍然找不到 libcupti.so.11.8 文件。尽管在终端中运行 Python 没有问题,但在 Jupyter Notebook 中可能没有正确继承环境变量。

问了chatgpt,给出的解决方案如下:
第一步:
import os
os.environ['CUPTI_PATH'] = '/usr/local/cuda/extras/CUPTI/lib64'
os.environ['CUDA_HOME'] = '/usr/local/cuda'
os.environ['LD_LIBRARY_PATH'] = f"{os.environ['CUDA_HOME']}/lib64:{os.environ['CUDA_HOME']}/extras/CUPTI/lib64:{os.environ.get('LD_LIBRARY_PATH', '')}"
print(os.environ['LD_LIBRARY_PATH'])第二步:
import ctypes
import ctypes.util
print(ctypes.util.find_library('cupti'))
print(ctypes.util.find_library('cuda'))
print(ctypes.util.find_library('cudart'))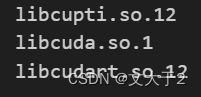
ctypes.util.find_library 找到了 libcupti.so.12 和 libcudart.so.12。表明系统上安装了 CUDA 12 的库,而PyTorch 版本是针对 CUDA 11.8 构建的。这种不匹配可能导致了一些问题。
第三步:
import ctypes
ctypes.CDLL('/usr/local/cuda/extras/CUPTI/lib64/libcupti.so.11.8', ctypes.RTLD_GLOBAL)
import torch
print(torch.__version__)
print(torch.cuda.is_available())(继续运行代码也可以了~,注意是/usr/local/cuda/extras/CUPTI/lib64/libcupti.so.11.8,不是libcupti.so.12)
解决!

明天继续............






















 482
482











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








