
题目: Protein-Ligand Interaction Graphs: Learning from Ligand-Shaped 3D Interaction Graphs to Improve Binding Affinity Prediction
文献来源: https://doi.org/10.1101/2022.03.04.483012 (bioRxiv)
代码:github.com/MarcMoesser/ProteinLigand-Interaction-Graphs
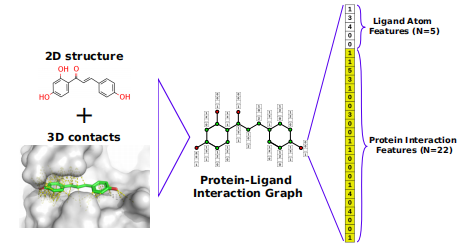
简介:图神经网络(GNNs)最近开始流行起来,挑战了分子指纹或基于SMILES的表征,逐渐成为表征分子结合亲和力预测的主要方法。虽然简单的基于配体的图已经对亲和预测有用,但包含三维结构信息的模型在多目标数据集上实现了更好的性能。最近的研究利用复杂的GNN结构,通过将配体相互作用的蛋白质原子作为图中的附加节点来捕获三维蛋白质配体信息;或者并行构建第二个基于蛋白质的图。这大大扩展了该图,同时模糊了潜在配体的形状,削弱了GNN在编码分子结构时的优势。因此,需要一个简单而优雅的分子图表示,它保留配体的拓扑结构,同时编码三维蛋白质与配体的相互作用。
本文提出了蛋白质-配体相互作用图(PLIGs):一种简单的方法来表示三维蛋白质-配体复合物的原子-原子接触作为GNN的节点特征。PLIGs通过描述每个原子的性质以及在一个距离阈值内与蛋白质原子进行的所有原子-原子接触,使分子图中的一个原子节点达到饱和。因此,该图的边缘与基于配体的图是相同的,但节点编码的三维蛋白质-配体接触。PLIG-based图注意网络(GATNet)获得了最好的性能(ρ=0.84,RMSE=1.22 pK)。总之,作者创建了一种新的基于图的表示方法,它将三维结构信息整合到配体状分子图的节点特征中。PLIG的表示方法简单、优雅、灵活、易于定制,这为将其他2D和3D属性合并到图中提供了许多可能性。
主要内容:
-------------------------------------------
欢迎点赞收藏转发!
下次见!























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








