任务:
基于下面的模板序列生成符合要求的大量随机序列:CAGGTCTCAAGTGGNNNNNGANNNNNNNNNGAAANNNNNNNNNAAGTNNNNNTAAGGCTAGTCCGTTATCAACTTG
要求:
(1)N为需要替换的碱基,第一个“N段”的元素和最后一个“N段”的要碱基互补配对,第二个“N段”的元素和第三个“N段”要碱基互补配对。
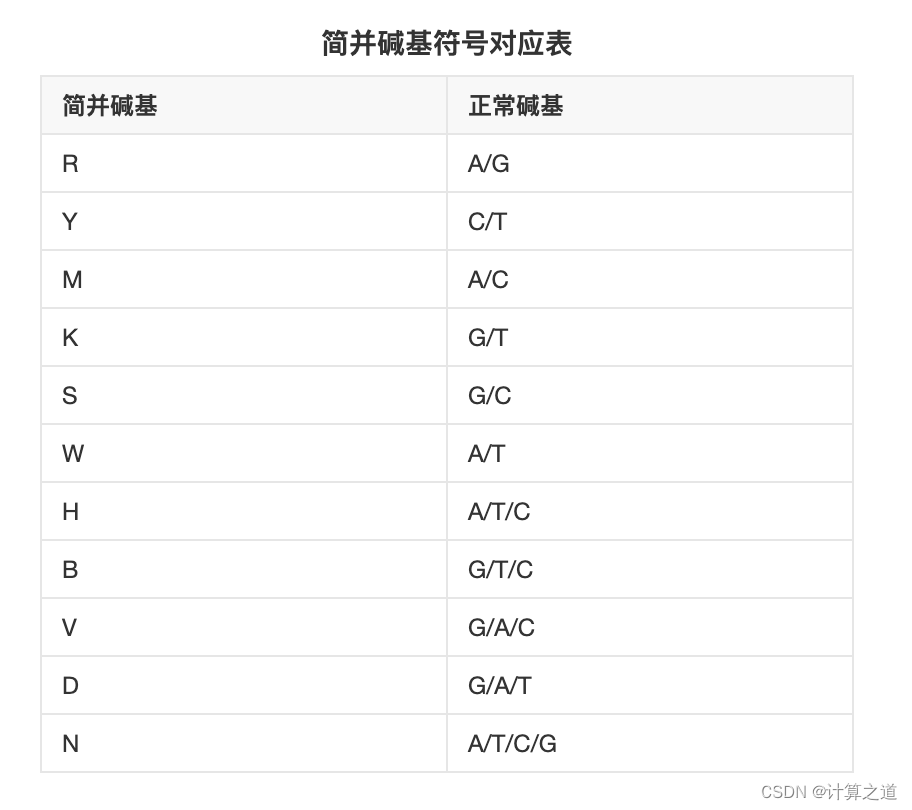
(2)要确保在生成随机序列的时候,不要生成这样的序列段:AAAA, CCCC, GGGG, TTTT, KKKKKK, MMMMMM, RRRRRR, SSSSSS, WWWWWW, YYYYYY。注意:上述K,M,R,S,W和Y为简并碱基(代表两种碱基),如下图所示:K代表了G/T,M代表了A/C,R代表了A/G,S代表了G/C,W代表了A/T,Y代表了C/T。
(3)确保没有重复序列
代码如下:
mrc@mrc-Precision-3660:~/project2/study/gene_random_DNAseq$ cat Random_DNAseq_gene2.py
import random
existing_sequences = set()
def is_valid_sequence(seq):
"""检查序列是否有效,即不包含连续的AAAA, CCCC, GGGG, TTTT或完全由某一个简并碱基表示的6碱基序列段"""
# 检查基本碱基的重复
for base in ['A', 'C', 'G', 'T']:
if base * 4 in seq:
return False
# 简并碱基对应的实际碱基
degenerate_bases = {
'K': 'GT', 'M': 'AC', 'R': 'AG', 'S': 'GC', 'W': 'AT', 'Y': 'CT'
}
# 检查是否存在完全由某一个简并碱基表示的6碱基序列段
if len(seq) >= 6:
for i in range(len(seq) - 5):
segment = seq[i:i+6]
# 检查每一个简并碱基
for bases in degenerate_bases.values():
if all(base in bases for base in segment):
return False
return True
def generate_valid_dna_sequence(length):
"""生成一个指定长度的有效DNA序列"""
while True:
sequence = ''.join(random.choice(['A', 'T', 'C', 'G']) for _ in range(length))
if is_valid_sequence(sequence):
return sequence
def generate_complementary_sequence(sequence):
"""生成互补序列"""
complement = {'A': 'T', 'T': 'A', 'C': 'G', 'G': 'C'}
return ''.join(complement[base] for base in sequence)
def create_paired_sequences(index):
"""创建配对的DNA序列片段,并在序列前添加序号"""
while True:
# 生成第一个片段
first_n_segment = generate_valid_dna_sequence(5)
# 生成第二个片段
second_n_segment = generate_valid_dna_sequence(9)
# 组合序列
dna_sequence = ("CAGGTCTCAAGTGG" + first_n_segment + "GA" + second_n_segment +
"GAAA" + generate_complementary_sequence(second_n_segment) +
"AAGT" + generate_complementary_sequence(first_n_segment) +
"TAAGGCTAGTCCGTTATCAACTTG")
# 如果组合序列不在已存在的序列集合中,则结束循环
if dna_sequence not in existing_sequences:
break
# 将组合序列添加到已存在的序列集合中
existing_sequences.add(dna_sequence)
# 格式化序号
formatted_index = str(index).zfill(6)
return f"{formatted_index} {dna_sequence}"
#return f"{index} {dna_sequence}"
# 生成10w条DNA序列,并添加序号
for i in range(1, 100001):
print(create_paired_sequences(i))
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








