文章目录
聚类
聚类是将数据集划分成组的任务,这些组叫做簇。聚类算法为每个数据点分配一个数字表示这个点属于哪个簇。
K均值聚类
k 均值聚类是最简单也最常用的聚类算法之一。它试图找到代表数据特定区域的簇中心(cluster center)。算法交替执行以下两个步骤:将每个数据点分配给最近的簇中心,然后将每个簇中心设置为所分配的所有数据点的平均值。如果簇的分配不再发生变化,那么算法结束。
import mglearn
mglearn.plots.plot_kmeans_algorithm()
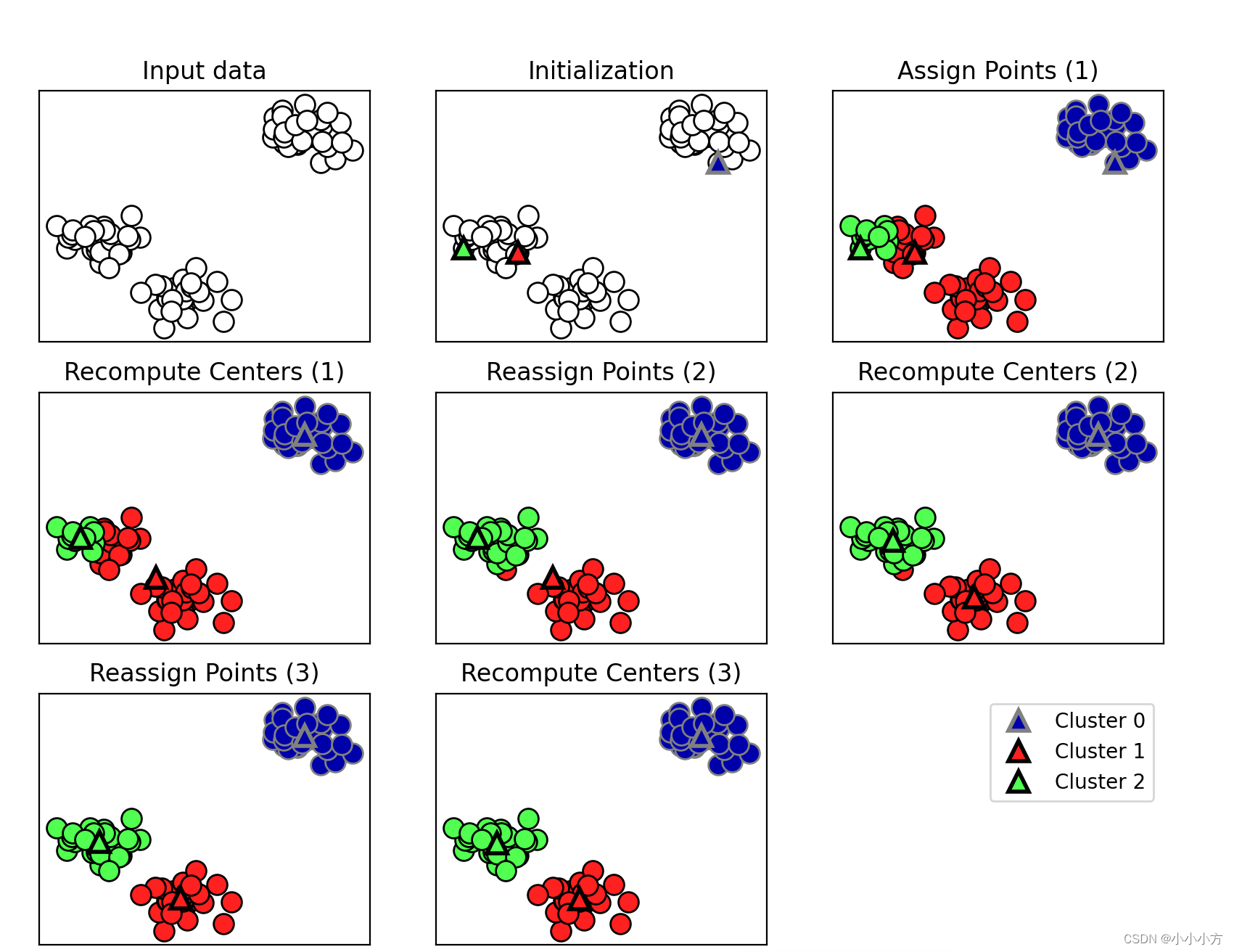
簇中心用三角形表示,而数据点用圆形表示。颜色表示簇成员。我们指定要寻找三个簇,所以通过声明三个随机数据点为簇中心来将算法初始化。然后开始迭代算法。首先,每个数据点被分配给距离最近的簇中心。接下来,将簇中心修改为所分配点的平均值。然后将这一过程再重复两次。在第三次迭代之后,为簇中心分配的数据点保持变,因此算法结束。
mglearn.plots.plot_kmeans_algorithm()
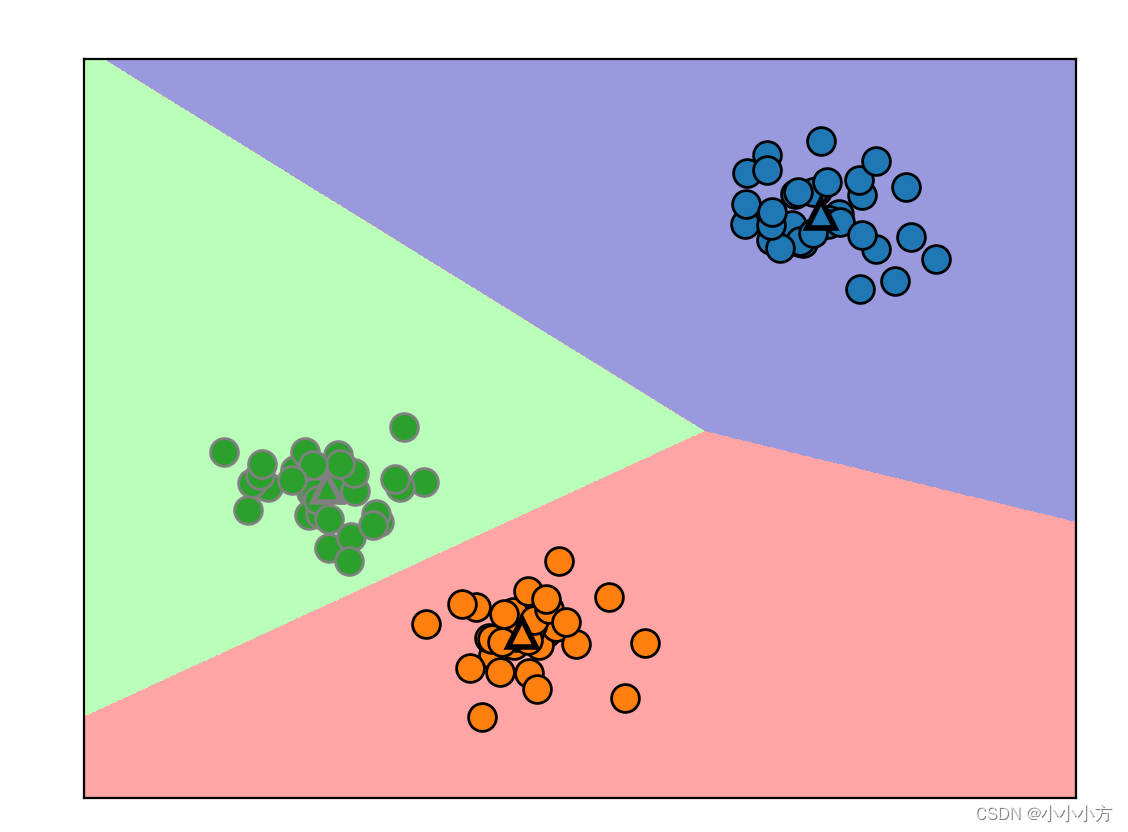
# 学到的簇中心的边界
mglearn.plots.plot_kmeans_boundaries()
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
# 生成模拟的二维数据
X, y = make_blobs(random_state=1)
# 构建聚类模型
kmeans = KMeans(n_clusters=3)
kmeans.fit(X)
# 算法运行期间,为 X 中的每个训练数据点分配一个簇标签。你可以在 kmeans.labels_ 属性中找到这些标签
print("Cluster memberships:\n{}".format(kmeans.labels_))
#用 predict 方法为新数据点分配簇标签
# 预测时会将最近的簇中心分配给每个新数据点,但现有模型不会改变
# 对训练集运行 predict 会返回与 labels_ 相同的结果
print(kmeans.predict(X))
运行结果:
Cluster memberships:
[0 2 2 2 1 1 1 2 0 0 2 2 1 0 1 1 1 0 2 2 1 2 1 0 2 1 1 0 0 1 0 0 1 0 2 1 2
2 2 1 1 2 0 2 2 1 0 0 0 0 2 1 1 1 0 1 2 2 0 0 2 1 1 2 2 1 0 1 0 2 2 2 1 0
0 2 1 1 0 2 0 2 2 1 0 0 0 0 2 0 1 0 0 2 2 1 1 0 1 0]
[0 2 2 2 1 1 1 2 0 0 2 2 1 0 1 1 1 0 2 2 1 2 1 0 2 1 1 0 0 1 0 0 1 0 2 1 2
2 2 1 1 2 0 2 2 1 0 0 0 0 2 1 1 1 0 1 2 2 0 0 2 1 1 2 2 1 0 1 0 2 2 2 1 0
0 2 1 1 0 2 0 2 2 1 0 0 0 0 2 0 1 0 0 2 2 1 1 0 1 0]
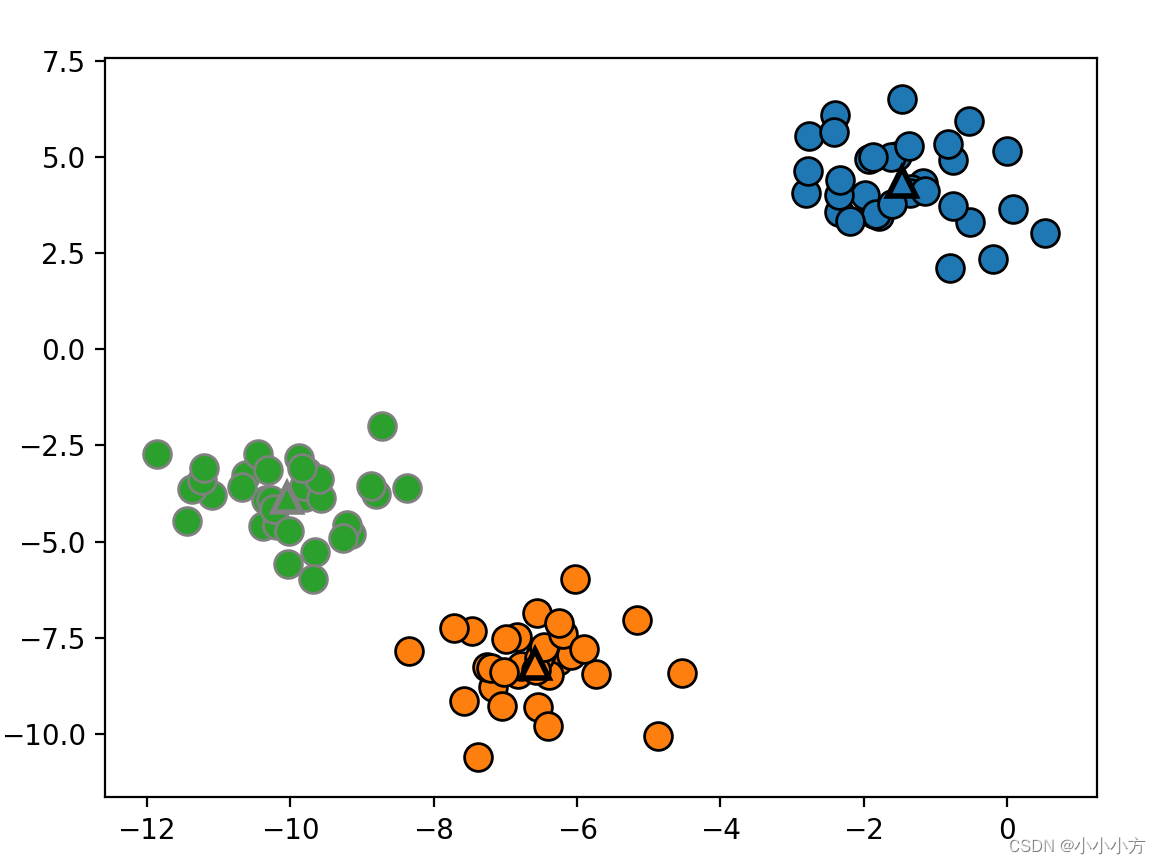
聚类算法与分类算法有些相似,每个元素都有一个标签。但并不存在真实的标签,因此标签本身并没有先验意义.
#簇中心被保存在 cluster_centers_ 属性中
mglearn.discrete_scatter(X[:, 0], X[:, 1], kmeans.labels_, markers='o')
mglearn.discrete_scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1], [0, 1, 2],markers='^', markeredgewidth=2)
plt.show()
# 使用更多或更少的簇中心
fig, axes = plt.subplots(1, 2, figsize=(10, 5))
# 使用2个簇中心:
kmeans = KMeans(n_clusters=2)
kmeans.fit(X)
assignments = kmeans.labels_
mglearn.discrete_scatter(X[:, 0], X[:, 1], assignments, ax=axes[0])
# 使用5个簇中心:
kmeans = KMeans(n_clusters=5)
kmeans.fit(X)
assignments = kmeans.labels_
mglearn.discrete_scatter(X[:, 0], X[:, 1], assignments, ax=axes[1])
plt.show()
K均值失败的案例
即使知道给定数据集中簇的正确个数,k值可能也不是总能找到它们。每个簇仅由其中心定义,这意味每个簇都是凸性的。k均值假设所有簇在某种程度上具有相同的直径,它总是将簇之间的边界刚好 画在簇的中间位置。
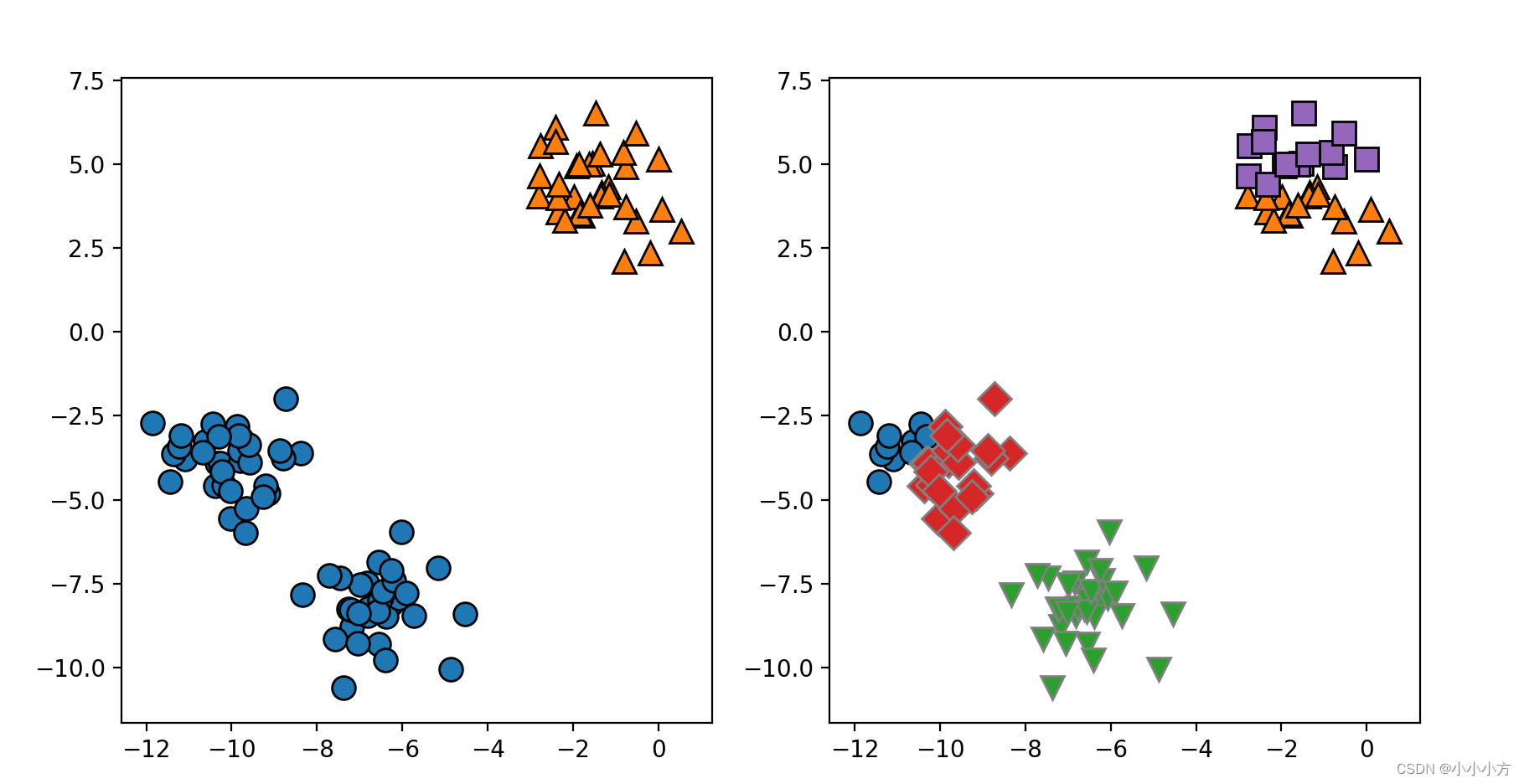
X_varied, y_varied = make_blobs(n_samples=200,cluster_std=[1.0, 2.5, 0.5],random_state=170)
y_pred = KMeans(n_clusters=3, random_state=0).fit_predict(X_varied)
mglearn.discrete_scatter(X_varied[:, 0], X_varied[:, 1], y_pred)
plt.legend(["cluster 0", "cluster 1", "cluster 2"], loc='best')
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
可能会认为,左下方的密集区域是第一个簇,右上方的密集区域是第二个,中间密度较小的区域是第三个。但事实上,簇 0 和簇 1 都包含一些远离簇中其他点的点。
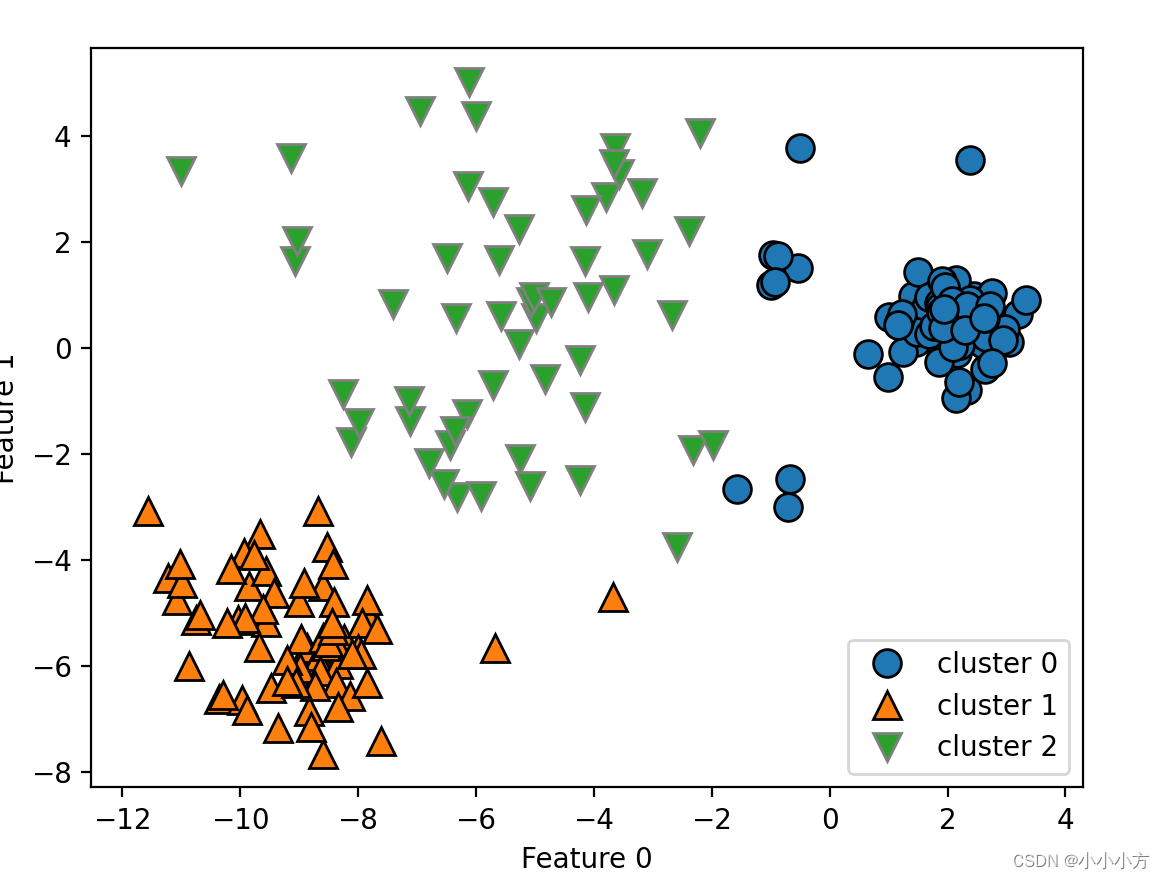
# 生成一些随机分组数据
X, y = make_blobs(random_state=170, n_samples=600)
# 通过numpy工具包生成模拟数据集,使用RandomState获得随机数生成器
rng = np.random.RandomState(74)
# 变换数据使其拉长
transformation = rng.normal(size=(2, 2))
X = np.dot(X, transformation)
# 将数据聚类成3个簇
kmeans = KMeans(n_clusters=3)
kmeans.fit(X)
y_pred = kmeans.predict(X)
# 画出簇分配和簇中心
plt.scatter(X[:, 0], X[:, 1], c=y_pred, cmap=mglearn.cm3)
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1],marker='^', c=[0, 1, 2], s=100, linewidth=2, cmap=mglearn.cm3)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
k均值假设所有方向对每个簇都同等重要,数据中包含明确分开的三个部分,但是这三个部分被沿着对角线方向拉长,由于K均值仅考虑到最近簇中心距离,所以它无法处理这种数据。
如果簇的形状更加复杂,k均值的表现也很差
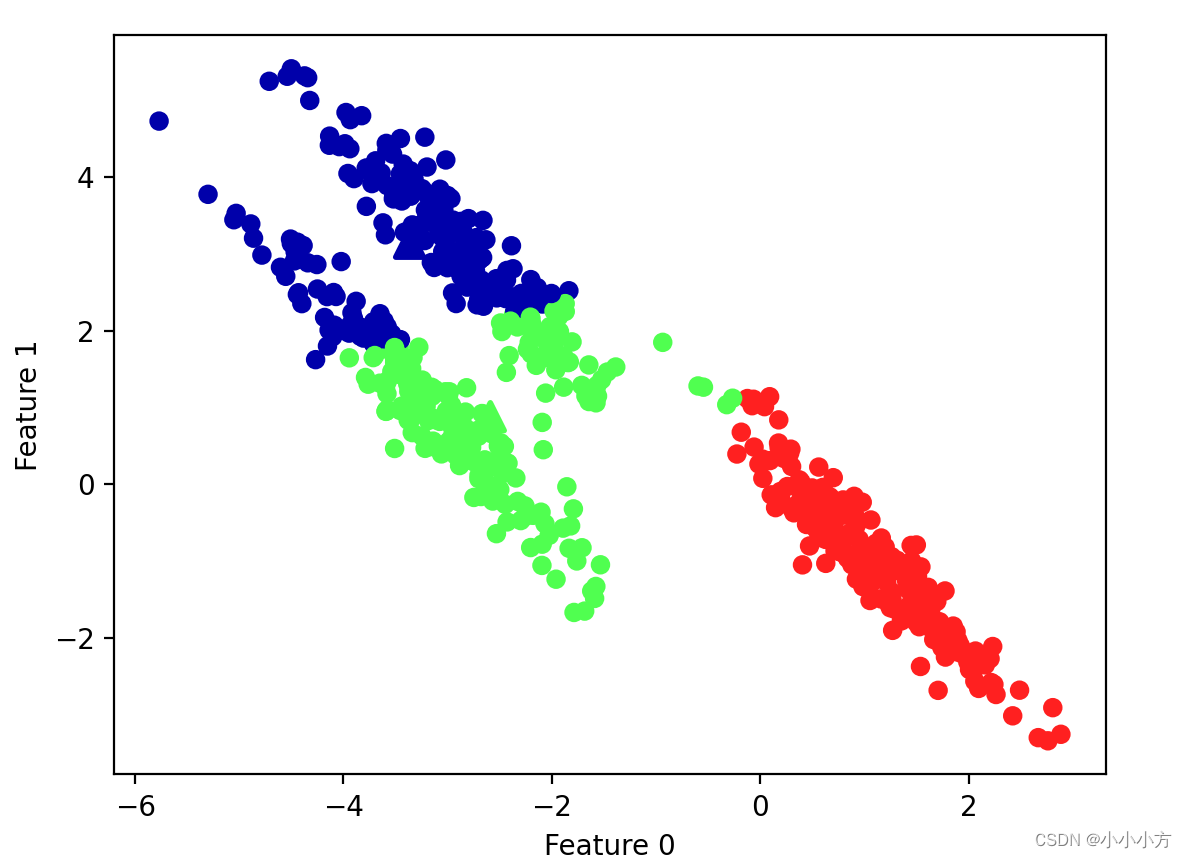
# 生成模拟的two_moons数据(这次的噪声较小)
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据聚类成2个簇
kmeans = KMeans(n_clusters=2)
kmeans.fit(X)
y_pred = kmeans.predict(X)
# 画出簇分配和簇中心
plt.scatter(X[:, 0], X[:, 1], c=y_pred, cmap=mglearn.cm2, s=60)
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1],marker='^', c=[mglearn.cm2(0), mglearn.cm2(1)], s=100, linewidth=2)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
k均值不能发现两个半月形的数据。
矢量化或将k值看作分解
PCA试图找到数据中方差最大的方向,NMF试图找到累加的分量,这两种方法都尝试将数据点表示为一些分量之和,K均值尝试利用簇中心来表示每个数据点,可以将其看作只用一个分量来表示每个数据点,该分量有簇中心给出。将k均值看作是一种分解方法,其中每个点用单一分量来表示,这种观点被称为矢量化。
from sklearn.datasets import fetch_lfw_people
from sklearn.decomposition import NMF, PCA
people = fetch_lfw_people(min_faces_per_person=20, resize=0.7)
image_shape = people.images[0].shape
mask = np.zeros(people.target.shape, dtype=np.bool)
for target in np.unique(people.target):
mask[np.where(people.target == target)[0][:50]] = 1
X_people = people.data[mask]
y_people = people.target[mask]
X_people = X_people / 255.
# 比较PCA、NMF和K均值分别提取的分量
X_train, X_test, y_train, y_test = train_test_split(X_people, y_people, stratify=y_people, random_state=0)
nmf = NMF(n_components=100, random_state=0)
nmf.fit(X_train)
pca = PCA(n_components=100, random_state=0)
pca.fit(X_train)
kmeans = KMeans(n_clusters=100, random_state=0)
kmeans.fit(X_train)
# 利用100个分量对测试机种人脸的重建
X_reconstructed_pca = pca.inverse_transform(pca.transform(X_test))
X_reconstructed_kmeans = kmeans.cluster_centers_[kmeans.predict(X_test)]
X_reconstructed_nmf = np.dot(nmf.transform(X_test), nmf.components_)
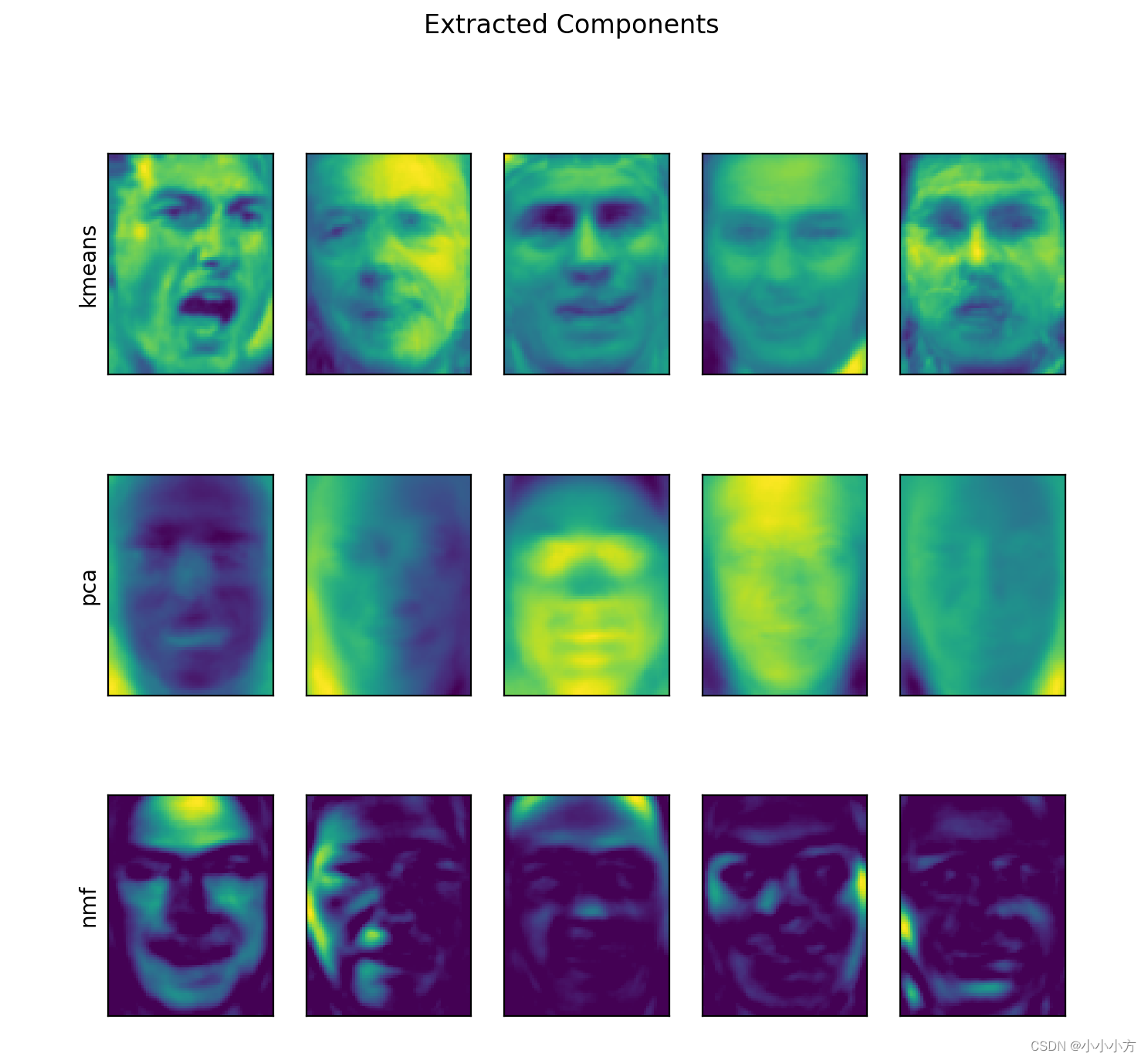
fig, axes = plt.subplots(3, 5, figsize=(8, 8),subplot_kw={'xticks': (), 'yticks': ()})
fig.suptitle("Extracted Components")
for ax, comp_kmeans, comp_pca, comp_nmf in zip(axes.T, kmeans.cluster_centers_, pca.components_, nmf.components_):
ax[0].imshow(comp_kmeans.reshape(image_shape))
ax[1].imshow(comp_pca.reshape(image_shape), cmap='viridis')
ax[2].imshow(comp_nmf.reshape(image_shape))
axes[0, 0].set_ylabel("kmeans")
axes[1, 0].set_ylabel("pca")
axes[2, 0].set_ylabel("nmf")
plt.show()
fig, axes = plt.subplots(4, 5, subplot_kw={'xticks': (), 'yticks': ()},figsize=(8, 8))
fig.suptitle("Reconstructions")
for ax, orig, rec_kmeans, rec_pca, rec_nmf in zip(axes.T, X_test, X_reconstructed_kmeans, X_reconstructed_pca,X_reconstructed_nmf):
ax[0].imshow(orig.reshape(image_shape))
ax[1].imshow(rec_kmeans.reshape(image_shape))
ax[2].imshow(rec_pca.reshape(image_shape))
ax[3].imshow(rec_nmf.reshape(image_shape))
axes[0, 0].set_ylabel("original")
axes[1, 0].set_ylabel("kmeans")
axes[2, 0].set_ylabel("pca")
axes[3, 0].set_ylabel("nmf")
plt.show()

利用 k 均值做矢量量化的一个有趣之处在于,可以用比输入维度更多的簇来对数据进行编码。让我们回到 two_moons 数据。利用 PCA 或 NMF,我们对这个数据无能为力,因为它只有两个维度。使用 PCA 或 NMF 将其降到一维,将会完全破坏数据的结构。但通过使用更多的簇中心,我们可以用 k 均值找到一种更具表现力的表示。
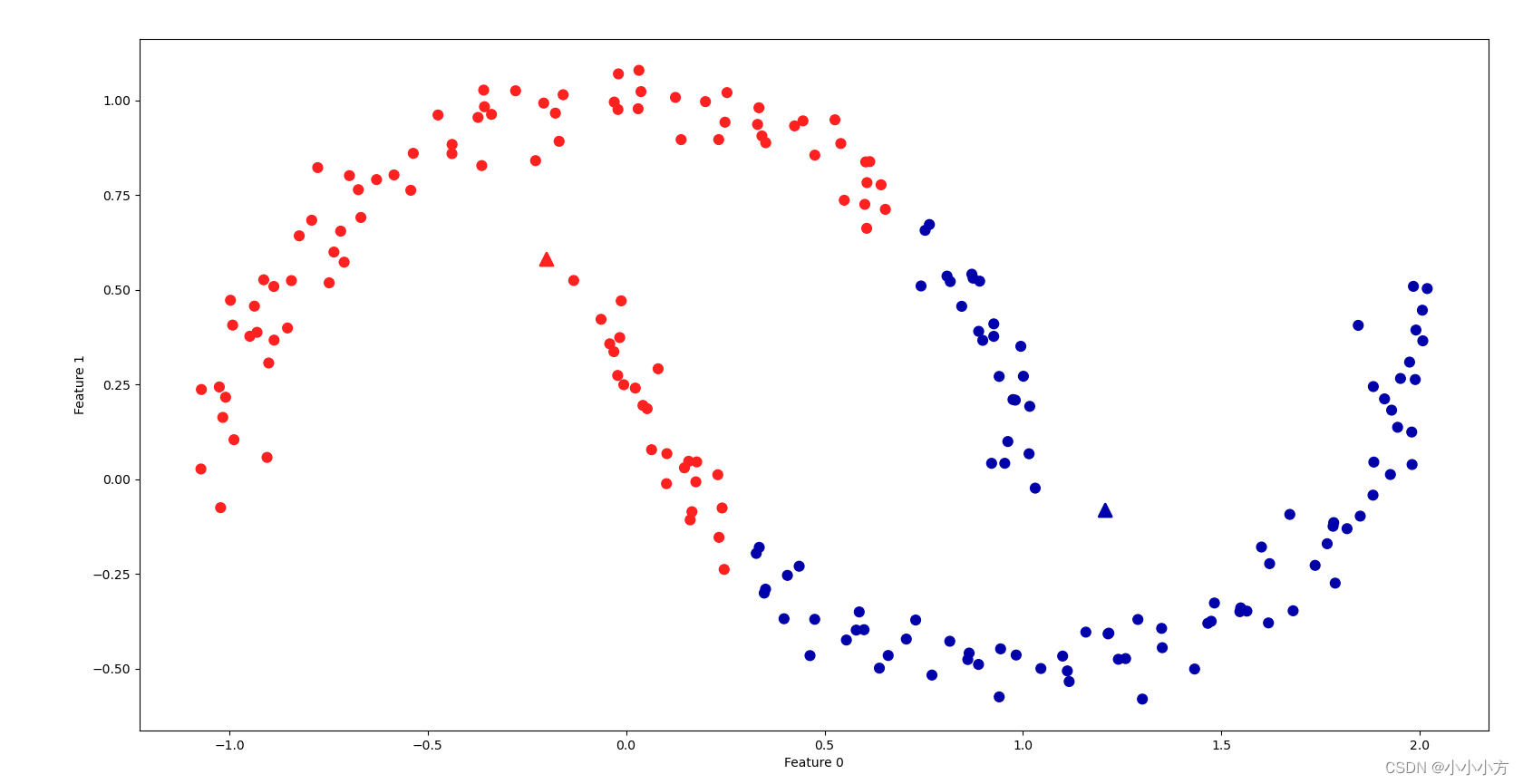
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
kmeans = KMeans(n_clusters=10, random_state=0)
kmeans.fit(X)
y_pred = kmeans.predict(X)
plt.scatter(X[:, 0], X[:, 1], c=y_pred, s=60, cmap='Paired')
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1], s=60,marker='^', c=range(kmeans.n_clusters), linewidth=2, cmap='Paired')
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
print("Cluster memberships:\n{}".format(y_pred))
运行结果:
Cluster memberships:
[9 2 5 4 2 7 9 6 9 6 1 0 2 6 1 9 3 0 3 1 7 6 8 6 8 5 2 7 5 8 9 8 6 5 3 7 0
9 4 5 0 1 3 5 2 8 9 1 5 6 1 0 7 4 6 3 3 6 3 8 0 4 2 9 6 4 8 2 8 4 0 4 0 5
6 4 5 9 3 0 7 8 0 7 5 8 9 8 0 7 3 9 7 1 7 2 2 0 4 5 6 7 8 9 4 5 4 1 2 3 1
8 8 4 9 2 3 7 0 9 9 1 5 8 5 1 9 5 6 7 9 1 4 0 6 2 6 4 7 9 5 5 3 8 1 9 5 6
3 5 0 2 9 3 0 8 6 0 3 3 5 6 3 2 0 2 3 0 2 6 3 4 4 1 5 6 7 1 1 3 2 4 7 2 7
3 8 6 4 1 4 3 9 9 5 1 7 5 8 2]

我们使用了 10 个簇中心,也就是说,现在每个点都被分配了 0 到 9 之间的一个数字。我们可以将其看作 10 个分量表示的数据(我们有 10 个新特征),只有表示该点对应的簇中心的那个特征不为 0,其他特征均为 0。利用这个 10 维表示,现在可以用线性模型来划分两个
半月形,而利用原始的两个特征是不可能做到这一点的。
# 将到每个簇中心的距离作为特征,还可以得到一种表现力更强的数据表示
distance_features = kmeans.transform(X)
print("Distance feature shape: {}".format(distance_features.shape))
print("Distance features:\n{}".format(distance_features))
运行结果:
Distance feature shape: (200, 10)
Distance features:
[[0.9220768 1.46553151 1.13956805 ... 1.16559918 1.03852189 0.23340263]
[1.14159679 2.51721597 0.1199124 ... 0.70700803 2.20414144 0.98271691]
[0.78786246 0.77354687 1.74914157 ... 1.97061341 0.71561277 0.94399739]
...
[0.44639122 1.10631579 1.48991975 ... 1.79125448 1.03195812 0.81205971]
[1.38951924 0.79790385 1.98056306 ... 1.97788956 0.23892095 1.05774337]
[1.14920754 2.4536383 0.04506731 ... 0.57163262 2.11331394 0.88166689]]
k均值是相对容易理解和实现,运行速度也相对较快,可以轻松扩展到大型数据集上。缺点之一在于它依赖于随机初始化,即算法的输出依赖于随机种子。另一个缺点是对簇形状的假设约束性较强,而且还要求指定所要寻找的簇的个数。
凝聚聚类
指的是许多基于相同原则构建的聚类算法。算法首先声明每个点是自己的簇,然后合并两个最相似的簇,知道满足某种停止准则为为止。scikit-learn 中实现的停止准则是簇的个数,因此相似的簇被合并,直到仅剩下指定个数的簇。还有一些链接(linkage)准则,规定如何度量“最相似的簇”。这种度量总是定义在两个现有的簇之间。
scikit-learn 中实现了以下三种选项:
ward:默认选项,挑选两个簇来进行合并,使得所有簇中的方差增加最小,通常会得到大小差不多相等的簇。
average链接将簇中所有点之间平均距离最小的两个簇合并。
complete链接将簇中所有点之间平均距离最小的两个簇进行合并。
import mglearn
mglearn.plots.plot_agglomerative_algorithm()
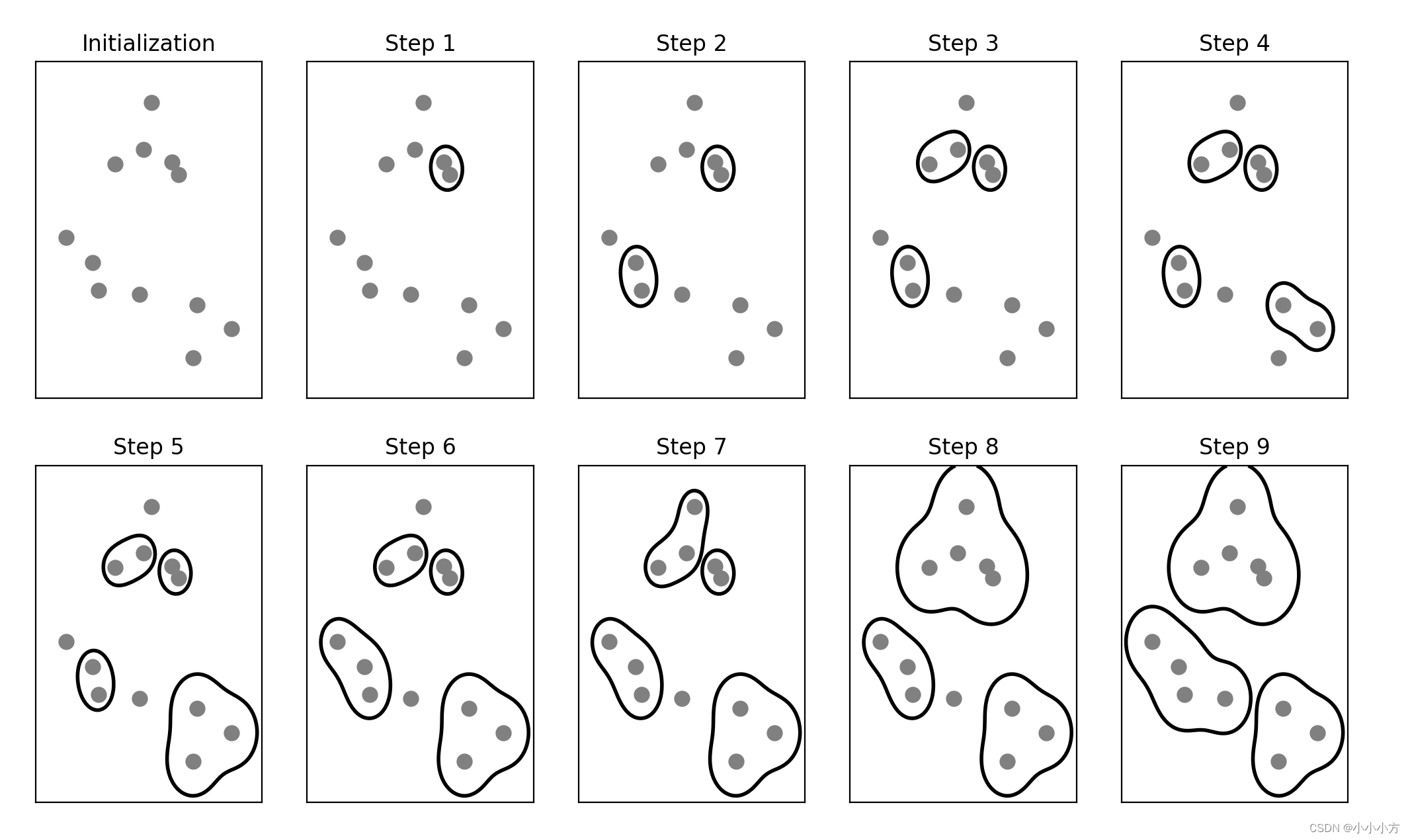
由于算法的工作原理,凝聚算法不能对新数据点做出预测,所以没有predict方法,为了构造模型并得到训练集上簇的成员关系,可以使用fit_predict方法。
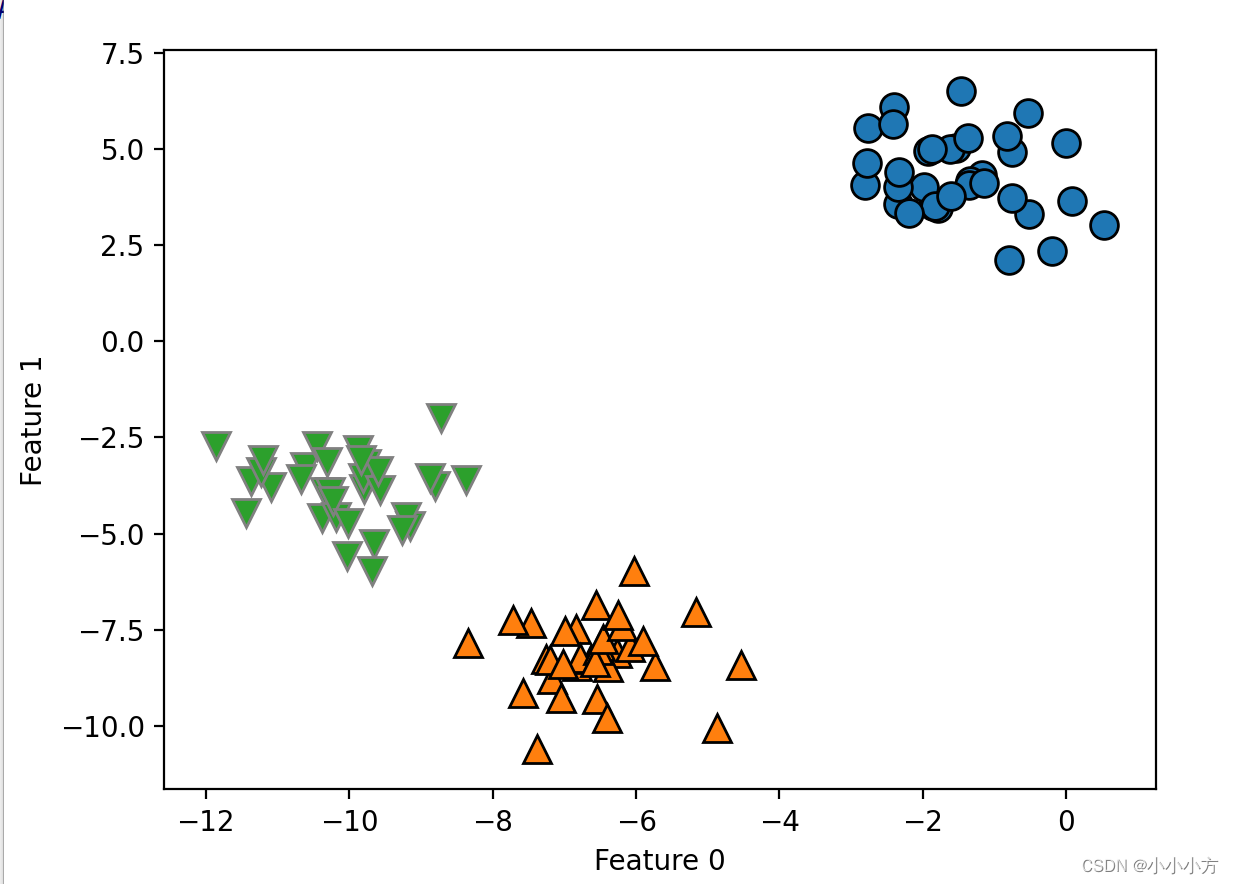
from sklearn.cluster import AgglomerativeClustering
X, y = make_blobs(random_state=1)
agg = AgglomerativeClustering(n_clusters=3)
assignment = agg.fit_predict(X)
mglearn.discrete_scatter(X[:, 0], X[:, 1], assignment)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
层次聚类与树状图
凝聚聚类生成了层次聚类。聚类过程迭代进行,每个点都从一个单点簇变为属于最终的某个簇。每个中间步骤都提供了数据的一种聚类(簇的个数也不相同),有时同时查看所有可能的聚类是有帮助的。
mglearn.plots.plot_agglomerative()

但是这种可视化不能用于具有两个以上特征的数据集。scikit-learn 没有绘制树状图的功能,可以利用 SciPy 轻松生成树状图。SciPy 的聚类算法接口与 scikit-learn 的聚类算法稍有不同。SciPy 提供了一个函数,接受数据数组 X 并计算出一个链接数组(linkage array),它对层次聚类的相似度进行编码。然后我们可以将这个链接数组提供给 scipy 的 dendrogram 函数来绘制树状图。
# 绘制树状图
# 从SciPy中导入dendrogram函数和ward聚类函数
from scipy.cluster.hierarchy import dendrogram, ward
X, y = make_blobs(random_state=0, n_samples=12)
# 将ward聚类应用于数据数组X
# SciPy的ward函数返回一个数组,指定执行凝聚聚类时跨越的距离
linkage_array = ward(X)
# 现在为包含簇之间距离的linkage_array绘制树状图
dendrogram(linkage_array)
# 在树中标记划分成两个簇或三个簇的位置
# 获取当前的坐标轴
ax = plt.gca()
# 设置x轴的上下边界
bounds = ax.get_xbound()
ax.plot(bounds, [7.25, 7.25], '--', c='k')
ax.plot(bounds, [4, 4], '--', c='k')
ax.text(bounds[1], 7.25, ' two clusters', va='center', fontdict={'size': 15})
ax.text(bounds[1], 4, ' three clusters', va='center', fontdict={'size': 15})
plt.xlabel("Sample index")
plt.ylabel("Cluster distance")
plt.show()
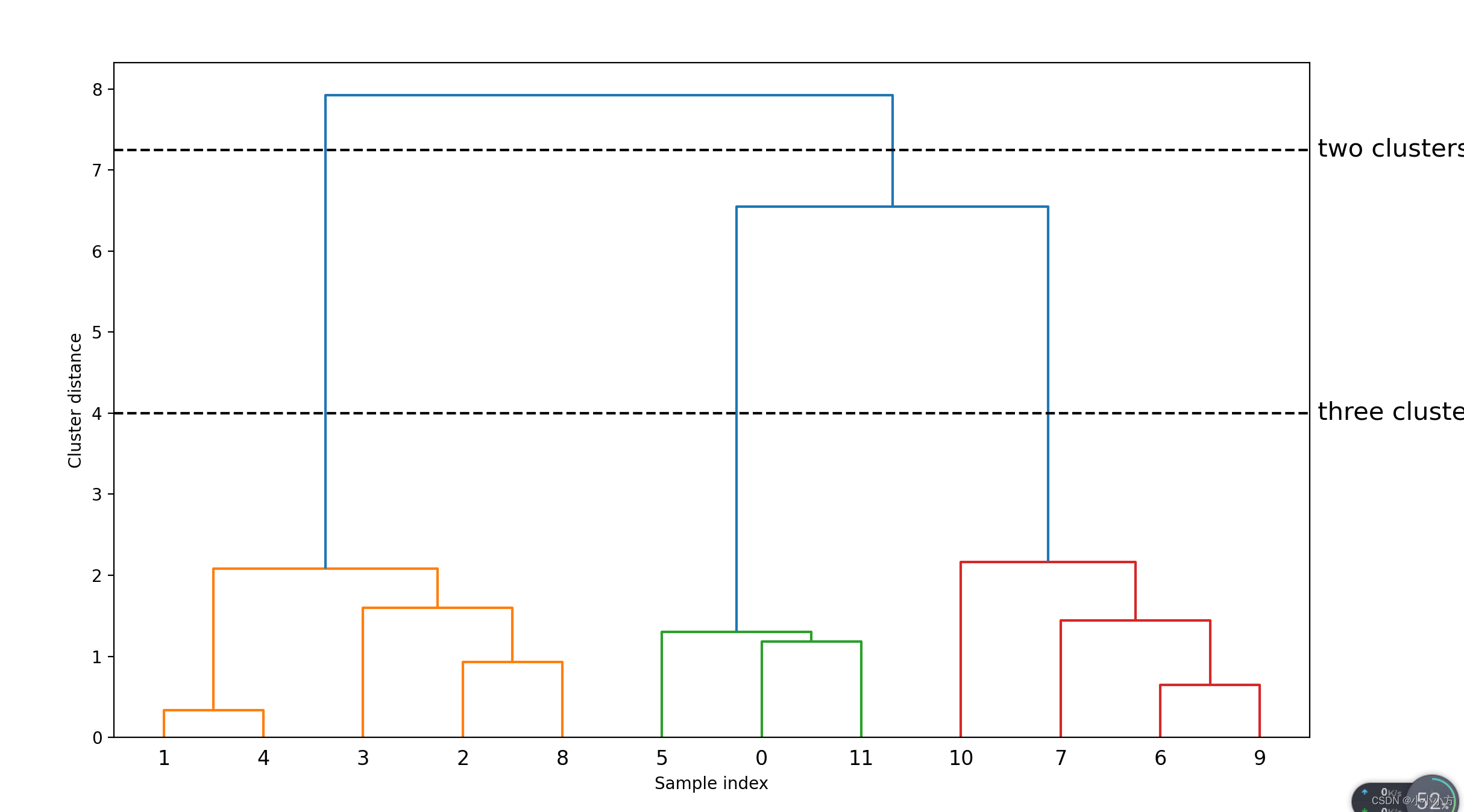
树状图在底部显示数据点,以这些点作为叶节点绘制一棵树,每合并两个簇就添加一个新的父节点。
从下往上看,数据点 1 和 4 首先被合并。接下来,点 6 和 9 被合并为一个簇,以此类推。在顶层有两个分支,一个由点 11、0、5、10、7、6 和 9 组成,另一个由点 1、4、3、2 和 8 组成。这对应于图中左侧两个最大的簇。
树状图的y轴不仅说明凝聚算法中两个簇何时合并,每个分支的长度还表示被合并的簇之间的距离。在这张树状图中,最长的分支是用标记为three clusters的虚线表示的三条线。它们是最长的分支,这表示从三个簇到两个簇的过程中合并了一些距离非常远的点。但是,凝聚聚类也无法分离 two_moons 数据集这样复杂的形状。
DNSCAN
DNSCAN是具有噪声的基于密度的空间聚类应用。主要优点是用户不必先验的设置簇的个数,可以划分具有复杂形状的簇,还可以找到不属于任何簇的点,虽然比较慢但是可以扩展到相对较大的数据集。他的原理是识别特征空间的拥挤区域中的点,在这些区域中许多数据点靠近在一起。这些区域被称为特征空间中的密集(dense)区域。DBSCAN 背后的思想是,簇形成数据的密集区域,并由相对较空的区域分隔开。在密集区域内的点被称为核心样本:DBSCAN有两个参数:min_samples 和 eps,如果在距一个给定数据点 eps 的距离内至少有 min_samples 个数据点,那么这个数据点就是核心样本。DBSCAN 将彼此距离小于 eps 的核心样本放到同一个簇中。
算法首先任意选取一个点,然后找到这个点的距离小于等于eps的所有的点,如果距离起始点的距离在eps之内的数据点个数小于min_samples,那么这个点被标记为噪声,也就是说它不属于任何簇,如果距离在eps之内的数据点个数大于min_samples,则这个点被标记为核心样本,并被分配给一个新的簇标签。然后访问该点的所有邻居(在eps距离之内),如果它们还没有被分配一个簇,就将刚刚创建的新的簇标签分配给它们,如果它们是核心样本,那么就依次访问其邻居,以此类推。簇逐渐增大,直到在簇的 eps 距离内没有更多的核心样本为止。然后选取另一个尚未被访问过的点,并重复相同的过程。
一共有三种类型的点:核心点、与核心点的距离在 eps 之内的点(叫作边界点,boundary point)和噪声。如果 DBSCAN 算法在特定数据集上多次运行,那么核心点的聚类始终相同,同样的点也始终被标记为噪声。但边界点可能与不止一个簇的核心样本相邻。因此,边界点所属的簇依赖于数据点的访问顺序。一般来说只有很少的边界点,这种对访问顺序的轻度依赖并不重要。
DBSCAN 不允许对新的测试数据进行预测,所以我们将使用 fit_predict 方法来执行聚类并返回簇标签。
from sklearn.cluster import DBSCAN
X, y = make_blobs(random_state=0, n_samples=12)
dbscan = DBSCAN()
clusters = dbscan.fit_predict(X)
print("Cluster memberships:\n{}".format(clusters))
mglearn.plots.plot_dbscan()
运行结果:
# eps 和 min_samples
# 默认参数设置的结果,对于小型的玩具数据集并没有调节这些参数
[-1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1]
min_samples: 2 eps: 1.000000 cluster: [-1 0 0 -1 0 -1 1 1 0 1 -1 -1]
min_samples: 2 eps: 1.500000 cluster: [0 1 1 1 1 0 2 2 1 2 2 0]
min_samples: 2 eps: 2.000000 cluster: [0 1 1 1 1 0 0 0 1 0 0 0]
min_samples: 2 eps: 3.000000 cluster: [0 0 0 0 0 0 0 0 0 0 0 0]
min_samples: 3 eps: 1.000000 cluster: [-1 0 0 -1 0 -1 1 1 0 1 -1 -1]
min_samples: 3 eps: 1.500000 cluster: [0 1 1 1 1 0 2 2 1 2 2 0]
min_samples: 3 eps: 2.000000 cluster: [0 1 1 1 1 0 0 0 1 0 0 0]
min_samples: 3 eps: 3.000000 cluster: [0 0 0 0 0 0 0 0 0 0 0 0]
min_samples: 5 eps: 1.000000 cluster: [-1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1]
min_samples: 5 eps: 1.500000 cluster: [-1 0 0 0 0 -1 -1 -1 0 -1 -1 -1]
min_samples: 5 eps: 2.000000 cluster: [-1 0 0 0 0 -1 -1 -1 0 -1 -1 -1]
min_samples: 5 eps: 3.000000 cluster: [0 0 0 0 0 0 0 0 0 0 0 0]
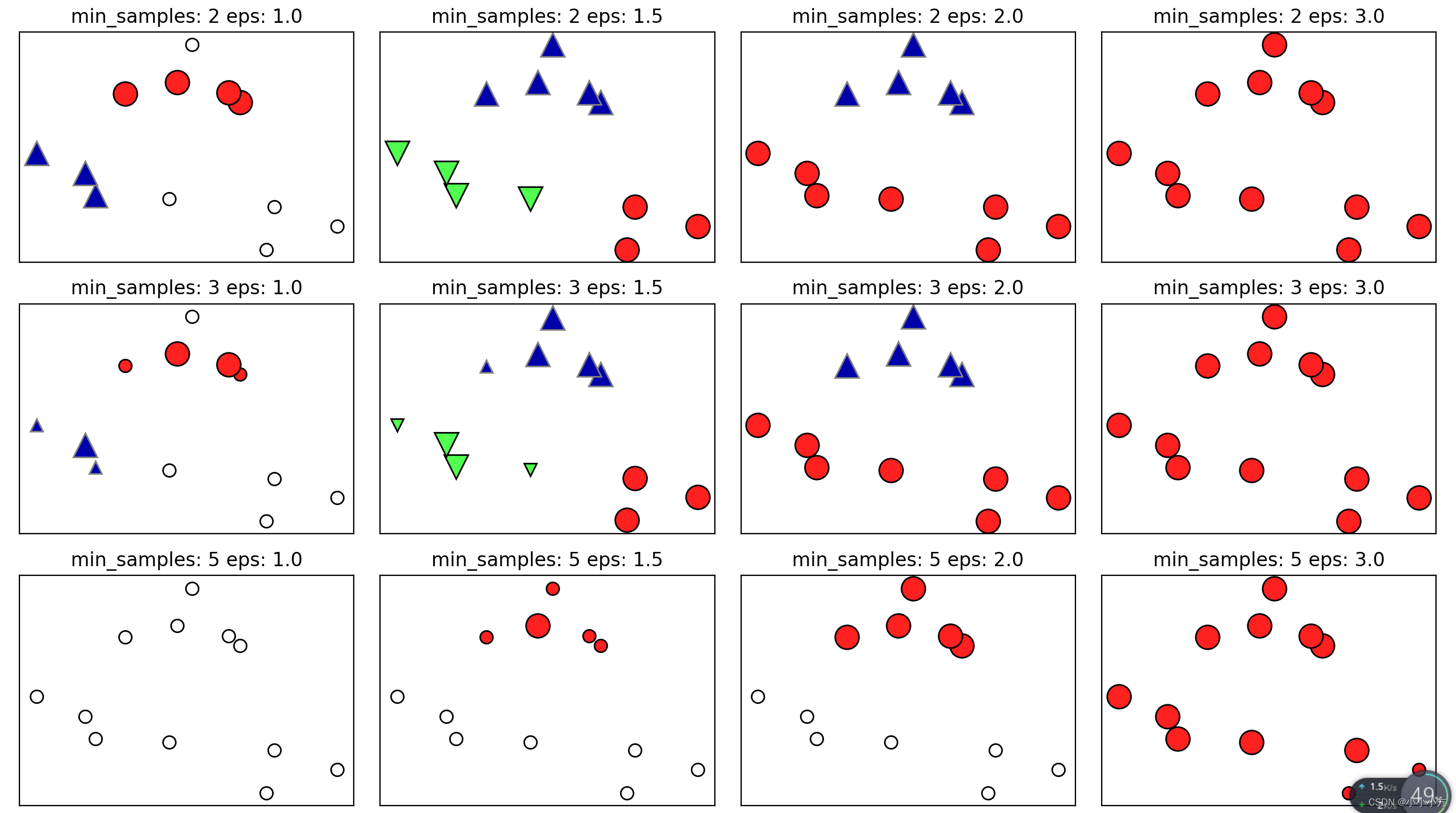
图中,属于簇的点是实心的,而噪声点则显示为空心的。核心样本显示为较大的标记,边界点显示为较小的标记。增大eps,更多的点会被包在一个簇中,让簇变大,也可能是多个簇合并成一个簇,增大min_samples核心点会变得更少,更多的点被标记为噪声。
参数 eps 在某种程度上更加重要,因为它决定了点与点之间“接近”的含义。将 eps 设置得非常小,意味着没有点是核心样本,可能会导致所有点都被标记为噪声。将 eps 设置得非常大,可能会导致所有点形成单个簇。
设置 min_samples 主要是为了判断稀疏区域内的点被标记为异常值还是形成自己的簇。min_samples 决定簇的最小尺寸。虽然 DBSCAN 不需要显式地设置簇的个数,但设置 eps 可以隐式地控制找到的簇的个数。
使用 StandardScaler 或 MinMaxScaler 对数据进行缩放之后,有时会更容易找到 eps 的较好取值,因为使用这些缩放技术将确保所有特征具有相似的范围。
# 进行数据缩放
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据缩放成平均值为0、方差为1
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
dbscan = DBSCAN()
clusters = dbscan.fit_predict(X_scaled)
# 绘制簇分配
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=clusters, cmap=mglearn.cm2, s=60)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
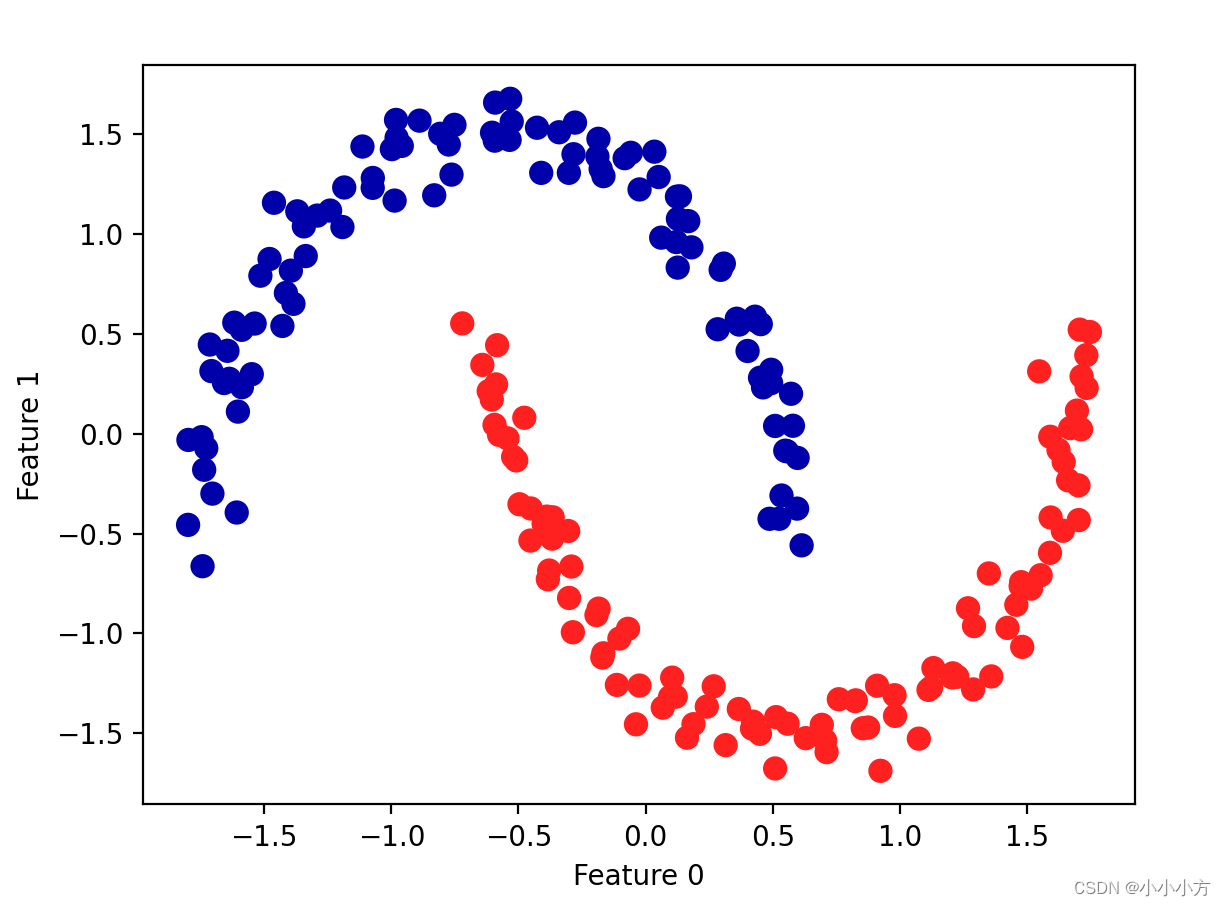
算法找到了我们想要的簇的个数,参数设置的效果似乎很好,如果将eps减小到0.2,我们将会得到8个簇,将eps增大到0.7则会导致一个簇。在使用DBSCAN时,要谨慎处理返回的簇分配,如果使用簇标签对另一个数据进行索引,那么使用-1表示噪声可能会产生意料之外的结果。
聚类算法的对比与评估
聚类算法的挑战之一就是很难评估一个算法的好坏,也很难比较不同算法的结果。
用真实值评估聚类
评估聚类算法相对于真实聚类结果的指标,最重要的是调整rand指数ARI和归一化信息NMI,都给出了定量的度量,最佳值为1,0表示不相关的聚类。
# 使用ARI
from sklearn.metrics.cluster import adjusted_rand_score
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据缩放成平均值为0、方差为1
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
fig, axes = plt.subplots(1, 4, figsize=(15, 3),subplot_kw={'xticks': (), 'yticks': ()})
# 列出要使用的算法
algorithms = [KMeans(n_clusters=2), AgglomerativeClustering(n_clusters=2),DBSCAN()]
# 创建一个随机的簇分配,作为参考
random_state = np.random.RandomState(seed=0)
random_clusters = random_state.randint(low=0, high=2, size=len(X))
# 绘制随机分配
axes[0].scatter(X_scaled[:, 0], X_scaled[:, 1], c=random_clusters,cmap=mglearn.cm3, s=60)
axes[0].set_title("Random assignment - ARI: {:.2f}".format(adjusted_rand_score(y, random_clusters)))
for ax, algorithm in zip(axes[1:], algorithms):
# 绘制簇分配和簇中心
clusters = algorithm.fit_predict(X_scaled)
ax.scatter(X_scaled[:, 0], X_scaled[:, 1], c=clusters,cmap=mglearn.cm3, s=60)
ax.set_title("{} - ARI: {:.2f}".format(algorithm.__class__.__name__,adjusted_rand_score(y, clusters)))
plt.show()
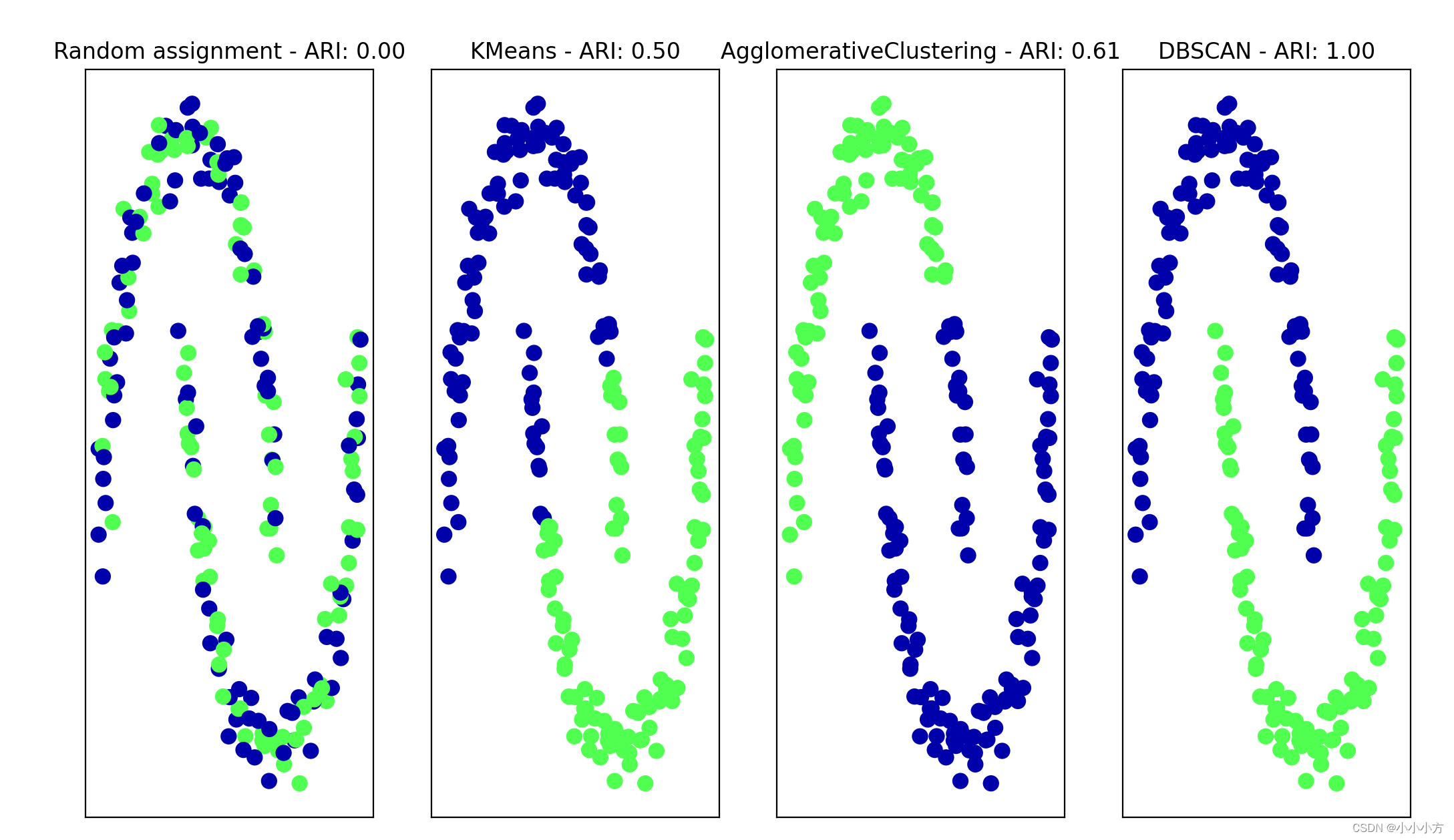
用这种方式评估聚类时,一个常见的错误是使用accuracy_score而不是使用adjusted_rand_score、normalized_mutual_info_score 或其他聚类指标。使用精度的问题在于,它要求分配的簇标签与真实值完全匹配。但簇标签本身毫无意义——唯一重要的是哪些点位于同一个簇中。
from sklearn.metrics import accuracy_score
# 这两种点标签对应于相同的聚类
clusters1 = [0, 0, 1, 1, 0]
clusters2 = [1, 1, 0, 0, 1]
# 精度为0,因为二者标签完全不同
print("Accuracy: {:.2f}".format(accuracy_score(clusters1, clusters2)))
# 调整rand分数为1,因为二者聚类完全相同
print("ARI: {:.2f}".format(adjusted_rand_score(clusters1, clusters2)))
运行结果:
Accuracy: 0.00
ARI: 1.00
在没有真实值的情况下评估聚类
在应用聚类算法时,通常没有真实值来比较结果,如果我们知道数据正确聚类,那么可以使用这一信息构建一个监督模型,所以使用ARI和NMI的之末仅仅有助于开发蒜贩,但是对评估应用是否成功没有帮助。有一些聚类的评分指标不需要真实值,比如轮廓系数。轮廓系数计算一个簇的紧致度,值越大越好,最高分数为1,虽然紧致的簇很好,但紧致度不允许复杂的形状。
from sklearn.metrics.cluster import silhouette_score
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据缩放成平均值为0、方差为1
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
fig, axes = plt.subplots(1, 4, figsize=(15, 3),subplot_kw={'xticks': (), 'yticks': ()})
# 创建一个随机的簇分配,作为参考
random_state = np.random.RandomState(seed=0)
random_clusters = random_state.randint(low=0, high=2, size=len(X))
# 绘制随机分配
axes[0].scatter(X_scaled[:, 0], X_scaled[:, 1], c=random_clusters,cmap=mglearn.cm3, s=60)
axes[0].set_title("Random assignment: {:.2f}".format(silhouette_score(X_scaled, random_clusters)))
algorithms = [KMeans(n_clusters=2), AgglomerativeClustering(n_clusters=2),DBSCAN()]
for ax, algorithm in zip(axes[1:], algorithms):
clusters = algorithm.fit_predict(X_scaled)
# 绘制簇分配和簇中心
ax.scatter(X_scaled[:, 0], X_scaled[:, 1], c=clusters, cmap=mglearn.cm3,s=60)
ax.set_title("{} : {:.2f}".format(algorithm.__class__.__name__,silhouette_score(X_scaled, clusters)))
plt.show()
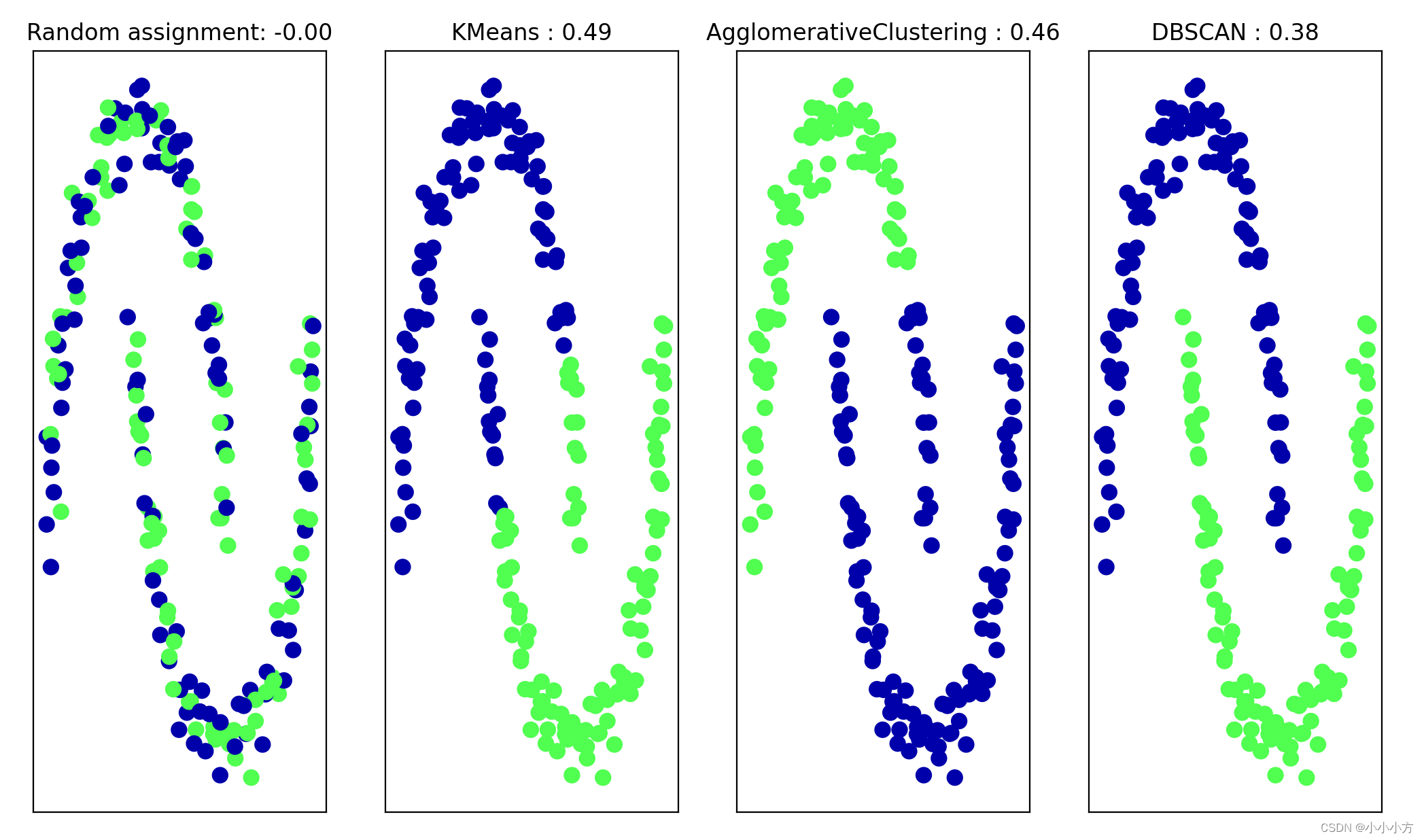
k均值的轮廓分数最高,但是DBSCAN的结果更正确。对于评估聚类,稍好的策略是使用基于鲁棒性的
在人脸数据集上比较算法
# 人脸数据集
# 从lfw数据中提取特征脸,并对数据进行变换
from sklearn.decomposition import PCA
pca = PCA(n_components=100, whiten=True, random_state=0)
pca.fit_transform(X_people)
X_pca = pca.transform(X_people)
# 用DBSCAN分析人脸数据集
# 应用默认参数的DBSCAN
dbscan = DBSCAN()
labels = dbscan.fit_predict(X_pca)
print("Unique labels: {}".format(np.unique(labels)))
运行结果:
Unique labels: [-1]
所有返回的标签都是-1,因此所有的数据都被DBSCAN标记为噪声。通过改变参数来进行改进这个结果:增加eps或者减小min_samples,从而将更小的组视为簇。
# 改变min_samples:
dbscan = DBSCAN(min_samples=3)
labels = dbscan.fit_predict(X_pca)
print("Unique labels: {}".format(np.unique(labels)))
运行结果:
Unique labels: [-1]
# 即使只考虑由三个点组成的簇,所有的点还是被标记为噪声,增大eps
# 使用更大的eps我们得到了单一簇和噪声点
dbscan = DBSCAN(min_samples=3, eps=15)
labels = dbscan.fit_predict(X_pca)
print("Unique labels: {}".format(np.unique(labels)))
运行结果:
Unique labels: [-1 0]
# 查看多少点是噪声,多少点在簇内
# 计算所有簇中的点数和噪声中的点数。
# bincount不允许负值,所以我们需要加1。 np.bincount()将函数中每一项的词频记录下来
# 结果中的第一个数字对应于噪声点。
print("Number of points per cluster:{}".format(np.bincount(labels + 1)))
运行结果:
Number of points per cluster: [ 32 2031]
# 查看所有的32个噪声点
noise = X_people[labels==-1]
fig, axes = plt.subplots(3, 9, subplot_kw={'xticks': (), 'yticks': ()},figsize=(12, 4))
for image, ax in zip(noise, axes.ravel()):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
plt.show()

被标记为噪声的原因:图像包含奇怪的角度,或者太近或者太宽的剪切,尝试找出奇怪的那一个,被称为异常值检测。
# 想要找到更有趣的簇,需要将eps设置的更小,取值在15和0.5之间
for eps in [1, 3, 5, 7, 9, 11, 13]:
print("\neps={}".format(eps))
dbscan = DBSCAN(eps=eps, min_samples=3)
labels = dbscan.fit_predict(X_pca)
print("Clusters present: {}".format(np.unique(labels)))
print("Cluster sizes: {}".format(np.bincount(labels + 1)))
运行结果:
eps=1
Clusters present: [-1]
Cluster sizes: [2063]
eps=3
Clusters present: [-1]
Cluster sizes: [2063]
eps=5
Clusters present: [-1]
Cluster sizes: [2063]
eps=7
Clusters present: [-1 0 1 2 3 4 5 6 7 8 9 10 11 12]
Cluster sizes: [2004 3 14 7 4 3 3 4 4 3 3 5 3 3]
eps=9
Clusters present: [-1 0 1 2]
Cluster sizes: [1307 750 3 3]
eps=11
Clusters present: [-1 0]
Cluster sizes: [ 413 1650]
eps=13
Clusters present: [-1 0]
Cluster sizes: [ 120 1943]
对于较小的 eps,所有点都被标记为噪声。eps=7 时,我们得到许多噪声点和许多较小的簇。eps=9 时,我们仍得到许多噪声点,但我们得到了一个较大的簇和一些较小的簇。从eps=11 开始,我们仅得到一个较大的簇和噪声。有趣的是,较大的簇从来没有超过一个。最多有一个较大的簇包含大多数点,还有一些较小的簇。这表示数据中没有两类或三类非常不同的人脸图像,而是所有图像或多或少地都与其他图像具有相同的相似度(或不相似度)。eps=7 的结果看起来最有趣,它有许多较小的簇。我们可以通过将 13 个较小的簇中的点全部可视化来深入研究这一聚类。
# eps=7查看13个较小簇中的点
dbscan = DBSCAN(min_samples=3, eps=7)
labels = dbscan.fit_predict(X_pca)
for cluster in range(max(labels) + 1):
mask = labels == cluster
n_images = np.sum(mask)
fig, axes = plt.subplots(1, n_images, figsize=(n_images * 1.5, 4),subplot_kw={'xticks': (), 'yticks': ()})
for image, label, ax in zip(X_people[mask], y_people[mask], axes):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
ax.set_title(people.target_names[label].split()[-1])
plt.show()





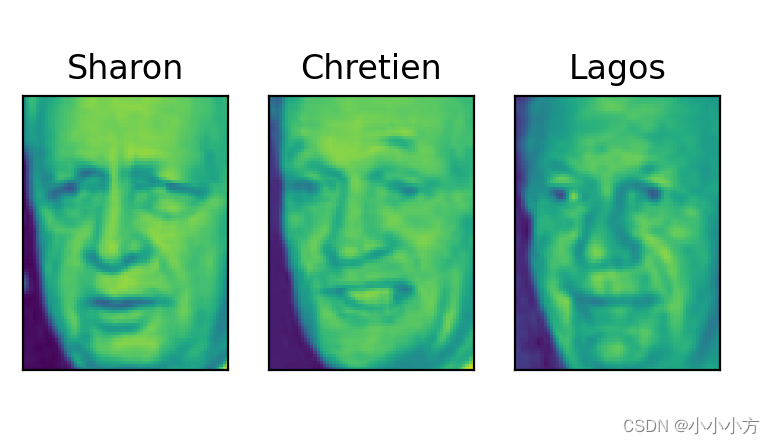



有一些簇对应于数据集中脸非常不同的人,在每个簇中,人脸方向和面部表情也是固定的,有些簇中包含多个人的面孔,但他们的方向和表情相似。
利用DBCSDN无法创建多于一个较大的簇。凝聚聚类和k均值更可能创建均匀大小的簇,但我们需要设置簇的目标个数。可以将簇的数量设置为数据集中的已知人数,虽然无监督聚类算法不太可能完全找到它们。相反我们可以先设置一个比较小的簇的数量。
# 用k均值提取簇
km = KMeans(n_clusters=10, random_state=0)
labels_km = km.fit_predict(X_pca)
print("Cluster sizes k-means:{}".format(np.bincount(labels_km)))
运行结果:
Cluster sizes k-means: [155 175 238 75 358 257 91 219 323 172]
k均值聚类将数据划分为大小相似的簇,大小在75和358之间。通过将簇中心近可视化来进一步分析k均值的结果。由于我们是在PCA生成的表示中进行聚类,需要使用pca.inverse_transform 将簇中心旋转回到原始空间并可视化。
# 簇中心可视化
fig, axes = plt.subplots(2, 5, subplot_kw={'xticks': (), 'yticks': ()},figsize=(12, 4))
for center, ax in zip(km.cluster_centers_, axes.ravel()):
ax.imshow(pca.inverse_transform(center).reshape(image_shape),vmin=0, vmax=1)
plt.show()

k均值找到的簇中心是非常平滑的人脸,因为每个簇中心都是64到386张人脸图像的平均。使用降维PCA表示,可以增加图像的平滑度。对每个簇中心给出了簇中5张典型的图像与5张最不典型的图像。
mglearn.plots.plot_kmeans_faces(km, pca, X_pca,X_people,y_people, people.target_names)
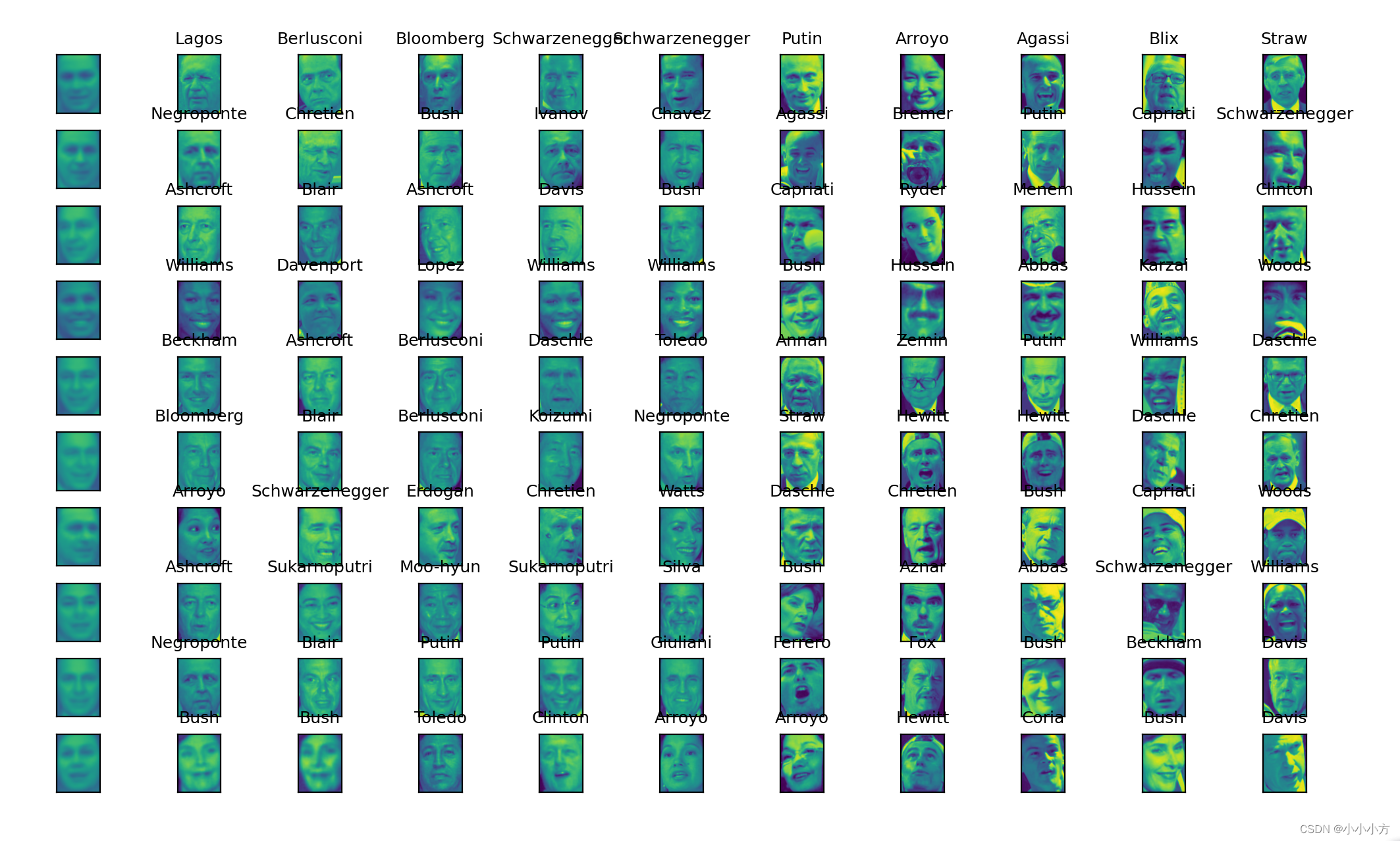
k均值对所有数据点进行划分,不像DBSCAN具有噪声的概念,利用更多数量的簇,算法可以找到更细微的区别,但添加更多的簇会是得人工检查更加困难。
# 用凝聚聚类分析人脸数据集
# 用ward凝聚聚类提取簇
agglomerative = AgglomerativeClustering(n_clusters=10)
labels_agg = agglomerative.fit_predict(X_pca)
print("Cluster sizes agglomerative clustering: {}".format(np.bincount(labels_agg)))
运行结果:
Cluster sizes agglomerative clustering: [169 660 144 329 217 85 18 261 31 149]
凝聚聚类生成的也是大小相近的簇,大小在18和660之间,这比k均值生成的簇更加不均匀。但是比DBSCAN更加均匀。
可以通过计算API来度量凝聚聚类和k均值给出的两种数据划分是否相似
print("ARI: {:.2f}".format(adjusted_rand_score(labels_agg,labels_km)))
运行结果:
ARI: 0.09
labels_agg 和 labels_km 这两种聚类的共同点很少。这并不奇怪,原
因在于以下事实:对于 k 均值,远离簇中心的点似乎没有什么共点。
# 绘制树状图 限制树的深度
linkage_array = ward(X_pca)
# 现在我们为包含簇之间距离的linkage_array绘制树状图
plt.figure(figsize=(20, 5))
dendrogram(linkage_array, p=7, truncate_mode='level', no_labels=True)
plt.xlabel("Sample index")
plt.ylabel("Cluster distance")
plt.show()
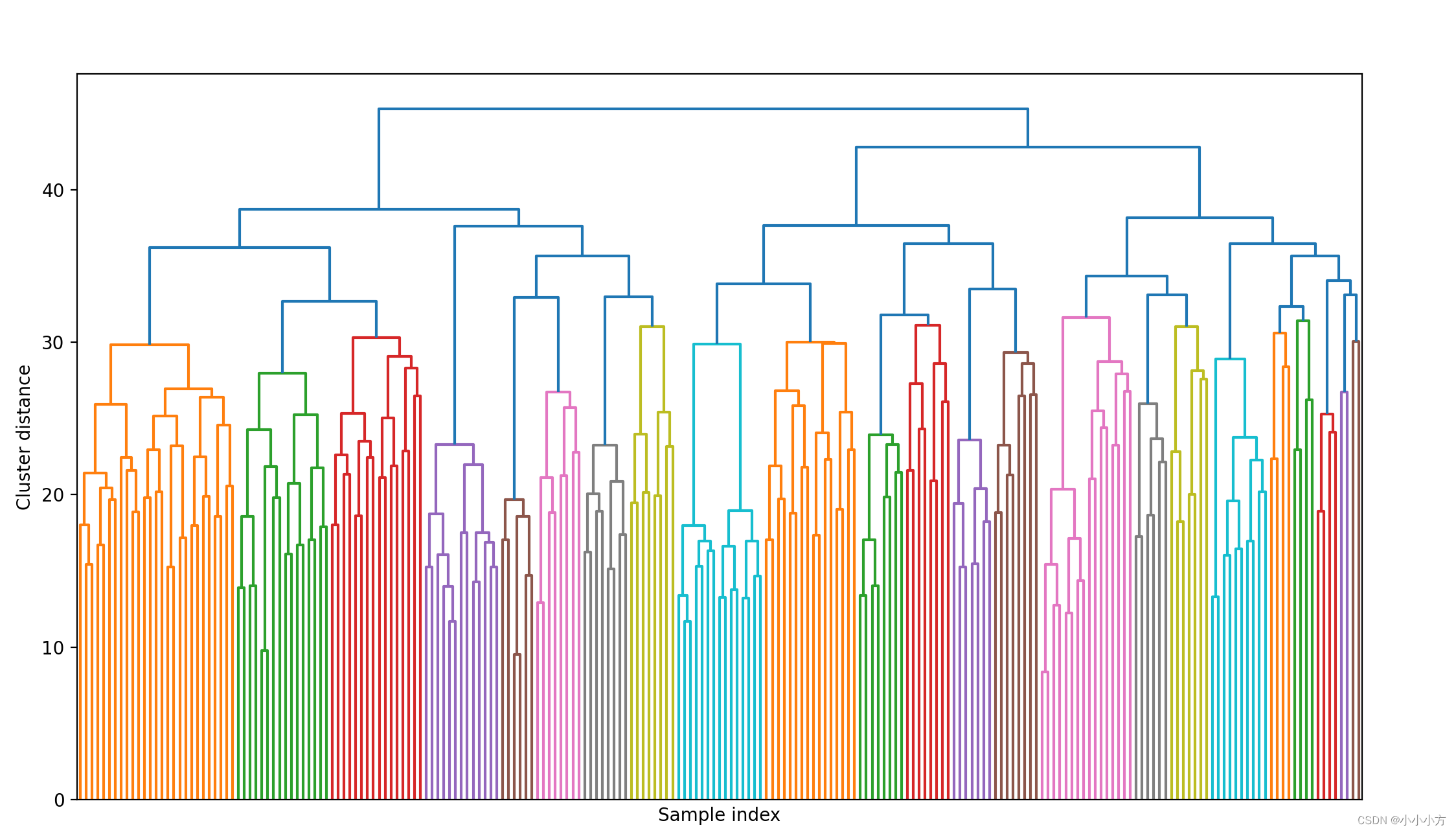
想要创建10个簇,在顶部有10条竖线的位置将树横切,但是对于人脸数据集好想没有非常自然的切割点。有一些分支代表更为不同的组,但似乎没有一个特别合适的簇的数量。这并不奇怪,因为 DBSCAN 的结果是试图将所有的点都聚类在一起。
# 将簇进行可视化
n_clusters = 10
for cluster in range(n_clusters):
mask = labels_agg == cluster
fig, axes = plt.subplots(1, 10, subplot_kw={'xticks': (), 'yticks': ()},figsize=(15, 8))
axes[0].set_ylabel(np.sum(mask))
for image, label, asdf, ax in zip(X_people[mask], y_people[mask],labels_agg[mask], axes):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
ax.set_title(people.target_names[label].split()[-1],fontdict={'fontsize': 9})
plt.show()

 





某些簇好像有语义主题,但许多簇都太大而实际上很难是均匀的,为了得到更均匀簇,我们可以再次运行算法,这次使用40个簇,并挑选出一些有趣的簇。
# 用ward凝聚聚类提取簇
agglomerative = AgglomerativeClustering(n_clusters=40)
labels_agg = agglomerative.fit_predict(X_pca)
print("cluster sizes agglomerative clustering: {}".format(np.bincount(labels_agg)))
n_clusters = 40
for cluster in [10, 13, 19, 22, 36]: # 手动挑选“有趣的”簇
mask = labels_agg == cluster
fig, axes = plt.subplots(1, 15, subplot_kw={'xticks': (), 'yticks': ()},figsize=(15, 8))
cluster_size = np.sum(mask)
axes[0].set_ylabel("#{}: {}".format(cluster, cluster_size))
for image, label, asdf, ax in zip(X_people[mask], y_people[mask],labels_agg[mask], axes):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
ax.set_title(people.target_names[label].split()[-1],fontdict={'fontsize': 9})
for i in range(cluster_size, 15):
axes[i].set_visible(False)
plt.show()
运行结果:
cluster sizes agglomerative clustering: [ 43 120 100 194 56 58 127 22 6 37 65 49 84 18 168 44 47 31
78 30 166 20 57 14 11 29 23 5 8 84 67 30 57 16 22 12
29 2 26 8]





这里聚类挑选出的似乎是“深色皮肤且微笑”“有领子的衬衫”“微笑的女性”“萨达姆”和“高额头”
聚类方法小结
聚类的应用与评估是一个非常定性的过程,通常在数据分析的探索阶段。k均值和凝聚聚类允许指定想要的簇的数量,DBSCANA允许使用eps参数定义接近程度,从而干扰间接的影响簇的大小。
每种算法的优点不同,k均值可以用簇的平均值来表示簇,他还可以看作一种分解方法,每个数据点都由其簇中心表示。DBSCAN可以检测到没有分配任何簇的噪声点,还可以帮助自动判读簇的数量,他允许簇具有复杂的形状。有时会生成大小差别很大的簇,这可能是缺点但也可能是优点。凝聚聚类可以提供数据的可能划分的整个层次结构,可以通过树状图查看。
总结
分解、流形学习和聚类都是加深数据理解的重要工具,在没有监督信息的情况下,也是理解数据的仅有的方法。即使是在监督学习中,探索性工具对于更好地理解数据性质也很重要。通常来说,很难量化无监督算法的有用性,但这不应该妨碍你使用它们来深入理解数据。
估计器接口
scikit-learn 中的所有算法——无论是预处理、监督学习还是无监督学习算法——都被实现为类。这些类在 scikit-learn中叫作估计器。为了应用算法,你首先需要将特定类的对象实例化。
在构建模型对象时,你应该设置模型的所有参数,这些参数包括正则化、复杂度控制、要找的簇的数量等等。所有的估计器都有fit方法,用于构建模型。fit方法要求第一个参数总是数据X,用一个NumPy数组或SciPy稀疏矩阵表示,其中每一行代表一个数据点。数据X总是被假定为一个具有连续值的NumPy数组或SciPy稀疏矩阵。监督算法还需要有一个y参数,它是一维 NumPy 数组,包含回归或分类的目标值(即已知的输出标签或响应)。
在 scikit-learn 中,应用学到的模型主要有两种方法。要想创建一个新输出形式(比如 y)的预测,可以用 predict 方法。要想创建输入数据 X 的一种新表示,可以用transform 方法。

所有监督模型都有 score(X_test, y_test) 方法,可以评估模型。在表 3-1 中,X_train 和 y_train 指的是训练数据和训练标签,而 X_test 和 y_test 指的是测试数据和测试标签。





















 1192
1192











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








