说明
在之前的几篇博文[1][2][3][4]中,我分别对遗传算法和粒子群算法做了一些探讨。在找资料的过程中读到参考资料[5]时,发现我们其实可以把多种优化算法(不限于本文将要讨论的粒子群和遗传算法之间)结合起来使用,当然,结合使用的目标自然是希望能够吸收多种算法的优势以达到更高的优化效果(比如增加收敛速度、让优化结果更容易朝向全局最优解)。不过本博文的目的在于建立算法混合使用时的思路和框架,(要使得混合下的算法效果由于各算法单独使用下的效果,还需要进行参数优化)
本文以Rastrigin测试函数为例,试图探讨粒子群和遗传两种算法的对比及其混合使用。需要说明的是:本文关于两算法的混合使用只能算是一个简单(浅显)的尝试,后续有机会我再更新丰富,希望本文的内容能给对优化算法感兴趣的读者一点参考。
Blog
2024.4.18 博文第一次撰写
目录
一、粒子群算法&遗传算法概述
我在博文[1]: 粒子群算法及基于该算法的典型问题求解实践-CSDN博客 和[3]: 粒子群算法及基于该算法的典型问题求解实践-CSDN博客 中已经做了详细的说明,这里不再赘述。
二、Rastrigin测试函数说明
在[1]中,我使用了Griewank测试函数来测试粒子群算法。用于优化算法测试的函数有很多,这些函数的共同点在于有诸多的极值点(凹凸不平),可以用来检测算法是否收敛、以及是否容易陷入局部最优解。本博文选择Rastrigin函数进行算法的测试。
首先对该函数做简单介绍:Rastrigin函数的通用表达式为:
![]() (2-1)
(2-1)
式中,A为常数,一般取10;d为x的维度。在二维下,该式可化为:
![]() (2-2)
(2-2)
取x和y的范围都是[-10 10],画出该函数的二维曲面图如下:

图2.1 二维Griewank函数对应的曲面
本博文将分别使用粒子群算法、遗传算法、以及粒子群与遗传算法的串行混合方法在x和y的限定范围内[-10 10]寻找二维Griewank函数最小值。注:Griewank函数的最小值位于[0 0]处。
公式(2-2)便可作为三种方法的适应度函数,Z值越小表示适应度越好。
三、仿真方案&参数
基于粒子群算法、遗传算法的优化问题求解思路和流程也已经在[1]和[3]中给出,这里不再重复。关于两种算法的混合使用,有所谓的并行混合、串行混合以及嵌入式混合(不过似乎网上相关的介绍比较少),在参考资料[5]中,作者给出了所谓的嵌入式混合的处理流程:

图3.1 嵌入式混合算法流程图
该流程图给出的整体处理流程其实很好理解。图中,越界处理及之前的操作是完整的粒子群算法处理流程,这里所谓的嵌入式混合是将遗传算法中的交叉和变异两个操作放到了粒子群算法处理流程之后:也就是对粒子群算法获得的结果再做一些变化。我对所谓的并行、串行以及嵌入式混合做了些简单的思索后,觉得前面给出的嵌入式似乎和我理解的串行很像(只是该流程中丢掉了部分处理:比如遗传算法中的复制操作、以及在做交叉操作之前是否应该更新一次适应度?)本文后续仿真中的串行混合处理流程如下图所示:
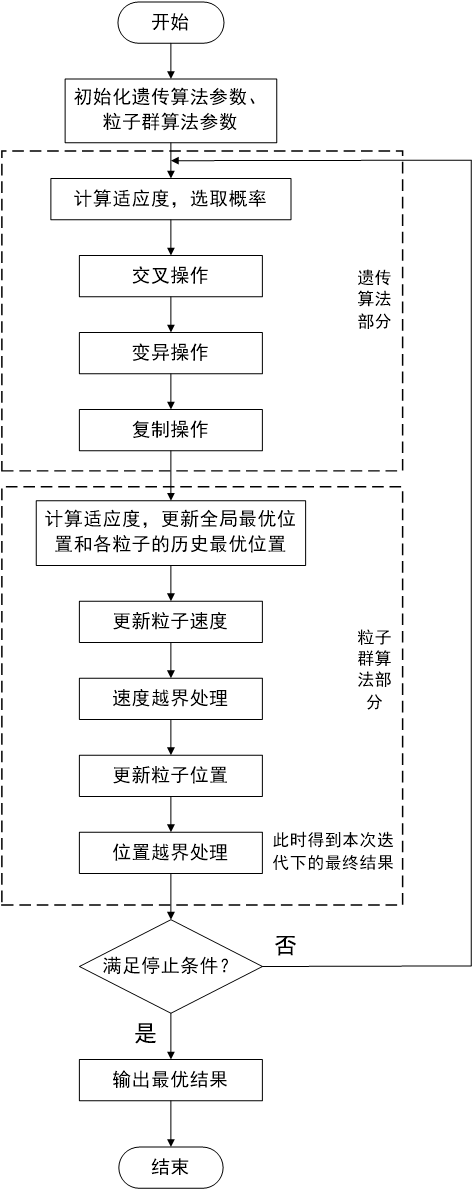
图3.2 串行混合算法流程图
图3.2提供的算法流程其实就是将两种算法进行叠加(所谓的串行应该也就是这个意思?),我这里将遗传算法放在了前面,读者也可以试试将粒子群算法放在前面。
本博文的仿真所用参数列表如下:
表3.1 算法参数列表
| 参数 | 值 | |
| 粒子群算法参数 | 粒子数量 | 15 |
| 粒子维度 | 2(对应二维坐标值) | |
| 粒子位置范围 | [-10 10](两个维度一样) | |
| 粒子速度范围 | [-1 1] | |
| 惯性权重 | [0.4 1.2],随迭代线性递减。 | |
| 个体学习因子 | 0.5(两个维度一样) | |
| 群体学习因子 | 0.8(两个维度一样) | |
| 迭代终止条件 | 迭代100次 | |
| 遗传算法参数 | 染色体数 | 15 |
| 单条染色体基因数 | 2 | |
| 基因数值范围 | [-10 10] | |
| 从上一代直接复制染色体条数 | 3 | |
| 变异率 | 0.1 | |
| 选择父代染色体的方法 | 轮盘赌算法 | |
| 交叉方法 | 父母染色体各拿一个基因 | |
| 变异方法 | 随机选择基因,并在限定范围内随机突变 |
注:后续三种仿真下,使用相同的初始化解(初始化的染色体和初始化的粒子一样)。 诚然,因为两种算法各自涉及到的参数很多,这些参数都会影响算法的收敛效果,所以很难简单对比两种算法的优劣。
四、仿真结果与探讨
在前文的基础上,本章分别给出:遗传算法、粒子群算法、以及两算法串行混合下的仿真结果:

图4.1 单独使用粒子群算法时,不同迭代次数下各粒子适应度值的变化
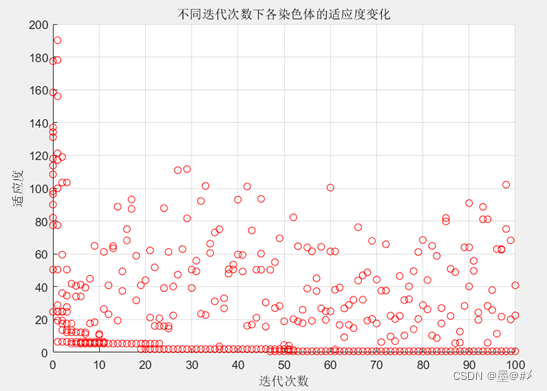
图4.2 单独使用遗传算法时,不同迭代次数下各染色体适应度值的变化

图4.3 串行混合算法下,不同迭代次数下各个解的适应度值变化
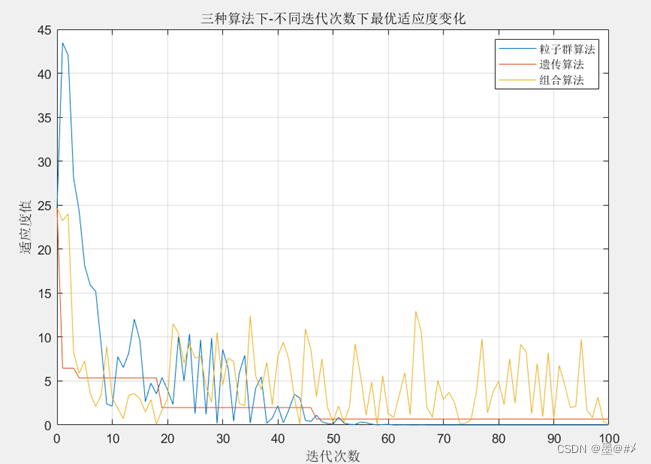
图4.4 三种算法下历次迭代下最优适应度值的变化
从仿真结果来看,三种方法都有收敛的趋势,其中粒子群算法的效果更好(当然,这与参数设置有关)。三种方法的结果都验证了代码的可行性,我在串行混合算法中使用的参数是沿用两种算法单独使用时的参数,所以参数可能并不是最优的,感兴趣的读者可以在我提供的代码上做优化。
五、总结
本博文基于Rastrigin测试函数探讨粒子群算法和遗传算法的混合使用,分别给出了嵌入式混合以及串行混合下的算法流程,并对串行混合进行了仿真验证,仿真的结果验证了算法流程的可行性。
本文提供的仿真结果下,串行混合的效果不如粒子群或遗传算法单独使用时的效果,但优化混合算法参数以使得该算法的结果优于单独使用某种算法时的效果不是本文的核心目标(因为算法涉及的参数很多)。本博文的目的在于建立算法混合使用时的思路和框架。
六、参考资料
[1] 遗传算法及基于该算法的典型问题的求解实践-CSDN博客
[3] 粒子群算法及基于该算法的典型问题求解实践-CSDN博客
[4] 基于粒子群算法的波束形成优化 – 仿真实践-CSDN博客
[5] 超详细 | 遗传-粒子群自适应优化算法及其实现(Matlab) CSDN博客








 本文探讨了如何将粒子群算法和遗传算法结合使用于Rastrigin测试函数的优化,介绍了串行混合方法的流程,并展示了单独与混合算法的仿真结果。着重于构建混合算法的思路框架,而非优化参数的详细比较。
本文探讨了如何将粒子群算法和遗传算法结合使用于Rastrigin测试函数的优化,介绍了串行混合方法的流程,并展示了单独与混合算法的仿真结果。着重于构建混合算法的思路框架,而非优化参数的详细比较。

















 4147
4147

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










