Bioinformatics: Assembling Genomes (week 1-2)
本文为Coursera课程 Assembling Genomes and Sequencing Antibiotics (Bioinformatics II) 笔记。
作者:ybw
Introduction
Motivation
To sequence a genome:
1. 目前缺少读取完整DNA链的技术;
2. 采用将DNA链打碎成reads并识别的方法(荧光标记法)来获取DNA信息;
3. 如何将reads拼接成原来的DNA。
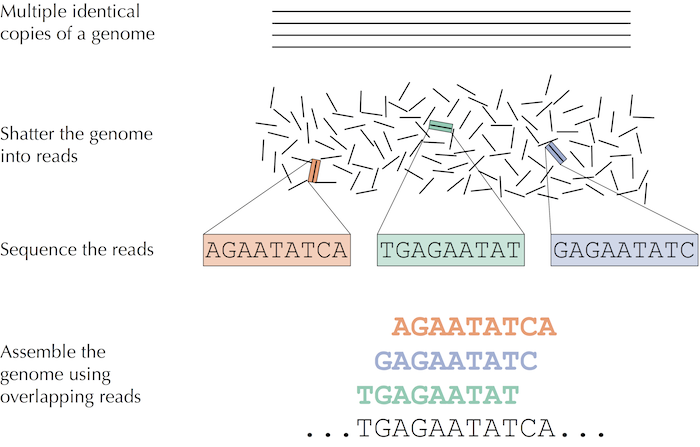
具体过程如图1所示, 将包含无数个相同DNA的组织样本或血样使用生物学方法打碎成短的fragments,并识别这些fragments,从而得到reads。
图1:
Problems
- DNA为双链结构(double-stranded),无法分辨每个read属于哪个链;
- 目前识别reads的方法并不完美,其中的氨基酸和DNA链中可能并非完全相同;
- DNA中的某些区域可能没有被任何reads覆盖,导致无法完整地重构基因(genome)。
Terminologies
- read: short DNA fragments called reads。
- k-mer:
- strand: DNA单链
The ideal model - From Hamilton to de Bruijn
The ideal model
首先在理想条件研究问题。假设理想情况如下:
* 所有的reads来自同一个strand, 且没有错误;
* Perfect coverage - 任何属于该strand的k-mer都会产生reads。
From a k-mers to a graph
为了在k-mers之间建立联系,使用prefix和suffix来分别表示一个k-mer的前k-1个核苷酸和后k-1个核苷酸(nucleotide)。例如Prefix (TAA) = TA, Suffix (TAA) = AA。
为了建图,用每个k-mer来表示图中的一个node,当 prefix(kmer1)=suffix(kmer2) ,则添加一条从 kmer1 到 kmer2 的有向边。
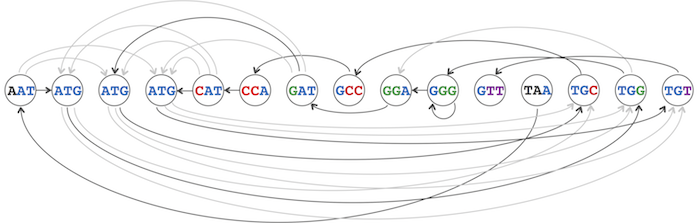
由此所建立的图称为overlap graph,记为 Overlap(Patterns) 。图2所示的是由DNA链TAATGCCATGGGATGTT所产生的k-mers组成的overlap graph,图中实线表示的路径代表基因序列“TAATGCCATGGGATGTT”。
图2-The genome path spelling out TAATGCCATGGGATGTT, highlighted in the overlap graph:
Using Grpahs to assemble - Hamiltonian Path
在overlap graph中,edges代表可能的DNA序列,例如图2中的边(TAA) -> (AAT)表示TAAT。如果可以在overlap graph中找到一个路径(path),该路径经过所有节点且只经过一次,则该路径就可以表示该图可产生的一个genome。根据定义,这条路径即是哈密顿路径。
A path in a graph visiting every node once is called a Hamiltonian path.
由于overlap graph中的Hamiltonian path不止一条,而且Hamiltonian path是NP-Complet问题,因此当问题规模太大时就无法处理了。为了解决该问题,研究人员发现了另一种方法 - de Bruijn Graph。
另外,人类基因组计划好像是通过Hamiltonian path来计算的,因为当时de Bruijn Graph并没有被发现。
From Hamiltonian Path to Eulerian Path
k-universal string
在引入de Bruijn Graph之前,我们首先看一下荷兰数学家Nicolaas de Bruijn的一项研究。1946年,de Bruijn对一个纯理论问题产生了兴趣,该问题表述如下。
A binary string is a string composed only of 0’s and 1’s; a binary string is k-universal if it contains every binary k-mer exactly once.
For example, 0001110100 is a 3-universal string, as it contains each of the eight binary 3-mers (000, 001, 011, 111, 110, 101, 010, and 100) exactly once.
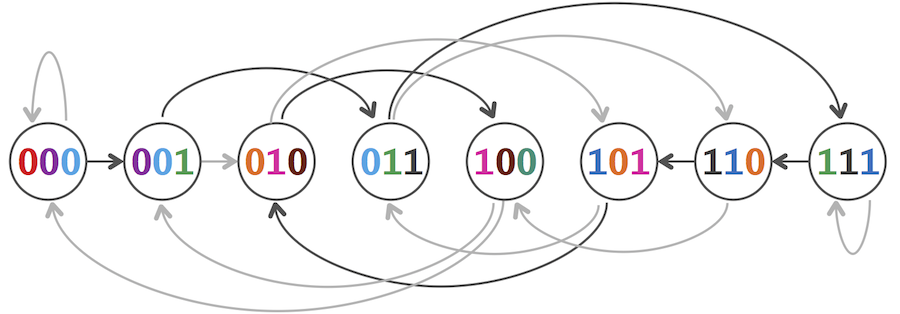
当图中的节点为所有的binary k-mers时,解决k-universal string问题和解决String Reconstruction Problem问题是等价的。我们已经知道使用哈密顿路径可以解决String Reconstruction Problem,那么哈密顿路径也一定可以解决该问题。图3为一个例子。
图3:A Hamiltonian path in the overlap graph of all binary 3-mers.
然而,de Bruijn当时并没有使用哈密顿路径来解决该问题,而是用了一个不那么直观的方法来表示k-mer:
使用边而非节点来表示k-mers。
de Bruijn Graph
将该思想引入String Reconstruction Problem,就可得到de Bruijn Graph。在String Reconstruction Problem问题中,de Bruijn graph的边根据输入的k-mers来建立,而边两边的节点分别为该边所表示的k-mer的prefix和suffix。问题表述如下:
DeBruijn Graph from k-mers Problem: Construct the de Bruijn graph from a set of k-mers.
Input: A collection of k-mers Patterns.
Output: The adjacency list of the de Bruijn graph DeBruijn(Patterns).
解决步骤包括:
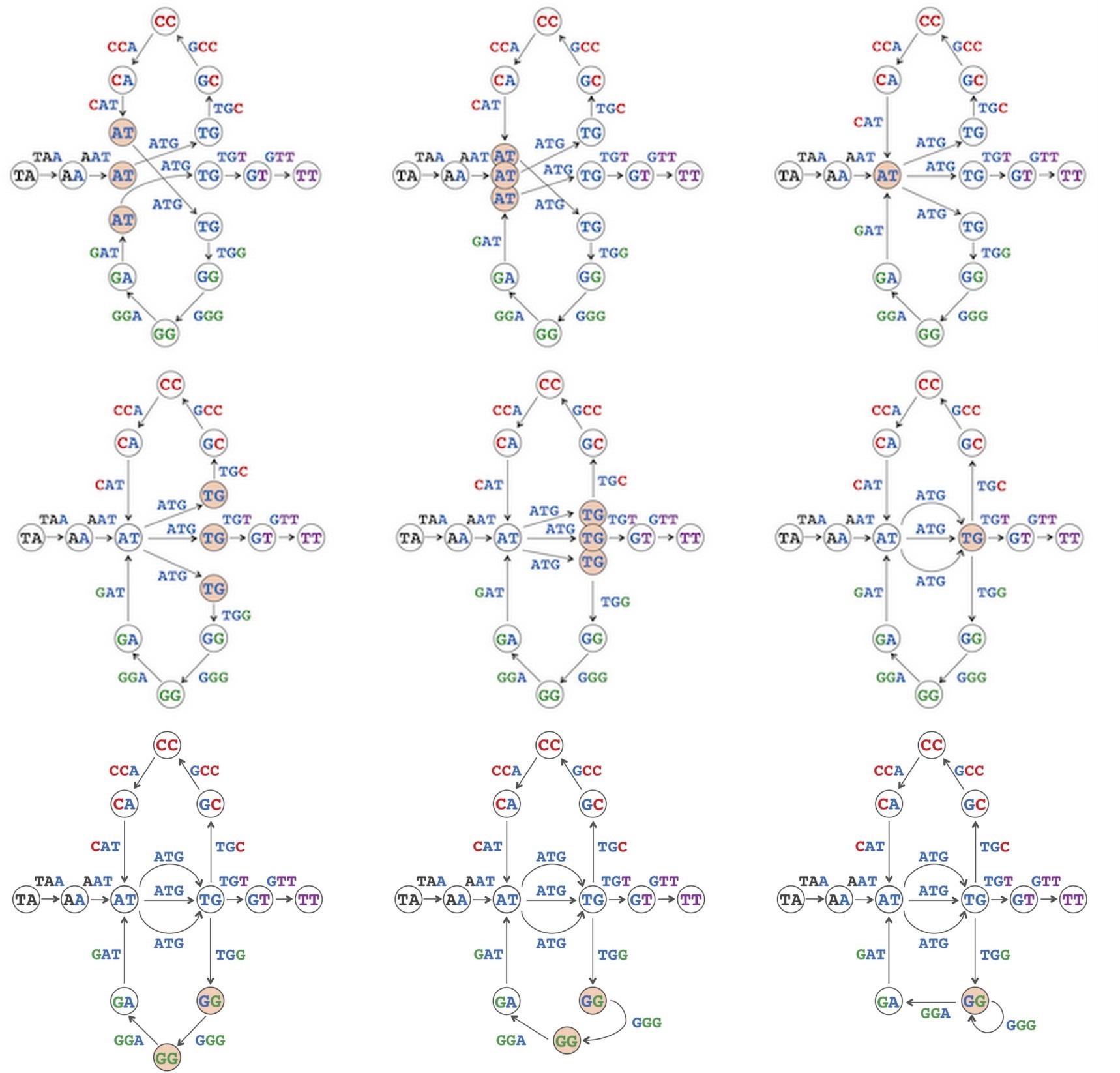
- 使用有向边来表示k-mer,则边的起点为该k-mer的prefix, 终点为suffix。如图4;
- 将相同节点合并,如图5;
图4:Genome TAATGCCATGGGATGTT represented as a path with edges (rather than nodes) labeled by 3-mers and nodes labeled by 2-mers.
图5:Gluing nodes
Eulerian Paths
尽管我们将de Bruijn graph中相同的nodes进行了合并,但由于图中的边并没有改变,因此图中所携带的有关genome的信息并没有发生变化。与之前类似,因为de Bruijn graph中每个边都代表一个k-mer,只需找到一个路径,经过图中所有的边且每个边只经过一次,即可解决String Reconstruction Problem问题。即,solving the String Reconstruction Problem reduces to finding an Eulerian Path in the de Bruijn graph.
注:
* 该图中的边由给出的k-mers来创建,在图建好之后,不能根据节点信息随意添加新的边;
* 图中的欧拉路径可能不止一条。
Eulerian Cycle and Eulerian Path
那么,如何从图中找到一条欧拉路径,这节将给出具体算法。
Existence
欧拉路径问题可以追述到哥尼斯堡七桥问题(Bridges of Königsberg Problem):Is it possible to set out from my house, cross each bridge exactly once, and return home? 哥尼斯堡的地图可以简化至图6。
图6:The graph Königsberg.
该问题等价于: 该图中是否存在欧拉回路(Eulerian Cycle)。
Eulerian cycle: A cycle that traverses each edge of a graph exactly once is called an Eulerian cycle.
对于无向图:
A graph has an Eulerian circuit if and only if it is connected and every node has ‘even degree’.
对有向图,为了欧拉回路是否存在,引入balance概念:
当一个节点的出度和入度相等,称该节点为balanced;
当一个图中所有的节点都为balanced,称该图为balanced。
(A node v is balanced if in(v)=out(v) , and a graph is balanced if all its nodes are balanced. )
那么是否balanced graphs一定存在欧拉回路呢? 答案是否定的,因为该图未必是强联通的(strongly connected)。
Euler’s Theorem: Every balanced, strongly connected directed graph is Eulerian.
(An Eulerian graph is a graph containing an Eulerian cycle.)
对于balanced and strongly connected directed graphs,如何找到an Eulerian cycle。下面给出算法。
Code
Pseudo-Code:
Hierholzer’s algorithm
find_cycle(node i)
if node i has no neighbors
cycle[pos] = node i
pos++
else
while(node i has neighbors)
pick a neighbor j of node i
delete_edges(node j, node <







 本文介绍了基因组组装的基础知识,包括基因组组装的动机、面临的问题和术语。文章深入探讨了理想模型,从Hamilton路径到Eulerian路径,以及de Bruijn图在基因组组装中的应用。此外,还讨论了Eulerian路径的实现、欧拉回路和解决字符串重构问题的方法,以及处理读对和错误读取的高级方法。
本文介绍了基因组组装的基础知识,包括基因组组装的动机、面临的问题和术语。文章深入探讨了理想模型,从Hamilton路径到Eulerian路径,以及de Bruijn图在基因组组装中的应用。此外,还讨论了Eulerian路径的实现、欧拉回路和解决字符串重构问题的方法,以及处理读对和错误读取的高级方法。

 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 332
332

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








