首先对不负责任的无脑转载行为表示谴责!
以下内容均为本人亲测可行。
原文地址
http://ngs-assist.com/forum.php?mod=viewthread&tid=46
1. 简述
OrthoMCL (http://orthomcl.org/orthomcl/)主要用来找直系同源基因以及旁系同源基因。它主要在比较完整的基因组之间找直系同源基因。OrthoMCL的使用主要13步,可以参考doc/OrthoMCLEngine/Main/UserGuide.txt。为了方便运行OrthoMCL,可以建立一个工作目录“my_orthomcl_dir”。
2. 软件安装
下载地址:
http://orthomcl.org/common/downloads/software/v2.0/orthomclSoftware-v2.0.9.tar.gz
3. 软件配置步骤(转载+再编辑)
1. 配置OrthoMCL程序
建议没接触过mysql的先查阅一下相关教程。
将orthomcl.config.template拷贝到你工作目录下(my_orthomcl_dir)。然后根据所建的mysql数据库名,用户名,密码。修改该文件。
例子如下:
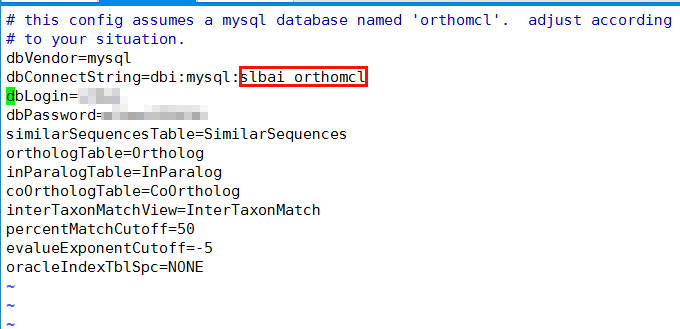
红框处填写自己创建的数据库名称。
主要有两个阈值参数:
percentMatchCutoff:
blastsimilarities with percent match less than this value are ignored.
evalueExponentCutoff:
blastsimilarities with evalue Exponents greather than this value are ignored.
由于我对此参数了解不深,所以单纯copy,不做解释。
2. 利用orthomclInstallSchema命令对Oracle或者Mysql数据库进行配置
Usage:
orthomclInstallSchema config_file sql_log_file table_suffix
比如在my_orthomcl_dir目录下运行:
../orthomclSoftware-v2.0.9/bin/orthomclInstallSchema orthomcl.config.template orthomcl.config.log
3. 利用orthomclAdjustFasta命令把输入文件转换为orthomcl所需的文件格式
Usage:
orthomclAdjustFasta taxon_code fasta_file id_field
这里从Ensembl下载了Ustilago maydis和Saccharomyces cerevisiae两个物种的蛋白质组文件。
../orthomclSoftware-v2.0.9/bin/orthomclAdjustFasta Ust Ustilago_maydis.fasta 1
../orthomclSoftware-v2.0.9/bin/orthomclAdjustFasta Sac Saccharomyces_cerevisiae1
就会生成两个文件:Ust.fasta 和Sac.fasta。这可以方便运行my_orthomcl_dir/compliantFasta/。
pwd
#/home/slbai/mysoftware/orthomcl/my_orthomcl_dir
../orthomclSoftware-v2.0.9/bin/orthomclAdjustFasta Ust ustilago_maydis_1_proteins.fasta 1
../orthomclSoftware-v2.0.9/bin/orthomclAdjustFasta Sac Saccharomyces_cerevisiae.R64-1-1.pep.all.fa 1
4. 利用orthomclFilterFasta命令过滤掉差的序列文件
Usage:
orthomclFilterFasta input_dirmin_length max_percent_stops [good_proteins_file poor_proteins_file]
例如运行:
“../orthomclSoftware-v2.0.9/bin/orthomclFilterFasta ./compliantFasta/”。
mkdir compliantFasta
mv Ust.fasta compliantFasta
mv Sac.fasta compliantFasta
../orthomclSoftware-v2.0.9/bin/orthomclFilterFasta compliantFasta/ 10 20
5. Blast比对
对上一步得到的goodProteins.fasta进行多对多的比对。推荐使用NCBIBlast.
我这里使用是ncbi-blast-2.2.28+。
运行命令:
“~/Universal_softwore_src/ncbi-blast-2.2.28+/bin/makeblastdb-in good_proteins.fasta -dbtype prot -out good_proteins.fasta”
“~/Universal_softwore_src/ncbi-blast-2.2.28+/bin/blastp-db goodProteins.fasta -querygoodProteins.fasta -outfmt 7 –out goodProteins_blastp.out ”。
然后生成tab delimited格式的输出文件goodProteins_blastp.out。生成的比对文件最好是tab文件格式。不同的版本的输出格式参数也许不一样。该软件就是-outfmt 7。
得到该文件之后需进一步处理之后才能被后面的步骤所使用(只把hits行挑选出来,注释信息丢掉)
可以运行如下命令得到:
“grep -v -P”^#” goodProteins_blastp.out > goodProteins_v1_blastp.out”。
../../ncbi-blast-2.3.0+/bin/makeblastdb -in goodProteins.fasta -dbtype prot -out goodProteins.fasta
../../ncbi-blast-2.3.0+/bin/blastp -db goodProteins.fasta -query goodProteins.fasta -outfmt 7 -out goodProteins_blastp.fasta
以上这一步运行时间较长,建议挂起(nohup)运行后出去溜圈。
grep -v -P "^#" goodProteins_blastp.fasta > goodProteins_v1_blastp.out
6. 利用orthomclBlastParser命令将上一步得到的blast比对结果进行解析,默认阈值为e-value:1e-5 ;Coverage:50%
Usage:
orthomclBlastParser blast_file fasta_files_dir
运行命令:
” ../orthomclSoftware-v2.0.9/bin/orthomclBlastParser goodProteins_v1_blastp.out ./compliantFasta”
运行完之后生成similarSequences.txt文件。
../orthomclSoftware-v2.0.9/bin/orthomclBlastParser goodProteins_v1_blastp.out ./compliantFasta > similarSequences.txt
7. 利用orthomclLoadBlast命令将blast结果导入到mysql数据库中
Usage:
orthomclLoadBlast config_file similar_seqs_file
运行命令如下:
“../orthomclSoftware-v2.0.9/bin/orthomclLoadBlast orthomcl.config.template
similarSequences.txt”
../orthomclSoftware-v2.0.9/bin/orthomclLoadBlast orthomcl.config.template similarSequences.txt
8. 利用” orthomclPairs”对数据库中的SimilarSequence表中数据,进行pairs的运算
Usage:
orthomclPairs config_file log_file cleanup=[yes|no|only|all]<\startAfter=TAG>
运行命令如下:
“../orthomclSoftware-v2.0.9/bin/orthomclPairs orthomcl.config.template pairs.log cleanup=yes ”
默认情况下,在mysql中生成三个表: PotentialOrthologs,PotentialInParalogs, PotentialCoOrthologs。
注明:我运行过程中没有生成‘PotentialOrthologs,PotentialInParalogs, PotentialCoOrthologs’这三个边,但后续步骤运行仍正常。
../orthomclSoftware-v2.0.9/bin/orthomclPairs orthomcl.config.template pairs.log cleanup=yes
按照示例步骤给的代码获得文件‘pairs.log’,但是这个文件好像对后续步骤没什么作用。
9. 利用命令orthomclDumpPairsFiles对数据库中的pairs表进行处理
Usage:
orthomclDumpPairsFiles config_file
运行命令如下:
“../orthomclSoftware-v2.0.9/bin/orthomclDumpPairsFiles paris.log”
../orthomclSoftware-v2.0.9/bin/orthomclDumpPairsFiles orthomcl.config.template
上一步所获得的’pairs.log’文件在该步骤不能成功运行,替换成‘orthomcl.config.template’即可。
生成mcllnput文件和pairs目录。这个目录包含三个文件:
ortholog.txt, coortholog.txt, inparalog.txt。
每一个文件有三列: proteinA, protein B, their normalized score (See the Orthomcl Algorithm Document)。
10. 利用mcl程序把上一步的结果进行聚类
运行命令如下:
mcl ./mclInput –abc-I 1.5 –o ./mclOutput 具体参数可以参考mcl文档。
mcl ./mclInput --abc -I 1.5 -o ./mclOutput
此步骤获得的out.mcl即为最终文件
11. 利用orthomclMclToGroups命令将mcl的输出结果转换为groups.txt
Usage:
orthomclMclToGroups my_prefix 1 < mclOutput > groups.txt
groups.txt就是最终的结果文件。文件中的每一行代表可能存在的蛋白质家族。
这一步我还没有运行成功,日后再说。不过上一步已经获得目标文件,就是没有为每个分类命名而已。






















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








