import numpy as np
from scipy.integrate import odeint
import matplotlib.pyplot as plt
# 设置Matplotlib以支持中文显示
plt.rcParams['font.sans-serif'] = ['SimHei'] # 设置中文字体为黑体,确保支持中文
plt.rcParams['axes.unicode_minus'] = False # 正确显示负号
# SIR模型参数
N = 10099 # 总人口
I0 = 1 # 初始感染者人数
R0 = 2 # 初始康复者人数
S0 = N - I0 - R0 # 初始易感者人数
beta = 0.2 # 感染率
gamma = 0.1 # 康复率
# SIR模型的微分方程
def deriv(y, t, N, beta, gamma):
S, I, R = y
dSdt = -beta * S * I / N
dIdt = beta * S * I / N - gamma * I
dRdt = gamma * I
return dSdt, dIdt, dRdt
# 初始条件向量
y0 = S0, I0, R0
# 时间点(以天为单位)
t = np.linspace(0, 160, 160)
# 集成SIR方程
ret = odeint(deriv, y0, t, args=(N, beta, gamma))
S, I, R = ret.T
# 绘制结果
fig, ax = plt.subplots()
ax.plot(t, S / 1000, 'b', alpha=0.5, lw=2, label='易感者(千人)')
ax.plot(t, I / 1000, 'r', alpha=0.5, lw=2, label='感染者(千人)')
ax.plot(t, R / 1000, 'g', alpha=0.5, lw=2, label='康复者(千人)')
ax.set_xlabel('时间 /天')
ax.set_ylabel('人数(千人)')
ax.set_title('SIR模型模拟结果')
ax.tick_params(axis='x', labelsize=10, labelcolor='b')
ax.tick_params(axis='y', labelsize=10, labelcolor='b')
ax.yaxis.set_tick_params(length=0)
ax.xaxis.set_tick_params(length=0)
ax.grid(True, which='major', c='w', lw=2, ls='-')
ax.legend()
for spine in ('top', 'right', 'bottom', 'left'):
ax.spines[spine].set_visible(False)
plt.show()这只是一个基于Python实现的简单的传染病模型作为一个示例。
实际过程中需要考虑的因素是很多的,人群的流动性、气候环境、地理位置、人群的康复等各种条件都需要考虑。
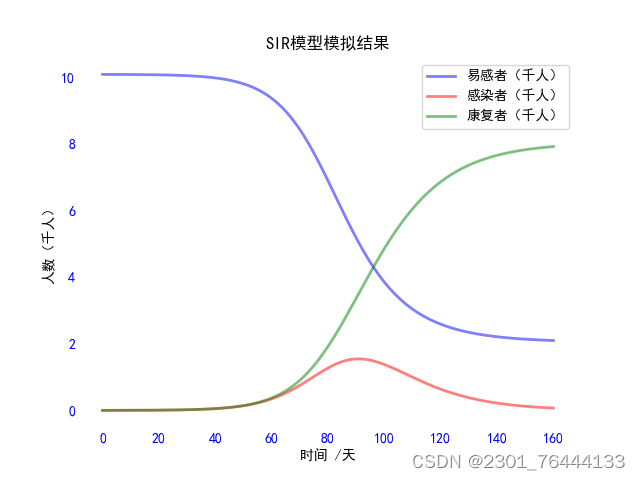
SIR模型模拟结果








 该文章展示了如何使用Python中的numpy、scipy和matplotlib库来实现SIR模型(Susceptible-Infected-Recovered模型),模拟传染病的传播过程,并对结果进行可视化。文章强调了在实际应用中考虑的多因素,如人群流动性、环境和康复情况。
该文章展示了如何使用Python中的numpy、scipy和matplotlib库来实现SIR模型(Susceptible-Infected-Recovered模型),模拟传染病的传播过程,并对结果进行可视化。文章强调了在实际应用中考虑的多因素,如人群流动性、环境和康复情况。














 230
230











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








