
摘要
理解玉米(Zea mays)如何应对寒冷胁迫对促进寒冷耐受品种的育种工作至关重要。尽管基因组范围关联研究(GWAS)方法已广泛应用于探索与玉米寒冷耐性相关的有利自然等位基因,但成功识别出对玉米寒冷耐性有贡献的候选基因的研究仍较少。本研究利用来自不同种质资源的多样化玉米自交系面板,对玉米在寒冷胁迫下真叶相对受伤面积的变异进行了GWAS分析,该特征与玉米寒冷耐性高度相关。我们鉴定了HSF21基因,该基因编码一种B类热休克转录因子(HSF),在种苗和萌发阶段积极调控寒冷耐性。寒冷耐性HSF21Hap1等位基因启动子中的自然变异,通过抑制基本亮氨酸拉链(bZIP68)转录因子的结合,增强了寒冷胁迫下HSF21的表达,而bZIP68是寒冷耐性的负调控因子。通过整合转录组深度测序、DNA亲和纯化测序和靶向脂质组分析,我们揭示了HSF21在调控脂质代谢稳态方面的作用,从而调节玉米的寒冷耐性。此外,我们还发现HSF21能够在不影响产量的情况下提高玉米的寒冷耐性。综上所述,本研究确立了HSF21作为增强玉米寒冷耐性的关键调控因子,为寒冷耐性玉米品种的育种提供了宝贵的遗传资源。
关键词:热休克因子HSF21,寒冷耐性,自然变异,bZIP68,脂质代谢,玉米
引言
玉米(Zea mays)由于其热带起源,本身对低温较为敏感(Matsuoka et al., 2002)。低温对玉米的生长和发育有显著影响,影响其种植范围、播种期和生长周期(Allen and Ort, 2001)。因此,迫切需要具有较强寒冷耐性的玉米品种。在过去的二十年里,许多研究致力于通过定量性状位点分析和基因组范围关联研究(GWAS)来识别影响寒冷相关性状的基因(Jompuk et al., 2005; Presterl et al., 2007; Leipner et al., 2008; Strigens et al., 2013; Yi et al., 2021; Zhang et al., 2021)。然而,许多提议的候选基因或其致病变异尚未得到充分验证和明确澄清。脂质重塑是植物应对寒冷胁迫的策略之一(Miquel et al., 1993; Okuley et al., 1994; Moellering et al., 2010; Chen and Thelen, 2013)。最近在拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)中的脂质谱研究揭示了寒冷胁迫期间脂质组成和脂肪酸去饱和度的显著变化(Welti et al., 2002; Tan et al., 2018; Liu et al., 2022)。这些发现建立了脂质代谢与寒冷反应之间的紧密联系。然而,控制寒冷胁迫期间脂质重塑和代谢的具体调控因子仍有待确定。
热休克转录因子(HSFs)是由一组保守的真核生物应激反应基因编码的,已在多种物种中对热应激的响应中得到广泛研究(Scharf et al., 2012)。植物中的HSF根据其C端聚合域的结构分为三类:HSFA、HSFB和HSFC(Scharf et al., 2012)。HSFA作为热应激反应的关键正调控因子,而HSFB在不同植物物种中可能扮演多种角色,既可以正向也可以负向调节热应激反应(Bharti et al., 2004; Ikeda et al., 2011; Scharf et al., 2012)。在拟南芥中,HsfB1和HsfB2b作为热耐性的抑制因子,但它们的表达对于获得性耐热性是必要的(Ikeda et al., 2011)。在番茄中,HsfB1被发现作为热应激反应的核心抑制因子,并且是HsfA1a的普遍抑制因子(Fragkostefanakis et al., 2019)。最近,玉米B类HSF ZmHSF20被鉴定为热应激反应的负调控因子,在热应激期间抑制了A类HSF基因和细胞壁相关基因的表达(Li et al., 2024)。HSFA1已被证明在拟南芥中促进寒冷响应基因(COR基因)的表达并增强寒冷耐性(Olate et al., 2018)。然而,HSFB在寒冷应激反应中的调控机制尚未被探索。
本研究通过对213个自交玉米系面板进行GWAS筛选,发现HSF21,一个HSFB基因,与寒冷耐性密切相关。具有寒冷耐性优势等位基因的玉米在寒冷应激下表现出较高的HSF21表达,这是由于转录因子bZIP68(一个寒冷耐性的负调控因子)结合亲和力的降低。多组学分析表明,HSF21是玉米在寒冷应激下脂质代谢的重要调控因子,并对寒冷耐性做出了重要贡献。HSF21Hap1等位基因在不影响产量的情况下增强玉米的寒冷耐性。因此,HSF21可能是开发寒冷耐性玉米品种的优良遗传靶标。
结果
GWAS鉴定出HSF21与玉米寒冷耐性相关
为了评估年轻玉米种苗的寒冷耐性,我们测量了在寒冷处理后V2阶段玉米种苗真叶(真叶是指玉米植株从胚芽中长出的第一批功能性叶片)的相对受伤面积,研究对象为由213个自交玉米系组成的玉米群体(Yang et al., 2011; Li et al., 2013)(补充图1A)。我们观察到,在这个群体中,由寒冷胁迫引起的相对受伤面积存在显著变异,范围从0%到100%不等(补充图1B)。我们还发现,温带玉米系(NSS)子群体表现出比热带/亚热带玉米系(TST)和B73衍生系(SS)子群体更强的寒冷耐性(补充图1C)。GWAS分析在所有213个自交系中鉴定出了16个显著SNP(P < 1.033 × 10⁻⁵),在TST子群体(121个自交系)中另外鉴定出了8个显著SNP,这些SNP与寒冷耐性显著相关(图1A;补充图1D–1F)。因此,我们共鉴定出了24个遗传变异,涉及14个候选基因,这些基因与寒冷耐性紧密相关(补充表1)。其中,最显著的SNP,chr7.s_125861101(P = 1.153 × 10⁻⁷),位于编码HSF21的基因的启动子区域(Zm00001d020714)。值得注意的是,HSF21启动子中位置-683(相对于翻译起始位点ATG,可能是G或T)处的两种核苷酸与寒冷耐性呈显著相关关系,通过相对受伤面积进行评估(图1A和1B;补充图1G)。

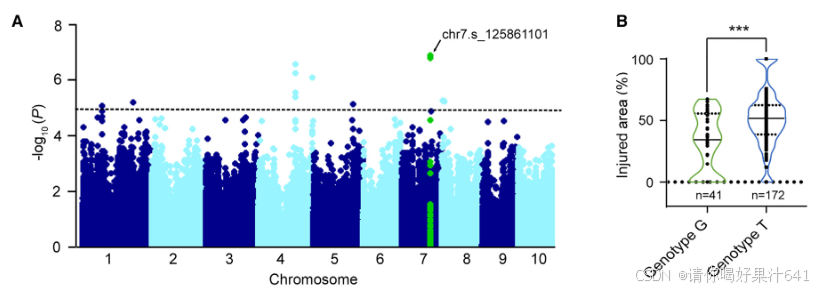

我们对HSF21进行了重新测序,并鉴定了41个SNP和28个插入/缺失(InDels),其次要等位基因频率(MAF)大于5%(补充表2)。上述提到的-683 bp处的SNP和HSF21启动子中的-693 bp处的InDel5与玉米寒冷耐性表现出最强的关联(P = 1.67 × 10⁻⁷),并在玉米群体中表现出强烈的连锁不平衡(LD)(r² = 0.95;图1C)。基于这一SNP(-683)和InDel5(-693),我们将460个自交玉米系分为三种单倍型组:Hap1、Hap2和Hap3(图1D;补充表3),分别占自交系的23%、31%和46%(补充图1H)。在寒冷条件下,Hap1组的玉米系相对受伤面积较低,而Hap2和Hap3组之间没有显著差异(图1E;补充图1I)。我们还检查了26个自交系中HSF21的表达,结果发现寒冷胁迫诱导了所有6个HSF21Hap1寒冷耐性玉米系的HSF21表达。然而,携带寒冷敏感等位基因(HSF21Hap2或HSF21Hap3)的20个自交系在寒冷处理前后HSF21的表达保持不变(图1F)。这些结果表明,HSF21Hap1对寒冷胁迫具有响应性,并且提示其在调节玉米寒冷耐性中起重要作用。

为了确定优越等位基因HSF21Hap1是否以及如何对玉米的寒冷耐性起作用,我们通过将寒冷耐性HSF21By815(Hap1)等位基因引入K22(Hap3)自交系,经过两代回交(F6植物,K22 × By815)后,开发了近等基因系(NILs)(Xiao et al., 2016)。NIL-Hap3植物表现出对寒冷胁迫的过敏反应,其相对受伤面积较NIL-Hap1植物更大(图1G和1H)。在NIL-Hap3植物中,HSF21的基础表达水平相对较低,且在寒冷处理后未发生变化。相比之下,在正常条件下,NIL-Hap1植物中HSF21的表达是NIL-Hap3植物的两倍,并且在寒冷胁迫下,HSF21的表达诱导水平是NIL-Hap3植物的三到四倍(图1I)。这些发现强烈表明,HSF21Hap1作为一个寒冷耐性等位基因,其表达在寒冷胁迫下被诱导。

HSF21促进玉米幼苗和萌发阶段的寒冷耐性
系统发育分析显示,玉米的HSF家族包括30多个成员,分为三个簇:HSFA、HSFB和HSFC(补充图2A)。HSF21属于HSFB亚家族(补充图2B)。定位研究表明,HSF21蛋白主要定位于转染了HSF21-GFP(绿色荧光蛋白)构建体的玉米原生质体的细胞核中(补充图2C)。表达分析显示,HSF21在根、叶和幼苗苗条中高度表达,而在生殖器官中的表达相对较弱(补充图2D)。
为了深入研究HSF21的功能,我们通过CRISPR-Cas9基因编辑技术在属于Hap3的LH244自交系中生成了hsf21突变体。hsf21-1突变体在第一外显子中有一个43-bp的缺失,hsf21-2突变体则在第二外显子中有一个22-bp的缺失(补充图3A)。在正常温度下,hsf21突变体与野生型在幼苗阶段表现相似(图2A);然而,两个突变体相比野生型植物对寒冷胁迫表现出更大的敏感性(图2A)。在4°C寒冷处理后,两个突变体的相对受伤面积和离子泄漏显著高于野生型植物(图2B和2C)。
我们还在携带寒冷敏感Hap3单倍型的LH244自交系中开发了通过泛素(Ubi)启动子驱动的HSF21过表达转基因系(HSF21-OE)。两个独立的HSF21-OE转基因系在寒冷胁迫后的相对受伤面积和离子泄漏量显著降低,显示出明显增强的寒冷耐性(图2D–2F;补充图3B)。这些结果表明,HSF21在幼苗阶段正向调节玉米的寒冷耐性。
寒冷胁迫通常会降低种子萌发初期的活力。在早播田间试验中,当白天/夜晚播种温度为21°C/10°C时(补充图3C),野生型的最终萌发率约为43%。相比之下,两个hsf21突变体的萌发率分别为28%和33%(图2G和2H)。而两个HSF21-OE转基因系则表现出较早的萌发,比野生型种子早1到2天发芽,最终萌发率分别为88%和92%(图2H)。在田间试验中,当白天/夜晚播种温度为27°C/13°C时,观察到类似现象(补充图3C–3E)。进一步的实验验证了HSF21-OE种子在寒冷条件下的萌发更好,而两个hsf21突变体在12°C恒定低温下的萌发较野生型差(图2I)。这些结果表明,HSF21不仅促进了早期活力,还增强了幼苗对寒冷胁迫的耐受性。
HSF21启动子中的自然变异影响bZIP68结合亲和力
详细的序列分析显示,HSF21启动子中存在自然变异的区域包含一个假定的A-box(50-TACGTA-30),这是bZIP转录因子识别的顺式作用元件(Jakoby et al., 2002)。在HSF21翻译起始位点上游2 kb的序列中,只有一个假定的A-box元件(约在-683 bp处)(补充表4)。HSF21Hap1系中的SNP(-683 bp)导致了假定A-box的错配(50-GACGTA-30;下划线部分为错配的核苷酸),而InDel5(-693 bp)导致了假定A-box相邻的5 bp缺失(补充图3F)。
我们之前的研究表明,bZIP68通过结合目标COR基因的A-box元件,负向调节玉米的寒冷耐性(Li et al., 2022b)。接下来,我们检查了bZIP68是否识别HSF21启动子中的A-box来调节其表达。我们发现,重组bZIP68能够特异性地结合含有假定A-box的HSF21启动子片段,并且这种结合会受到与未标记完整DNA探针的竞争抑制,但不受带有突变A-box的冷探针的抑制,如电泳迁移率变动实验(EMSA)所示(图3A)。随后的染色质免疫沉淀(ChIP)实验表明,bZIP68在假定的A-box处富集,并且在寒冷处理下,这种富集程度得到了增强(图3B)。与这些结果一致的是,bzip68突变体中HSF21的表达上调,而bZIP68-OE转基因植物中HSF21的表达下调(图3C)。
为了评估HSF21与bZIP68之间的遗传相互作用,我们通过将hsf21-1与bzip68-1交配生成了hsf21 bzip68双突变体。与野生型相比,bzip68-1突变体表现出更强的寒冷耐性,这与之前的研究结果一致(Li et al., 2022b)。然而,hsf21 bzip68双突变体表现出与hsf21单突变体相当的寒冷敏感性,且两者对寒冷胁迫的敏感性均高于野生型(图3D–3F)。综合来看,这些结果表明,bZIP68直接结合HSF21启动子中的A-box,负向调节其表达。
我们进一步使用电泳迁移率变动实验(EMSA)分析了bZIP68与来自HSF21启动子三种不同单倍型的探针的结合亲和力。重组bZIP68与来自HSF21Hap1启动子的探针的结合亲和力明显低于与来自HSF21Hap2或HSF21Hap3启动子探针的结合亲和力,后两者与bZIP68的结合亲和力相当(图3G)。与这一结果一致,瞬时转录激活实验表明,bZIP68抑制了HSF21Hap3启动子活性,无论是否进行寒冷处理,但对HSF21Hap1启动子活性几乎没有影响(图3H)。
接下来,我们在LH244背景下生成了两个基因编辑突变体,hsf21-p1和hsf21-p2,这两个突变体在HSF21启动子中包含了含有主要SNP和InDel5的区域缺失(补充图3G)。无论是否进行寒冷处理,hsf21-p1和hsf21-p2突变体中的HSF21表达水平都比野生型略高,并且突变体保留了寒冷诱导HSF21的能力(图3I)。这些突变体表现出较强的寒冷耐性,与野生型相比,相对受伤面积和离子泄漏有所减少(图3J–3L)。这些结果强调了HSF21启动子中的自然变异在bZIP68调控HSF21表达中的重要性,而HSF21的表达进而影响玉米的寒冷耐性。
HSF21在寒冷胁迫响应中调控脂质代谢
为了表征HSF21在寒冷胁迫下调控的通路,我们对暴露在4°C或对照条件下12小时的14天龄野生型和hsf21-1幼苗进行了转录组深度测序(RNA-seq)。通过比较野生型和hsf21-1幼苗在4°C或对照条件下的差异表达基因(DEGs),我们共鉴定出1672个HSF21调控的DEGs(在25°C时1442个DEGs,在4°C时328个DEGs)(P < 0.01且绝对倍数变化 ≥ 1.5;图4A;补充表5和6)。基因本体(GO)分析显示,差异表达基因在光合作用、寒冷应激响应、脂质代谢、细胞因子反应和质体组织等通路中富集(图4B)。先前的研究表明,脱水响应元件结合因子1(DREB1)基因是植物寒冷耐性的核心转录因子(JagloOttosen et al., 1998;Shi et al., 2018;Zeng et al., 2021;Li et al., 2022b)。然而,我们观察到无论是否进行寒冷处理,野生型与hsf21突变体之间DREB1的表达没有明显差异(图4C–4E),这表明HSF21在玉米寒冷耐性中的调节作用可能与DREB1无关。
脂质代谢已被发现与植物的寒冷耐性密切相关(Steponkus, 1984;Miquel et al., 1993;Wang et al., 2006;Moellering et al., 2010)。在HSF21调控的差异表达基因中,脂质代谢相关基因显著富集,包括甘油-3-磷酸酰基转移酶(GPATs)、硬脂酰-酰基载体蛋白去饱和酶(SADs)、脂肪酸去饱和酶(FADs)和3-酮酰基-CoA合成酶(KCSs),这些酶是甘油脂代谢的关键酶(Li-Beisson et al., 2013)(图4F;补充表7)。随后,我们使用DNA亲和纯化测序(DAP-seq)分析了HSF21的基因组范围靶点(补充表8)。我们发现热休克元件(TTCTAGAANNTTC)是HSF21结合位点中最丰富的顺式作用元件,且该元件的最大基序长度为13-14 nt,位于转录起始位点附近(通过MEME-ChIP分析得出)(Machanick和Bailey, 2011)(图4G)。在两个独立的重复实验中,共鉴定出2272个HSF21靶基因(补充表9)。有趣的是,通过整合基因组查看器(IGV)观察到,HSF21的结合峰出现在或接近GPAT6、GPAT10、FAD7/8、SAD1和KCS29/34(图4H和4I)。此外,寒冷处理后,在hsf21幼苗中,七个GPATs和七个SADs或FADs的表达下调,而八个KCSs的表达显著上调(图4J)。在这些基因中,Male sterile 33(Ms33,也称GPAT2)和SAD2已被证明通过调节甘油脂合成促进玉米的寒冷耐性(Gao et al., 2024)。KCS是植物中合成超长链脂肪酸的关键限速酶(Li-Beisson et al., 2013)。实际上,在LH244中通过泛素(Ubi)启动子驱动的KCS29过表达转基因植物在寒冷胁迫下表现出更强的敏感性,相对受伤面积和离子泄漏量高于野生型植物(图4K–4N)。此外,代谢物GWAS已鉴定出与寒冷响应代谢物相关的KCS1和KCS3的自然变异(Jiang et al., 2022)(补充图4A和4B)。
综上所述,这些发现表明,HSF21通过调控脂质代谢通路有助于提高玉米的寒冷耐性。
为了进一步探究HSF21在寒冷胁迫下调控脂质代谢的作用,我们对14天龄的野生型和hsf21-1突变体幼苗在对照或寒冷条件(4°C,48小时)下进行了一次全面的靶向脂质组分析。为了检测主要的脂质类群,我们分别使用液相色谱法检测了糖脂(硫代氯酸甘油二酯[SQDG],二半乳糖甘油二酯[DGDG],单半乳糖甘油二酯[MGDG])、甘油脂(甘油三酯[TAG]和甘油二酯[DAG])、以及磷脂(溶血磷脂酸[LPA],磷脂酸[PA],磷脂酰胆碱[PC],磷脂酰乙醇胺[PE],磷脂酰甘油[PG],和磷脂酰肌醇[PI])(图5A;补充表10)。通过绘制整个脂质组的热图,我们发现寒冷处理组与未处理组的数据之间存在明显差异。同样,主成分分析(PCA)也显示了寒冷处理组与对照组之间的显著差异,尤其在4°C下,hsf21突变体组的差异更为显著(图5B和5C)。尽管所有组在寒冷处理后总甘油脂含量均有所下降,但在寒冷处理的hsf21幼苗中,这一下降更为显著(图5D)。近期的研究表明,野生型植物在寒冷暴露(4°C 48小时)后,脂质组成和含量发生了选择性变化(Gao et al., 2024)。在本研究中,我们发现,寒冷处理的hsf21突变体中,磷脂(PG和PE)和糖脂(DGDG和SQDG)的含量明显低于野生型(图5E–5H)。相反,野生型幼苗的PA、DAG和LPA水平有所增加,而hsf21幼苗则在寒冷胁迫下表现为下降或没有变化(图5F–5H),这表明在寒冷胁迫下,hsf21的脂质代谢平衡被破坏。
细胞膜中不饱和脂质的含量已被证明与植物的寒冷耐性相关(Steponkus, 1984;Miquel et al., 1993;Orvar et al., 2000)。显著的是,在寒冷处理的hsf21突变体中,关键的不饱和糖脂、磷脂和甘油脂的水平,如多不饱和DGDG、单/二不饱和PE和不饱和DAG,尤其是DGDG36:6、PE34:2和DAG36:4,都明显低于野生型(图5I–5K)。这些结果表明,HSF21在维持寒冷胁迫下细胞脂质代谢稳态中起着至关重要的作用。
HSF21Hap1等位基因通过调节脂质代谢稳态赋予玉米寒冷耐性而不影响产量
为了评估HSF21Hap1优越等位基因对脂质代谢稳态的影响,我们分析了14天龄的NIL-HSF21K22(Hap3)和NIL-HSF21By815(Hap1)植物在有无寒冷胁迫(4°C 48小时或25°C 48小时)下的主要脂质类群含量,采用高覆盖度靶向脂质组学方法。 在25°C下,NIL-HSF21K22和NIL-HSF21By815植物之间的脂质水平没有显著差异(补充图5A–5C)。然而,在4°C寒冷处理48小时后,NIL-HSF21K22植物中的磷脂(PA、PG、PE和PC)和糖脂(DGDG和MGDG)含量明显低于NIL-HSF21By815植物(图6A–6C;补充图5D)。具体来说,大多数主要的不饱和磷脂和糖脂,特别是PC34:2、PE34:2、DGDG36:6和MGDG36:6,在寒冷处理的NIL-HSF21K22(Hap3)植物中显著低于寒冷处理的NIL-HSF21By815(Hap1)植物(图6D–6G)。这些发现表明,HSF21Hap1的有利等位基因,通过较高的表达水平,有助于在寒冷胁迫下维持脂质代谢稳态,从而调控玉米的寒冷耐性。
接下来,我们进行了田间试验,以评估HSF21是否在不影响生长和产量的情况下增强寒冷耐性。在正常生长条件下,其中NIL-HSF21By815与NIL-HSF21K22或野生型与启动子编辑等位基因hsf21-p1和hsf21-p2之间的HSF21表达差异仅为1.5到2.0倍(图1I和3I),但在植物形态、叶数、开花时间、穗长、穗行数、穗级数、每穗籽粒重和千粒重等农艺性状或产量性状上没有显著差异(补充图6和7)。这些发现表明,HSF21Hap1的有利等位基因赋予玉米寒冷耐性,而在正常生长条件下不对生长和产量产生明显的负面影响。
HSF21在田间提高玉米产量
提高早期发育阶段的寒冷耐性并增强后期的产量是缓解玉米生长与防御之间权衡的有效策略。因此,我们进行了田间试验,使用通过交配HSF21-OE(OE1、OE2和OE3)纯合系或野生型LH244与选定自交系(T13)生成的F1转基因杂交种与对照杂交种进行比较。2017年和2018年连续两年在四个地点的试验结果显示,转基因F1杂交种的产量显著提高,分别为7.5%到10.6%和5.4%到10.3%(补充图8A和8B)。这些F1转基因杂交种表现出增强的穗形态,包括更长的穗长和更大的穗径(补充图8C–8E)。此外,每穗籽粒重和千粒重在这些转基因系中也显著高于对照组(补充图8F和8G)。进一步地,当HSF21-OE转基因系与两个优质自交系(中3和长7-2)进行交配时,F1转基因杂交种的产量始终高于对应的优质杂交对照组,同时伴随有改善的穗表型(补充图8H–8Q)。综上所述,这些结果表明,HSF21过表达对农艺性状有积极影响,导致玉米在不同环境下产量的显著提高。
讨论
玉米从热带起源向温带气候扩展的过程要求其适应寒冷胁迫。尽管已有多项基因组范围关联研究(GWAS)旨在揭示玉米寒冷耐性的遗传基础(Yi et al., 2021;Zhang et al., 2021),但其分子机制仍大部分未知,且仅有少数基因已被功能性地表征。本研究鉴定了HSF21基因作为与玉米寒冷耐性显著相关的位点。HSF21能够提高玉米幼苗的寒冷耐性,并增强种子早播时的早期活力。HSF21Hap1等位基因的自然变异通过其启动子内的变异,抑制了bZIP转录因子(如bZIP68,玉米寒冷耐性的负调控因子)的结合,从而增加了HSF21的表达(Li et al., 2022b)。此外,我们揭示了HSF21在维持寒冷胁迫下脂质稳态中的不可或缺作用,从而正向调节玉米的寒冷耐性(图7)。脂肪酸去饱和度和磷脂含量等脂质组成方面的变化早已与寒冷耐性相关联(Miquel et al., 1993;Iba, 2002;Welti et al., 2002;Moellering et al., 2010;Chen and Thelen, 2013)。然而,对于这些变化背后的调控机制知之甚少。通过使用hsf21无效突变体,我们最大化了检测基因表达和脂质代谢物谱变化的能力,从而揭示了HSF21调控的全面寒冷信号和代谢通路。我们的多组学方法揭示,HSF21主要通过调节脂质代谢影响寒冷耐性。重要的是,B型HSF21作为转录激活因子,直接调控编码脂质代谢关键酶的GPAT、FAD、SAD和KCS基因的表达(Li-Beisson et al., 2013)。HSFBs通常被认为是拟南芥热应激反应的转录抑制因子,这是由于其保守的C端LFGV基序(Ikeda et al., 2011)。在本研究中,我们发现,寒冷处理后,hsf21突变体中总量和不饱和的叶绿体特异性糖脂的水平显著下调,导致脂质代谢缺陷,从而对寒冷胁迫表现出过度敏感性。这一发现扩展了HSFB转录因子的已知功能,并揭示了寒冷适应与脂质代谢通路之间的内在联系。值得注意的是,KCS基因的自然变异与寒冷响应代谢物相关联(Jiang et al., 2022),这表明这些HSF靶向的脂质代谢基因是提高作物寒冷耐性的潜在遗传资源。
增强植物防御通常会阻碍生长并导致产量损失(He et al., 2022)。近期研究表明,通过遗传重编程(Campos et al., 2016)、基因编辑(Oliva et al., 2019)、诱导基因表达(Wang et al., 2018)和自然变异(Li et al., 2017)等手段,可能有机会解耦生长与防御之间的权衡。例如,通过基因组编辑将TaMlo-R32等位基因引入优质小麦(Triticum aestivum)品种,提高了对白粉病的抗性,而没有影响生长或产量(Li et al., 2022a)。在水稻中,两个自然等位基因IDEAL PLANT ARCHITECTURE1(IPA1)的ipa1-1D和ipa1-2D与IPA1表达增加、粮食产量提高、抗病性增强和寒冷耐性提升相关(Jiao et al., 2010;Wang et al., 2018;Jia et al., 2022)。此外,RESISTANCE TO BLAST1(RBL1)的一个等位基因赋予了水稻广谱抗病性,同时不影响产量(Sha et al., 2023)。在本研究中,我们展示了HSF21Hap1和HSF21启动子编辑等位基因在提高寒冷耐性的同时保持产量。这是通过战略性遗传改造解耦作物寒冷耐性特征与产量惩罚的一个良好例子。该发现与先前的定量性状位点分析结果一致,表明与寒冷相关的幼苗性状对玉米总产量的影响最小(Leipner et al., 2008)。此外,HSF21在玉米中的过表达增强了早期生长阶段的寒冷耐性,同时改善了后期发育中的农艺性状和产量,表明除了寒冷耐性外,还具有更广泛的益处。由于HSF21的有利变异尚未广泛应用于品种育种,我们推测HSF21是发展抗寒冷应力、同时保持产量的先进作物品种的关键遗传成分,从而促进未来农业的可持续发展。
方法
质粒构建与基因转化
玉米转基因植物和CRISPR-Cas9突变体是在LH244自交系中生成的,实验由中国农业大学作物功能基因组学与分子育种中心进行。bZIP68质粒以及bZIP68的转基因植物和突变体已在先前的研究中报告(Li et al., 2022b)。HSF21的全长编码序列从LH244中扩增并克隆到Super:1300-GFP、pBECXUN-HF和pBCXUN载体中,用于构建过表达载体。为了获得pHSF21:LUC构建体,扩增并克隆了HSF21的-1.5 kb启动子序列到pGreenII0800-LUC载体中。
位于HSF21编码区的靶点被克隆到pBUE411载体中,通过CRISPR-Cas9编辑系统生成hsf21突变体(hsf21-1和hsf21-2)(Xing et al., 2014)。为了生成HSF21启动子编辑系(hsf21-p1和hsf21-p2),设计了两条针对HSF21启动子的引导RNA序列,并将其插入pXUE411C-BG载体中。引物和引导RNA序列列在补充表11中。转基因植物通过农杆菌介导的玉米转化获得。
植物材料
本研究使用的玉米自然群体已在先前的研究中报告(Yang et al., 2011;Li et al., 2013)。通过将寒冷耐性HSF21By815等位基因(Hap1)引入K22自交系(Hap3),生成了近等基因系(NILs)。这一过程通过对F6植物(K22 × By815)进行两代连续回交,与K22自交系交配(Xiao et al., 2016)。每一代都选择HSF21杂合植物并与K22回交。经过一代自交后,我们获得了携带同源的HSF21By815或HSF21K22等位基因的分离BC2F8植物。
对于hsf21 bzip68突变体,将hsf21-1植物与bzip68-1突变体进行顶端交配,生成F1转基因种子,并在F1转基因种子自交一代后鉴定出纯合的hsf21 bzip68突变体。
对于F1转基因杂交种(OE1 × T13, OE2 × T13, OE3 × T13, OE1 × Chang7-2, OE2 × Chang7-2, OE1 × Zong3, 和OE2 × Zong3),将HSF21过表达系与选定的自交系测试品系(T13, Chang7-2, 和 Zong3)进行顶端交配,生成F1转基因种子;LH244自交系与T13、Chang7-2和Zong3进行顶端交配,生成F1对照杂交种(LH244 × T13, LH244 × Chang7-2 和 LH244 × Zong3)。
植物生长与处理
玉米种子被播种在一个塑料花盆(30 × 20 × 15 cm),花盆内填充有植物灰、珍珠岩和Pindstrup土壤(丹麦,1:1:1的混合物),并在温室中生长14天(V2阶段),温度为25°C,光照周期为16小时光照/8小时黑暗,光照强度为150 μmol m⁻² s⁻¹,湿度为60%。寒冷处理和离子泄漏测量按照之前的方法进行(Zeng et al., 2021)。14天龄的幼苗在4°C下处理3-4天后,转移至25°C并在黑暗中保持12小时,再恢复至25°C处理2天。恢复后拍摄照片。相对受伤面积使用以下公式计算:相对受伤面积(%)= (L3 + L4) / (L1 + L2) × 100,其中L1为第一片叶的总面积,L2为第二片叶的总面积,L3为第一片叶的受伤面积,L4为第二片叶的受伤面积。
对于离子泄漏测定,取叶片放入含有10毫升去离子水的15毫升试管中,测量初始电导率S0。将试管轻轻摇晃1小时后,测量电导率S1。然后将样品煮沸30分钟,再在22°C下摇晃10分钟,随后测量S2。离子泄漏按以下公式计算:(S1 - S0) / (S2 - S0)。
对于早播田间试验,野生型、HSF21-OE(OE-1和OE-2)和hsf21(hsf21-1和hsf21-2)各取24颗种子,播种在一个塑料桶中(34 × 34 cm)。播种后每天观察并统计表型和萌发率。
玉米寒冷耐性表型分析与GWAS
玉米寒冷耐性的表型分析是在一个由213个自交系组成的自然变异群体中进行的,该群体来自一个多样化的玉米关联面板,之前已有相关描述(Yang et al., 2011)。对于每个自交系,选择五个V2阶段的幼苗,在寒冷室(4°C)中处理3天,并在恢复2天后拍摄冷冻幼苗的真叶图像。测量每个自交系的相对受伤面积,并计算平均值(n = 3)。GWAS分析使用Tassel 5.0进行,采用了覆盖整个玉米基因组的558,659个高质量SNP(MAF ≥ 0.05)(Fu et al., 2013;Li et al., 2013)。在比较了控制群体结构(Q)的普通线性模型后,使用了混合线性模型。根据PLINK软件(窗口大小50,步长50,r² ≥ 0.2)筛选出97,438个独立的SNPs,通过过滤强连锁不平衡(LD)的SNP,最终设定GWAS的显著性阈值为P < 1.03 × 10⁻⁵(1/97,438)。对于基于HSF21的关联分析,从460个自交系中鉴定出了69个变异(MAF ≥ 0.05),包括SNP和InDel。通过Tassel 5.0计算这些变异与表型的关联及其成对LD(Bradbury et al., 2007)。一个位于-683处的SNP和-693处的InDel5在强LD下与玉米寒冷耐性显著相关。变异信息见补充表2。
系统发育分析
HSF21同源基因和旁系基因的氨基酸序列来自Gramene数据库(https://ensembl.gramene.org/Zea_mays/Info/Index)。使用MEGA 7.0软件和默认的成对与多重比对参数,采用最大似然法生成联合无根树。
亚细胞定位
为了确定HSF21的亚细胞定位,Super:HSF21-GFP融合构建体被转化到玉米原生质体中。经过16小时的孵育后,在共聚焦激光扫描显微镜(Carl Zeiss LSM 710)下捕捉荧光信号。GFP的激发波长为488 nm,检测范围为515-530 nm。
EMSA
EMSA实验使用LightShift化学发光EMSA试剂盒(Thermo Fisher)按照制造商的说明进行,略作修改(Li et al., 2022b)。简而言之,向结合反应缓冲液中加入1.5 μg的纯化MBP-bZIP68蛋白(在pMAL-c5X载体中表达)。将反应在25°C下进行20分钟,并使用热循环仪(Bio-Rad)。用于生成生物素标记和非标记探针的互补寡核苷酸序列见补充表11。
转录活性测定
双荧光素酶(LUC)转录活性测定按照先前的方法进行(Li et al., 2022b)。将玉米原生质体与Super:bZIP68-GFP构建体和pHSF21By815:LUC或pHSF21K22:LUC报告基因共同转染,在25°C下孵育14小时。寒冷处理时,样品在4°C下再处理2小时。Super:GFP作为内对照。使用LUC底物(Promega)检测LUC活性。使用GloMax 20/20光度计(Promega)测定萤火虫LUC/Renilla LUC比值,以确定HSF21启动子的活性。
RT–qPCR和RNA-seq分析
从14天龄温室中生长的玉米幼苗中提取总RNA,使用TRIzol试剂(Invitrogen)。经过DNase(Takara)处理后,使用M-MLV反转录酶(Promega)进行反转录(RT),然后在7500实时PCR系统(Applied Biosystem)上使用SYBR Green试剂(Takara)和基因特异性引物(补充表11)进行定量PCR(RT–qPCR)分析。Ubiquitin(Ubi)基因作为对照(Zeng et al., 2021)。
RNA-seq分析按照先前的方法进行(Li et al., 2022b)。收集14天龄的玉米材料(野生型和hsf21-1),在有或没有4°C处理12小时的条件下,用TRIzol试剂提取RNA。进行了三次独立的重复实验。RNA-seq文库的构建和测序由Berry Genomics(北京)使用Illumina NovaSeq 6000平台完成。得到的读数使用HISAT2(v.2.0.4)并使用默认参数进行清洗,随后映射到玉米参考基因组(B73 RefGen_v4,AGPv4)。原始数据(原始读数)以fastq格式进行质量控制,使用FastQC(v.0.11.9)。每个基因的读数计数通过FeatureCounts(v.2.0.1)获得。差异表达基因(DEGs)使用P < 0.01和hsf21-1系与野生型植物的绝对倍数变化≥1.5的阈值进行识别。使用R包pheatmap(v.1.0.12)进行DEGs的聚类分析。DEG簇的GO富集分析使用agriGO(http://systemsbiology.cau.edu.cn/agriGOv2)进行。
ChIP和DAP-seq分析
ChIP分析按照先前的方法进行(Li et al., 2022b)。14天龄的野生型、Ubi:bZIP68-GFP植物在有或没有4°C处理12小时后进行处理。然后,将幼苗在真空条件下用1%甲醛固定10分钟。通过加入2 M甘氨酸使反应停止,最终浓度为0.125 M,混合物在25°C下真空孵育5分钟。接着,分离染色质并进行超声破碎,使用GFP琼脂糖珠(ChromoTek)共同免疫沉淀与bZIP68-GFP蛋白相关的DNA片段。通过qPCR定量DNA片段的富集,使用补充表11中列出的引物。
DAP-seq分析按照先前的方法进行,略作修改(O’Malley et al., 2016)。从14天龄的野生型幼苗中提取基因组DNA,并使用Bioruptor(Diagenode)将其破碎至约300 bp。GST-HSF21蛋白与谷胱甘肽Sepharose 4B珠(GE Healthcare)孵育2小时,然后将包裹的珠子与500 ng碎片化的DNA孵育1小时。去除未结合的DNA后,将亲和纯化的DNA洗脱并用于测序。进行了两次重复实验,每个重复获得500万条读数。清洗后的数据使用HISAT2(v.2.2.4)和默认参数映射到参考基因组(Zm-B73-REFERENCE-NAM-5.0)。为了识别有效的结合基序,提取每个峰值顶部周围的500 bp序列,并使用默认设置通过MEME-ChIP数据库进行分析(Machanick和Bailey, 2011)。
脂质组学分析
脂质组学分析按照先前的方法进行(Gao et al., 2024)。简而言之,14天龄的野生型和hsf21-1植物在有或没有4°C处理48小时后进行处理。收集新鲜叶片样本(约60 ± 3 mg)以去活脂质水解酶,并在-80°C下保存,直至分析。所有定量实验均使用内标进行。磷脂使用Phenomenex Luna Silica 3-μm硅胶柱(内径150 × 2.0 mm)分离。对于甘油脂和糖脂,按照先前报告的方法(Lam et al., 2017;Liu et al., 2020)使用Phenomenex Kinetex 2.6-μm C18柱(内径100 × 4.6 mm)进行定量。大多数内标来自Avanti Polar Lipids(美国阿拉巴马州阿拉巴斯特)、Matreya LCC(美国宾夕法尼亚州州学院)和CDN Isotopes(加拿大魁北克省庞特克莱尔)。脂质含量以叶组织中的毫摩尔/克鲜重表示。
基因编辑植物和NIL植物的田间试验
所有基因编辑植物(hsf21-p1和hsf21-p2)和野生型植物的田间试验都采用随机区组设计进行,并在2022年在河北省卓州(39°29’01.000"N, 115°58’05.000"E, ZZ)进行三次重复试验。每个重复为一个种植区,每个区块为5米长的双行,行间距为0.5米。所有植物都进行自然授粉。在授粉后评估农艺性状,收获后测量产量相关性状。为了研究粮食产量,从三个区块中选取21株植物测量农艺性状和产量相关性状。对于NIL植物,田间试验采用随机区组设计,2022年在三亚(18°38’09.500"N, 109°19’06.600"E)进行三次重复试验。所有植物均为自然授粉。在三个区块中测量了30株植物。
F1杂交种田间试验
F1杂交种LH244 × T13和HSF21-OE × T13植物于2017年和2018年在四个地点进行田间试验:卓州(39°29’00.000"N, 115°58’05.000"E)、银川(38°28’23.3000"N, 106°14’34.2200"E)、公主岭(43°30’16.000"N, 124°49’02.200"E)和包头(41°46’09.9500"N, 109°58’25.3900"E)。所有植物均为自然授粉。异合F1转基因杂交种与F1野生型对照的田间试验采用随机区组设计,至少有12个重复/区块。每个重复为一个种植区,每个区块为5米长的双行,行间距为0.5米。F1转基因杂交种(HSF21-OE × Chang7-2 和 HSF21-OE × Zong3)与其野生型对照(LH244 × Zong3 和 LH244 × Chang7-2)于2022年在卓州(39°29’00.000"N, 115°58’05.000"E)种植。收获后,从三个区块中的21穗玉米测量了与粮食产量相关的性状。
统计分析
两个样本之间的差异使用双尾学生t检验进行评估。对于多重比较,使用单因素方差分析(ANOVA)后跟随Tukey的多重比较检验进行评估。所有统计分析均使用GraphPad Prism 8.0版本进行。
数据与代码可用性
支持本研究结果的数据可在论文及其补充信息文件中找到。基因组信息和序列已从MaizeGDB(https://www.maizegdb.org)和Gramene数据库(https://ensembl.gramene.org/Zea_mays/Info/Index)下载。RNA-seq和DAP-seq数据已存储在NCBI基因表达综合数据库(www.ncbi.nlm.nih.gov/geo/)的存取号PRJNA996745和中国国家基因组数据中心(https://ngdc.cncb.ac.cn)下的存取号PRJCA028660中。广泛使用的群体和基因型信息可以从http://www.maizego.org/Resources.html下载。

























 613
613

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








