Logistic回归全流程代码,包含:导入数据 数据划分 基线表生成 LASSO回归
批量单因素logistic 多因素logistic 列线图 ROC 校准曲线
DCA
这个程序主要是对一个数据集进行分析和建模。下面我会逐步解释程序的每个部分。
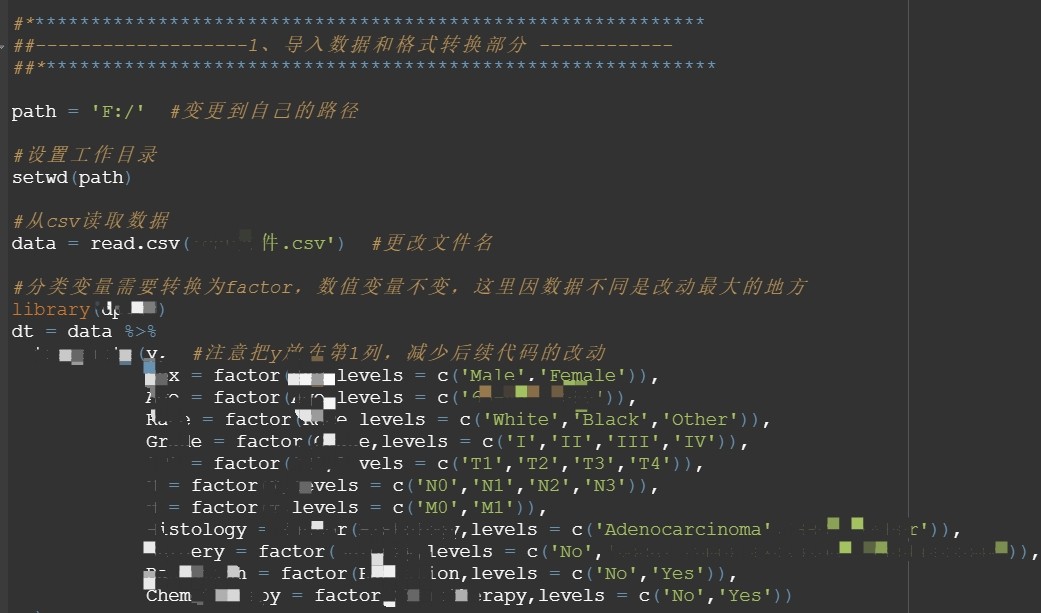
导入数据和格式转换部分:
首先,程序指定了一个路径变量path,你需要将其更改为你自己的路径。
然后,程序使用read.csv函数从一个csv文件中读取数据,并将其存储在变量data中。
接下来,程序使用dplyr包中的transmute函数对数据进行格式转换。它将一些列转换为因子变量(分类变量),并保持其他列不变。这里使用了factor函数和levels参数来指定每个因子变量的水平。
最后,程序使用VIM包中的aggr函数检查数据中是否存在缺失值,并以可视化的方式显示缺失情况。
数据划分部分:
首先,程序使用set.seed函数设置随机数种子,以确保结果的可重现性。
然后,程序使用caret包中的createDataPartition函数将数据集划分为训练集和测试集,比例为70:30。划分的结果存储在变量num中。
最后,程序使用划分结果将数据集分为训练集和测试集,分别存储在变量train和test中。
基线表生成部分:
首先,程序使用tableone包中的CreateTableOne函数生成基线表。该函数接受一些参数,包括要汇总的变量、分层变量、数据来源等。
在这个程序中,基线表中的变量由myVars和catVars指定。myVars是要汇总的所有变量,catVars是分类变量。
然后,程序使用print函数打印基线表,并将结果存储在变量tabMat中。
最后,程序使用write.csv函数将基线表保存为csv文件。
LASSO筛选变量部分:
首先,程序使用model.matrix函数将自变量数据转换为矩阵形式,以满足LASSO回归的要求。这里使用了model.matrix和data.matrix函数来进行数据转换。
然后,程序使用glmnet包中的glmnet函数进行LASSO回归。该函数接受自变量矩阵x、因变量向量y、回归类型family和正则化参数alpha等。
接下来,程序使用plot函数绘制LASSO回归的结果图,显示不同正则化参数下的系数收缩情况。
然后,程序使用交叉验证方法选择最佳的正则化参数,并使用cv.glmnet函数进行交叉验证。该函数接受自变量矩阵x、因变量向量y、回归类型family、正则化参数范围lambda和交叉验证折数nfolds等。
最后,程序使用coef函数提取最小正则化参数下的非零系数,并将结果存储在变量Coefficients中。
批量单因素Logistic回归部分:
首先,程序定义了一个函数Uni_log,用于进行批量单因素Logistic回归。该函数接受一个自变量名作为参数,并返回回归结果的数据框。
然后,程序使用colnames函数提取训练集中的变量名,并存储在变量var.names中。
接下来,程序使用lapply函数对var.names中的每个变量应用Uni_log函数,得到一个包含多个回归结果的列表。
最后,程序使用ldply函数将列表中的回归结果整合成一个数据框,并将结果保存为csv文件。
多因素Logistic回归部分:
首先,程序使用as.formula函数构建多因素Logistic回归的公式,其中包含单因素回归中显著的变量。
然后,程序使用glm函数进行多因素Logistic回归。该函数接受公式form、回归类型family和数据来源data等。
接下来,程序使用summary函数对回归结果进行总结,并提取系数、标准误差、置信区间和p值等。
最后,程序将提取的结果整合成一个数据框,并将结果保存为csv文件。
列线图、模型验证部分:
这部分包括ROC曲线、校准曲线和DCA曲线的绘制。
首先,程序使用pROC包中的roc函数计算训练集和测试集的ROC曲线。
然后,程序使用plot函数绘制ROC曲线,并标出AUC值。
接下来,程序使用calibrate函数计算训练集和测试集的校准曲线。
最后,程序使用ggDCA包绘制DCA曲线。
这个程序涉及到的知识点包括数据导入和格式转换、数据划分、基线表生成、LASSO回归、批量单因素Logistic回归、多因素Logistic回归、模型验证等
以下是一个示例的Logistic回归全流程代码,包含了你提到的各个部分。请注意,这只是一个示例,具体实现可能因为数据集和软件包的不同而有所不同。你可以根据自己的需求进行修改和调整。
```R
# 导入所需的包
library(readr)
library(dplyr)
library(VIM)
library(caret)
library(tableone)
library(glmnet)
library(pROC)
library(calibrate)
library(ggDCA)
# 导入数据和格式转换部分
path <- "your_data_path" # 将路径更改为你自己的路径
data <- read_csv(file.path(path, "data.csv"))
# 格式转换
data <- data %>%
mutate(factor_var1 = as.factor(var1),
factor_var2 = as.factor(var2),
factor_var3 = as.factor(var3))
# 检查缺失值
aggr(data)
# 数据划分部分
set.seed(123) # 设置随机数种子
num <- createDataPartition(data$target, p = 0.7, list = FALSE)
train <- data[num, ]
test <- data[-num, ]
# 基线表生成部分
myVars <- c("var1", "var2", "var3", "var4")
catVars <- c("factor_var1", "factor_var2", "factor_var3")
tabMat <- CreateTableOne(vars = myVars, strata = catVars, data = train)
print(tabMat)
write.csv(tabMat, file.path(path, "baseline_table.csv"))
# LASSO回归
x <- model.matrix(target ~ var1 + var2 + var3 + var4, data = train)
y <- train$target
lasso_model <- glmnet(x, y, family = "binomial", alpha = 1)
plot(lasso_model)
cv_model <- cv.glmnet(x, y, family = "binomial", alpha = 1)
Coefficients <- coef(cv_model, s = "lambda.min")
# 批量单因素Logistic回归
Uni_log <- function(var_name) {
formula <- as.formula(paste("target ~", var_name))
model <- glm(formula, data = train, family = "binomial")
result <- summary(model)$coefficients
return(result)
}
var.names <- colnames(train)[1:4]
results <- lapply(var.names, Uni_log)
results_df <- ldply(results)
write.csv(results_df, file.path(path, "univariate_logistic_results.csv"))
# 多因素Logistic回归
formula <- as.formula("target ~ var1 + var2 + var3 + var4")
multi_model <- glm(formula, data = train, family = "binomial")
summary_df <- summary(multi_model)$coefficients
write.csv(summary_df, file.path(path, "multivariate_logistic_results.csv"))
# 列线图、模型验证部分
roc_train <- roc(train$target, predict(multi_model, type = "response"))
roc_test <- roc(test$target, predict(multi_model, newdata = test, type = "response"))
plot(roc_train, main = "ROC Curve (Train)", col = "blue")
lines(roc_test, col = "red")
legend("bottomright", legend = c("Train", "Test"), col = c("blue", "red"), lty = 1)
calibration_train <- calibrate(roc_train, method = "boot", B = 100)
calibration_test <- calibrate(roc_test, method = "boot", B = 100)
plot(calibration_train, main = "Calibration Curve (Train)", col = "blue")
lines(calibration_test, col = "red")
legend("bottomright", legend = c("Train", "Test"), col = c("blue", "red"), lty = 1)
dca_train <- ggDCA(roc_train)
dca_test <- ggDCA(roc_test)
plot(dca_train, main = "DCA Curve (Train)", col = "blue")
lines(dca_test, col = "red")
legend("bottomright", legend = c("Train", "Test"), col = c("blue", "red"), lty = 1)
```
请注意,这只是一个示例代码,具体实现可能因为数据集和软件包的不同而有所不同。你需要根据自己的数据集和需求进行适当的修改和调整。
YID:92350698037809482
听雨统计工作室

在现代数据科学和机器学习中,Logistic回归是一种非常重要的分类算法,它在工业和学术界都有广泛的应用。本文将深入探讨Logistic回归的全流程代码,并对其中的关键步骤进行详细说明和分析。
首先,我们需要导入数据。数据的导入是任何数据科学项目的第一步,它决定了后续分析的可行性和准确性。在Logistic回归中,数据可以来自于各种来源,如数据库、文本文件或网络API。我们需要先确保数据的质量和完整性,然后使用合适的方法将数据导入到我们的工作环境中。
接下来,我们需要对数据进行划分。数据划分是为了将整个数据集分为训练集和测试集,以便我们可以使用训练集来训练模型,然后使用测试集来评估模型的性能。划分的比例可以根据实际问题和需求进行调整,通常常见的比例是将数据划分为70%的训练集和30%的测试集。
在数据划分完成后,我们需要生成基线表。基线表是在没有任何特征工程或模型训练的情况下,使用训练集直接预测测试集结果的效果。它可以作为我们后续模型性能评估的基准,以比较模型对比效果的提升。基线表的生成可以通过简单地使用训练集的整体情况对测试集进行预测得到。
接下来,我们将使用LASSO回归来进行特征选择和模型训练。LASSO回归是一种利用L1正则化来约束模型系数的回归方法。它可以通过自动的特征选择,将对模型预测结果影响较小的特征的系数置为零,从而提高模型的泛化能力和解释能力。使用LASSO回归进行特征选择后,我们可以得到一组较为重要的特征,并用这些特征训练模型。
在模型训练完成后,我们可以使用批量单因素logistic和多因素logistic来进行分类预测。批量单因素logistic是一种基于单个特征进行预测的方法,它忽略了不同特征之间的相互影响,只利用各个特征的单独信息进行预测。而多因素logistic则考虑了多个特征之间的相互作用,通过综合考虑各个特征的信息来进行预测。这两种方法在实际应用中可以根据具体问题的需求选择合适的方式。
为了更直观地展示模型性能,我们可以使用列线图、ROC曲线和校准曲线等可视化方法来进行评估和比较。列线图可以将不同特征
【相关代码,程序地址】:http://fansik.cn/698037809482.html






















 655
655











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








