大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
衰老的特点是分子、细胞和器官损伤的逐渐积累,导致生物功能下降,对疾病和死亡的敏感性增加。酿酒酵母(Saccharomyces cerevisiae)作为首个实现完整基因组测序的模式真核细胞,具有繁殖迅速、培养条件简单、基因组小等优点,并且与人类的衰老曲线相似,成为衰老寿命研究的理想模型。基于酵母模型的衰老研究揭示了多种保守的寿命调控通路,并可以揭示衰老与mRNA出核之间的有趣关系。THO复合体是一个保守的多亚基组装体,包括Hpr1、Tho2、Mft1、Thp2和Tex1,与其他因子(如TREX和TREX-2)错综复杂地连接,这些因子对mRNA出核至关重要。Nrd1与衰老之间的直接相关性尚未确定,但Nrd1、Rrp6和RNA监测通路之间的复杂相互作用可能与细胞衰老过程密切相关。总之,衰老与RNA生物发生和出核之间的关系越来越引起人们的兴趣,但其确切机制尚不清楚。
2024年5月20日,韩国汉阳大学Soo Young Cho/Hong-Yeoul Ryu/Seong Hoon Ahn合作研究发现,THO复合体的Hpr1和Tho2两个组分缺失会导致复制寿命(replicative lifespan,RLS)减少,且与一种新的不依赖于Sir2的RLS调控通路相关。相关成果以“Tho2-mediated escort of Nrd1 regulates the expression of aging-related genes”为题发表在《Aging Cell》(IF 7.8 / 1区)期刊上。

标题:Tho2-mediated escort of Nrd1 regulates the expression of aging-related genes(Tho2介导Nrd1调控衰老相关基因表达)
时间:2024-5-20
期刊:Aging Cell
影响因子:IF 7.8 / Q1
技术平台:ChIP-seq、RNA-seq等
研究摘要:
THO复合体对于mRNA共转录成熟和出核至关重要。尽管核糖体组分Rrp6对hpr1Δ或tho2Δ突变体中的转录本螯合产生抗性,但这种螯合缺失未能减轻hpr1Δ中的RLS缺失。hpr1Δ或tho2Δ中的RLS缺失被与Rrp6互作的Nrd1特异性突变体的额外表达所抵消。这种作用依赖于Nrd1与RNA聚合酶II互作,Nrd1是衰老相关基因的转录调节因子,包括核糖体生物发生或RNA代谢基因。Nrd1过表达降低了Tho2依赖性通路中的RLS。而Tho2缺失通过诱导Nrd1在染色质上的任意结合来响应Nrd1过表达效应。全基因组ChIP-seq分析揭示在Tho2缺失后,Nrd1对与衰老相关的翻译相关基因招募增加。本研究结果强调了Tho2介导的Nrd1通过衰老相关基因转录调控在寿命通路调控中的重要性。这项研究提供了对衰老分子机制的新见解,特别是关于RNA代谢和转录调控如何与衰老过程互作的理解,对Tho2-Nrd1轴的进一步研究可能有助于开发延缓衰老和改善健康寿命的策略。
实验方法:
本研究使用酿酒酵母Saccharomyces cerevisiae作为模型生物,通过基因敲除和过表达等方法,结合斑点实验、RLS分析、CLS分析、ChIP-seq和RNA-seq等技术手段进行研究。
结果图形:
(1)THO复合体以不依赖于Sir2方式调控RLS

图1:THO复合体中的Hpr1和Tho2以Sir2非依赖性方式影响RLS。
(a、b)WT和指示突变体的RLS分析。平均寿命显示在括号中。
(2)Hpr1不依赖于核酸外切酶成分Rrp6影响寿命

图2:RRP6缺失可以挽救Rho诱导的细胞中受损的RLS,但不能挽救hpr1Δ细胞。
(a) 用空载体(pCM189)或表达Rho的质粒(pCM189-Ro)转化的WT和rrp6∆菌株进行RLS分析,如图1所示。为了进行生长分析,将所示菌株的十倍稀释液点样在Rho抑制(+Doxy)或Rho诱导(−Doxy)板上,然后在25°C下生长2-3天。
(b) 两种不同遗传背景(BY4741和W303α)中WT和指示突变菌株的RLS分析,如图1所示。
(c) WT和指示突变体的CLS分析。括号中的两个数字表示中位寿命和最长寿命,根据培养物分别达到50%和10%活性天数计算。
(3)Nrd1突变恢复rho诱导的RLS缩短
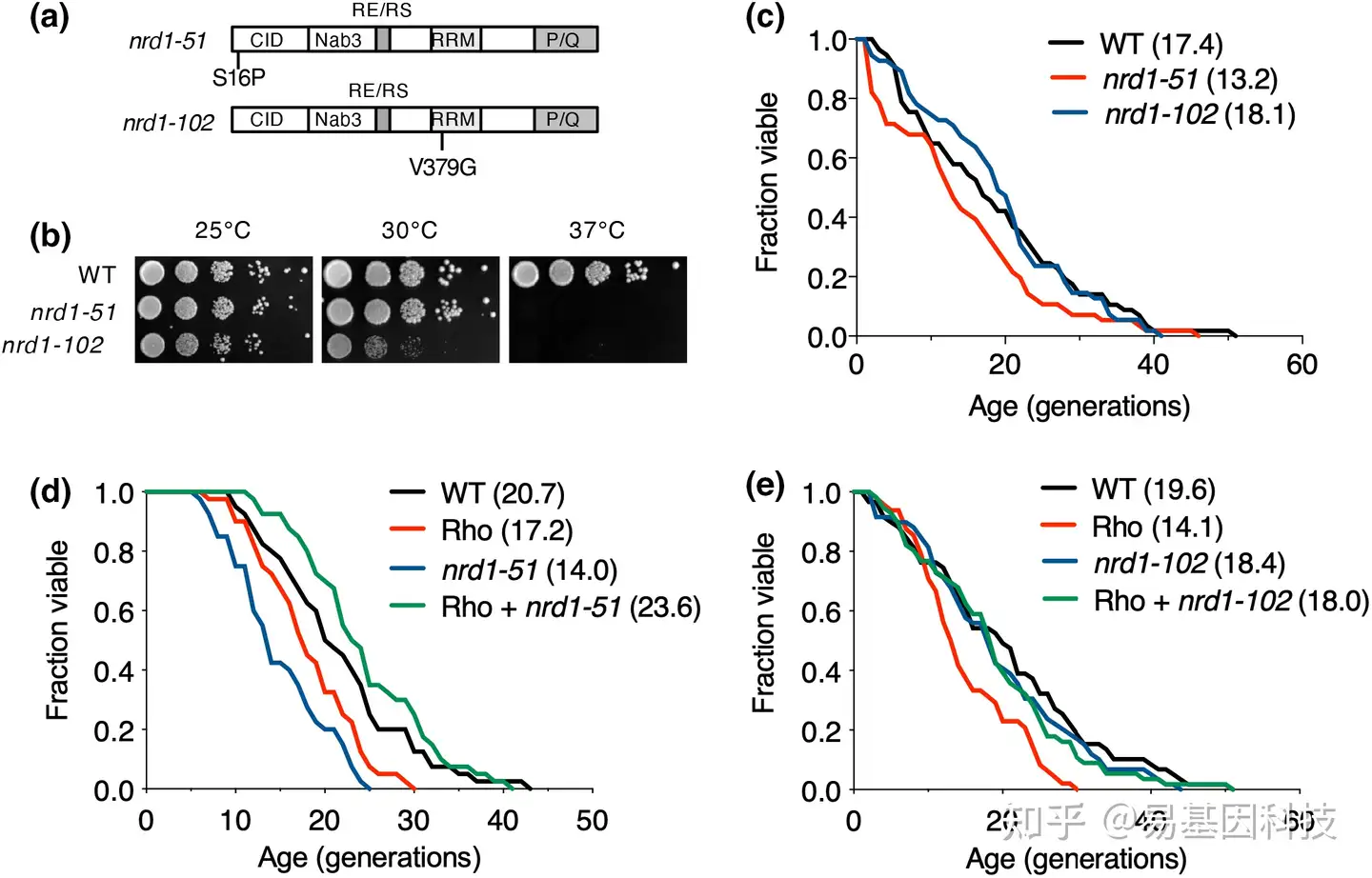
图3:Nrd1突变体中Rho表达可以恢复缩短的复制性寿命(RLS)。
- Nrd1温敏突变体nrd1-51(S16P)和nrd1-102(V379G)的示意图。Nrd1结构域包括RNA聚合酶II CTD互作域(CID)、Nab3互作域(Nab3)、精氨酸-谷氨酰胺和精氨酸-丝氨酸富含区域(RE/RS)、RNA识别结构域(RRM)以及脯氨酸和谷氨酰胺富含区域(P/Q)。
- 在YPD培养基板上对nrd1-51和nrd1-102菌株在25、30和37°C下进行生长分析,如图2a所述。
(c-e) 对野生型(WT)和指示突变体进行RLS分析,这些突变体被转化空载体(c)、pCM189或pCM189-Rho(d, e),如图1所述。
(4)Nrd1的不同截短变体能够缓解tho2Δ细胞中的寿命缺陷。
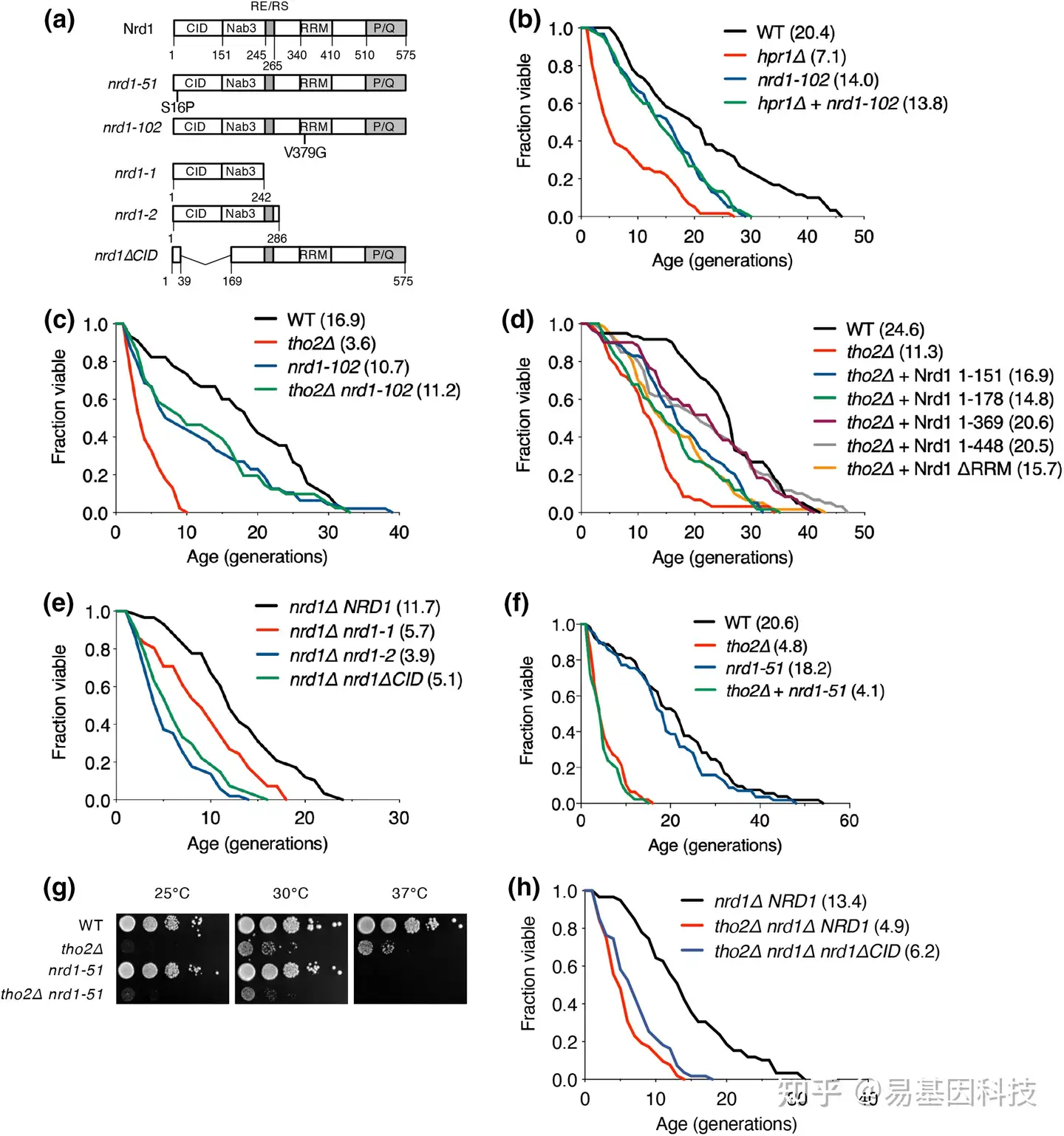
图4:Nrd1蛋白截短体过表达显著恢复了tho2Δ细胞中受损的复制性寿命(RLS)。
- Nrd1点突变体和截短体示意图。
(b、c、f) 如图1所述,对野生型(WT)和指定突变体进行RLS分析。
(d) 如图1所述,对WT或tho2Δ菌株转化空载体(pAG425GPD)或表达指定Nrd1截短变体的质粒后进行RLS分析。
(e、h) 如图1所述,对nrd1Δ或tho2Δ nrd1Δ菌株转化表达NRD1或nrd1ΔCID的质粒后进行RLS分析。(g) 如图2a所述,在25、30和37°C下对(f)中使用的菌株在YPD平板上进行生长分析。
(5)Nrd1通过Tho2依赖性通路调控寿命

图5:Nrd1过表达以Tho2依赖性方式损害RLS。
- PMA1、PYK1和ADH1基因示意图。TATA/启动子(Pro)和ORF分别由黑色和白色框表示。基因下方的条形图显示用于ChIP-qPCR分析的PCR产物的相对位置。
- 在表达TAP标记的Nrd1菌株中使用IgG-琼脂糖珠进行ChIP分析。BY4741(无标签)菌株用作阴性对照。所指示基因的qPCR信号被定量并标准化到内部背景对照和input DNA。使用的引物对在(a)中指示。
- 对WT和tho2Δ菌株转化pAG425GPD或pAG425GPD-NRD1(NRD1-OE)进行RLS分析,如图1所述。
- WT和tho2Δ菌株中Nrd1-TAP的ChIP-seq数据。饼图显示Nrd1峰在WT(左图)和tho2Δ(右图)菌株中的启动子、外显子、下游和远端间隔区的分布。括号中的数字表示检测到的位点数量。
- 在(d)中的Nrd1峰的维恩图。
- 对WT中Nrd1富集基因的GO富集分析点图。
- 在(d)中的ChIP-seq数据的聚类热图。通过K-means聚类,将转录起始位点(TSS)和转录结束位点(TES)处显著富集的Nrd1结合峰分为三个聚类。括号中的数字表示检测到的Nrd1峰数量。
- 在(g)中的ChIP reads密度分布。剖面图显示了每个聚类的平均ChIP信号。
- 在(g)中基因的GO富集分析。条形图表示每个聚类的GO生物过程的倍数富集。
- 综合基因组学浏览器(IGV)截图显示tho2Δ增加(红色)或减少(蓝色)Nrd1招募的水平。从WT中的IP/INPUT峰值比率中减去tho2Δ中的峰值比率,以说明在指定基因中观察到的变化。
(6)与翻译和核转运相关的基因在tho2Δ和衰老细胞中均下调

图6:在酵母细胞中,Tho2蛋白缺失与衰老细胞之间mRNA表达谱比较。
- 火山图显示tho2Δ菌株与野生型(WT)相比差异表达的基因。
- 对先前的RNA-seq实验(Hendrickson等人,2018年)中的衰老细胞基因进行GO富集分析,并在(a)中展示。通过过滤数据(p值≤0.05和|倍数变化|>1),获取了衰老细胞与年轻细胞相比差异表达的基因列表。条形图表示在上调(红色)或下调(蓝色)基因中前11个GO生物过程术语的富集倍数。
- Nrd1 ChIP读取密度分布,显示了与WT相比,tho2Δ菌株中差异表达的基因。对于上调和下调基因的分析(分别是上图和下图),通过过滤数据(p值≤0.05和|倍数变化|>2),分别获取了145个和182个基因。
- 对(c)中上调(红色)或下调(蓝色)基因的GO分析,如(a)中所述。
- IGV(综合基因组学浏览器)截图,显示了tho2Δ改变了Nrd1的招募水平,如图5J所述。
(f–h) 对衰老细胞(Hendrickson等人,2018年)中差异表达基因的Nrd1结合峰进行聚类分析,这些基因在WT和tho2Δ菌株中的Nrd1占据情况。热图(f)、密度剖面图(g)和GO分析(h)是K-means聚类比对 reads位置结果。
- Tho2在调控复制性寿命(RLS)中作用的示意图。tho2缺失未能防止Nrd1的过度招募,导致与翻译相关的基因表达下降,进而导致RLS减少。
(7)参与翻译和RNA加工的基因亚群是Tho2和Nrd1的靶标

图7:衰老细胞中Nrd1结合基因与差异表达基因的比较分析。
(a) 在衰老细胞中,Nrd1结合基因与上调(红色)或下调(蓝色)基因重叠的GO分析,如图6b所示。条形图显示了在ORF或3′UTR中结合位点的Nrd1结合基因的前七个GO生物过程的倍数富集。如果Nrd1在这些区域有结合位点,那么这些基因在衰老细胞中的表达变化可能与Nrd1结合有关。
(b) (a)中显示的Nrd1结合基因的维恩图。中间的表格显示了Nrd1在ORF和3′-UTR或仅在3′-UTR结合的代表性基因,以及它们与相邻基因的长度和距离。

图8:tho2Δ细胞中Nrd1结合基因与差异表达基因的比较。
(a) Nrd1结合基因表达由tho2Δ上调(红色)或下调(蓝色)的前八到九个GO分析,如图7a所示。这些基因在tho2Δ细胞中的表达水平与野生型细胞相比有显著变化,并且这些变化的基因与Nrd1的结合位点有关。
(b) (a)中显示的Nrd1结合基因的Venn图分析,如图7b所示。确定哪些基因仅在tho2Δ细胞中上调或下调,哪些基因同时在tho2Δ和其他条件下也有差异表达,以及哪些基因不受tho2Δ影响。
关于易基因染色质免疫共沉淀测序 (ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
应用方向:
ChIP 用来在空间上和时间上不同蛋白沿基因或基因组定位
- 转录因子和辅因子结合作用
- 复制因子和 DNA 修复蛋白
- 组蛋白修饰和变异组蛋白
技术优势:
- 物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
- 微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
- 方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
技术路线:
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900.

参考文献:
Liu Y, Park JM, Lim S, Duan R, Lee DY, Choi D, Choi DK, Rhie BH, Cho SY, Ryu HY, Ahn SH. Tho2-mediated escort of Nrd1 regulates the expression of aging-related genes. Aging Cell. 2024 May 20:e14203. doi: 10.1111/acel.14203. PubMed PMID: 38769776.
相关阅读:
项目文章 | Nature子刊:ChIP-seq等揭示c-di-AMP与DasR互作以调控细菌生长、发育和抗生素合成
项目文章 | NAR:ChIP-seq等揭示蛋白质酰基化与c-di-GMP协同调控放线菌发育与抗生素合成机制




























 6667
6667

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








