pathview R 包是一个集成 pathway 通路数据与可视化的工具集。它用于把用户的数据映射并渲染到相关的 pathway 通路图上,用户只需要提供基因或者化合物数据(gene or compound data)并指定目标通路(specify the target pathway)即可。
pathview 会产生 native KEGG view 和 Graphviz view 两种 pathway 查看方式,前者以 native KEGG graph (.png) 进行渲染,后者则使用 graphviz layout engine (.pdf)。pathview 作为主程序提供了 downloader, parser, mapper 以及 viewer 四部分功能:自动下载通路图表数据,解析并映射用户数据,最后把 mapped 的数据渲染到通路图上。
Pathview automatically downloads the pathway graph data, parses the data file, maps user data to the pathway, and renders pathway graph with the mapped data.
在 R 命令行下 pathview 安装:
# pathview 依赖包
> source( "http://bioconductor.org/biocLite.R" )
> biocLite(c("Rgraphviz", "png", "KEGGgraph", "org.Hs.eg.db"))
# pathview 安装
> biocLite("pathview")
我们也可以通过 R-forge 的方式安装:
> install.packages("pathview", repos="http://R-Forge.R-project.org")
或者通过下载 pathview 的源码包进行安装,这里不介绍。
利用 pathview 自带的 example 数据(data(package="pathview" 可查看 pathview 包所有的 example 数据)绘制人 hsa04110 通路图:
> library(pathview)
> data(gse16873.d)
> pv.out <- pathview(gene.data = gse16873.d[, 1], pathway.id = "04110", species = "hsa", out.suffix = "gse16873")
当前目录得到 hsa04110.gse16873.png 通路图:
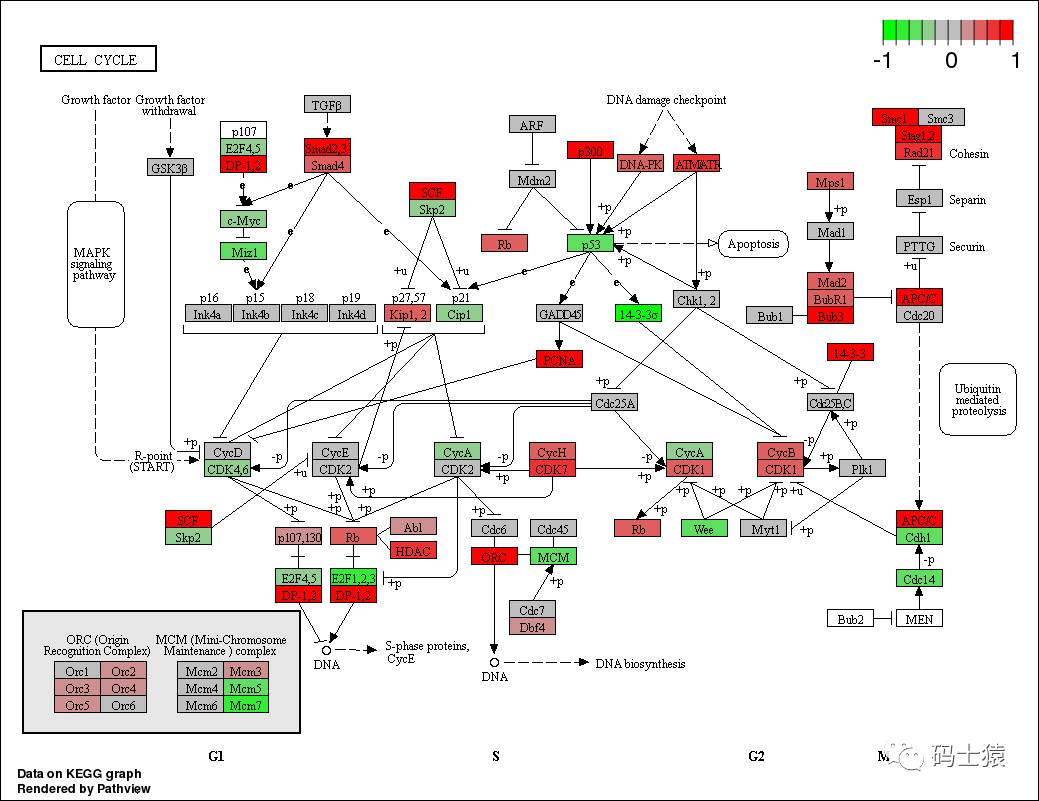
其中 gene.data 接收的是矩阵(或向量)的基因数据,这些数据既可以是数值型(like log2 fold change or absolute expression levels)也可以是基因 id 数据(默认为 entrez 的 gene id,gene.idtype = "entrez"),取决于我们想要得到什么样的可视化结果。
使用 gene IDs 的数据,得到的 hsa04110.geneid.png 如下:
> pv.out <- pathview(gene.data = c("1029"), pathway.id = "04110", species = "hsa", out.suffix = "geneid")
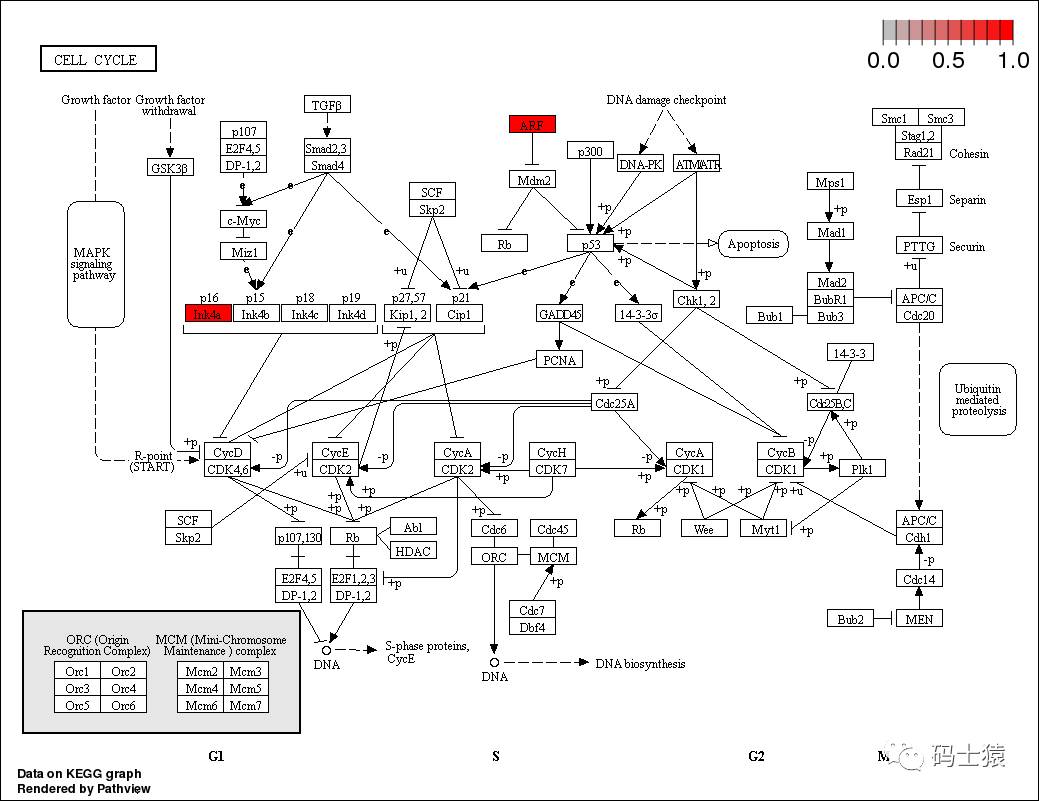
pathview 是一款功能强大的工具集,除了可以展示规范信号通路图外,还支持代谢通路图。利用 pathview 的化合物、基因内置数据,绘制代谢通路图如下:
> data(demo.paths)
> sim.cpd.data = sim.mol.data(mol.type = "cpd", nmol = 3000)
> i <- 3
> print(demo.paths$sel.paths[i])
[1] "00640"
> pv.out <- pathview(gene.data = gse16873.d[, 1], cpd.data = sim.cpd.data, pathway.id = demo.paths$sel.paths[i], species = "hsa", out.suffix = "gse16873.cpd",keys.align = "y", kegg.native = T, key.pos = demo.paths$kpos1[i])
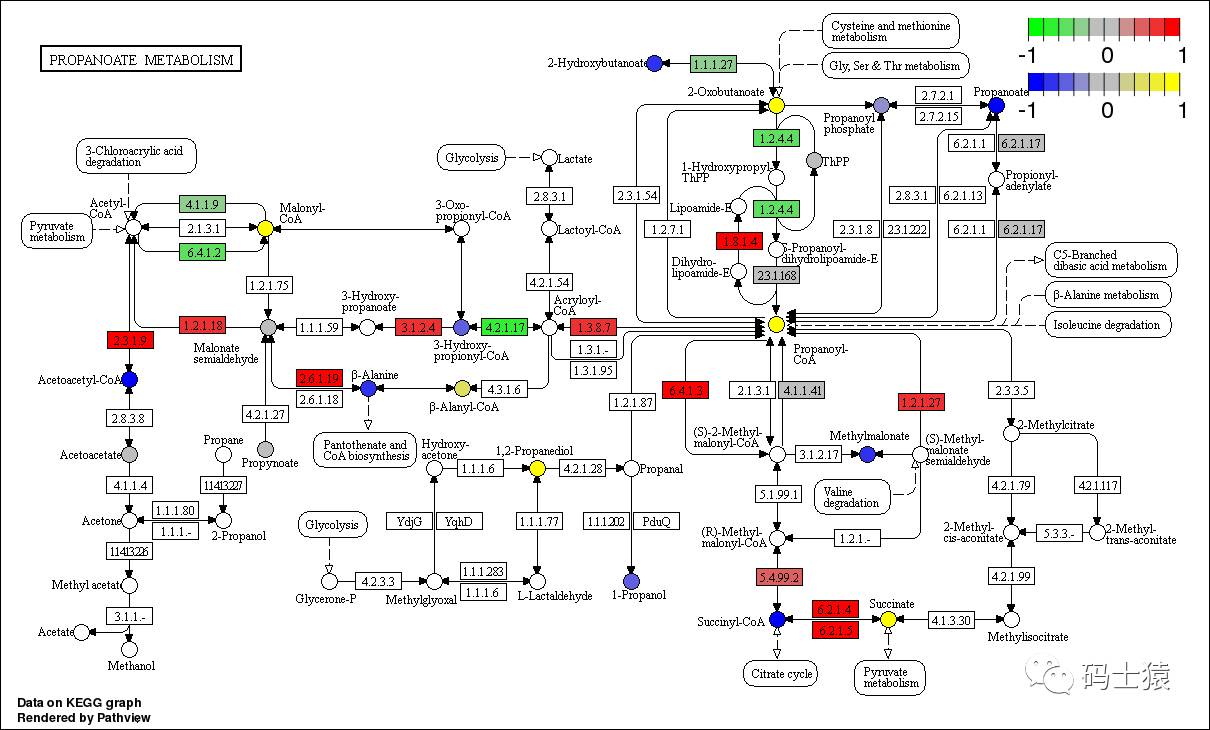
其中,cpd.data(与 gene.data 一样)为 KEGG 的化合物 IDs(KEGG compound IDs),CHEMBL 数据库中超过 20 种 ID 都可以用在这里。gene.data 与 cpd.data 不能同时为空。
ok,就先介绍到这里,更加详细的使用请参考:
http://pathview.r-forge.r-project.org/
https://www.rdocumentation.org/packages/pathview/versions/1.12.0/topics/pathview
本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。




















 901
901











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








