继扩增子、转录组、蛋白组、基因组数据上传教程发布后,组学数据上传教程最后一弹--代谢组数据上传教程来罗,有需要的一定要点赞加收藏!后续上传数据遇到问题可以私聊小编!接下来我们来了解下代谢组学常用数据库MetaboLights以及如何上传实验的组学数据至该数据库。
MetaboLights 是EMBL-EBI数据库的子数据库,该数据库是跨平台、跨物种的代谢组学研究通用、开放存取的数据库,其涵盖了代谢物结构、参考光谱、生物学作用以及来自代谢组实验数据等。MetaboLights是许多领先期刊的推荐代谢组学资料库。
1 账号注册与登录
打开网址(https://www.ebi.ac.uk/metabolights/),注册MetaboLights帐户;
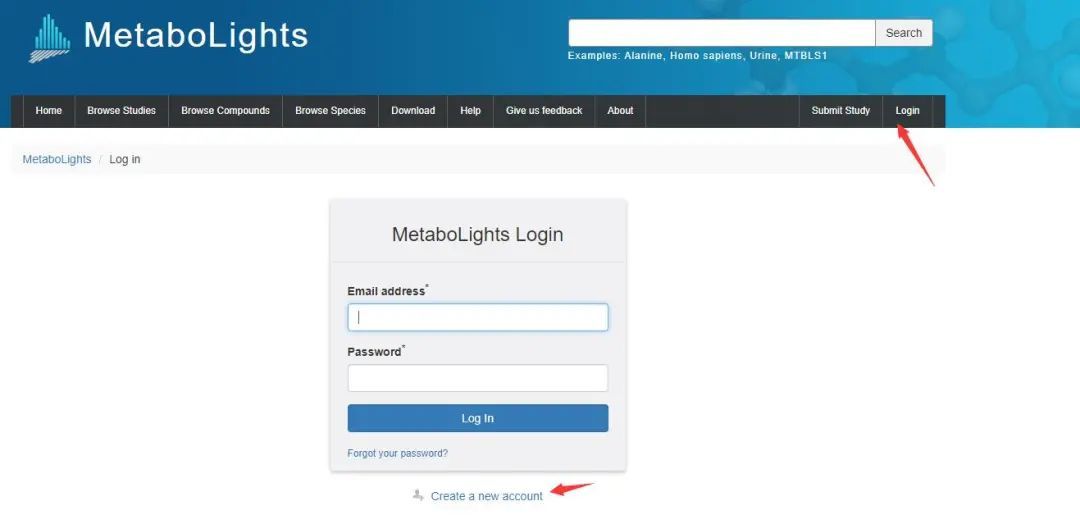
添加图片注释,不超过 140 字(可选)
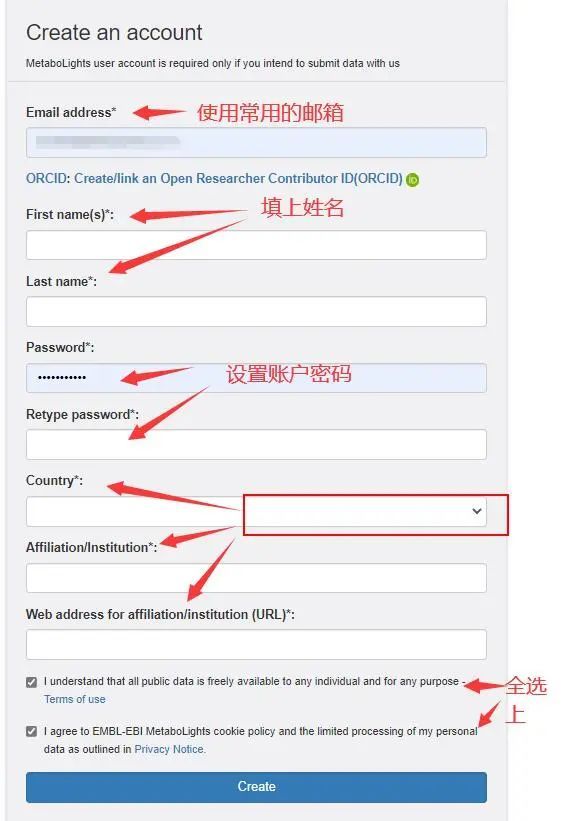
登陆注册邮箱,激活账户,点击邮箱中的链接即可激活账户。点击右上角“Logn in”,输入注册的账号和密码即可登录。
2 数据上传
登录后点击右上角“Submit Study” ,首次提交,选择“Submit a new study”;
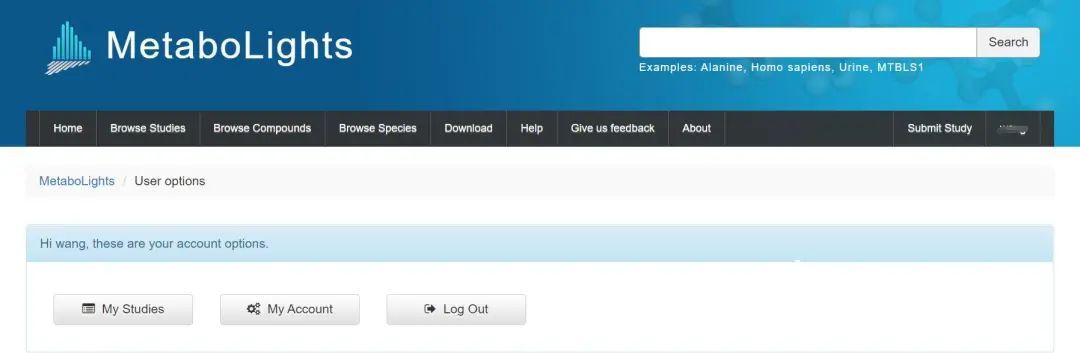
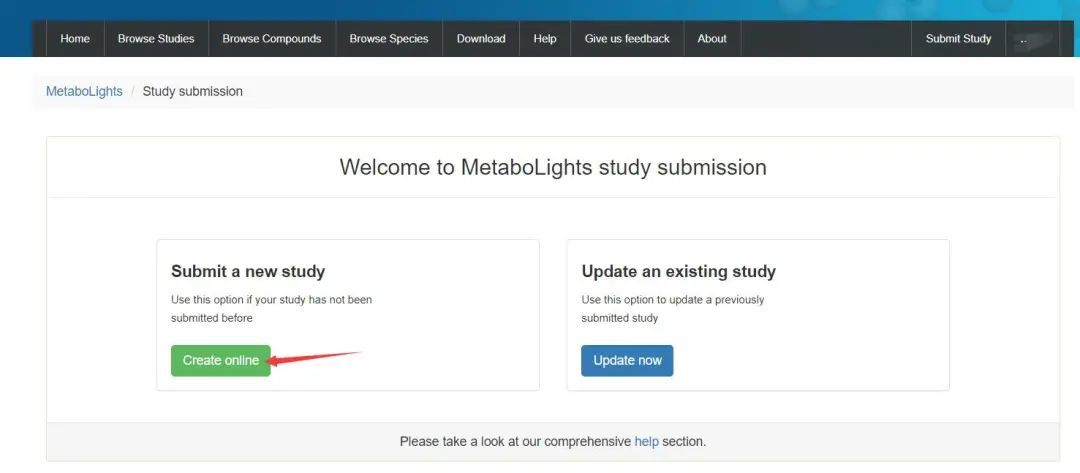
然后点击“Create online”,选择“Create New Study”之后,跳转的页面选择“Let’s get started”。
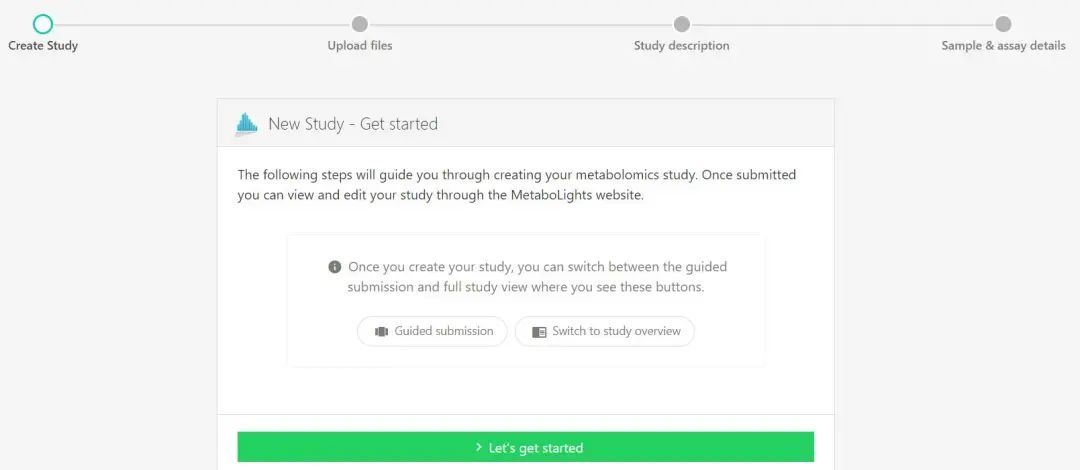
页面跳转后,存在“现在上传原始数据”和“延后上传原始数据”2种选择,可以根据需要进行选择。下面以上传数据为例。选第一个选项,点击next。
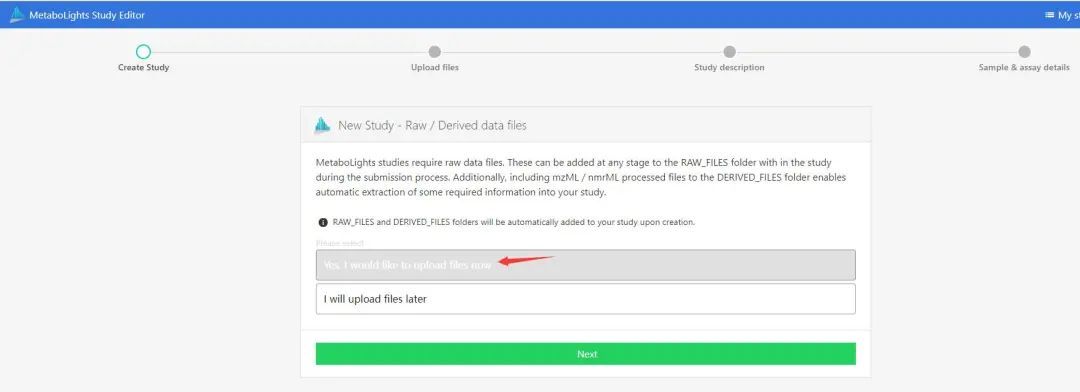
系统会生成一个project,包含项目编号、项目数据引用网址等信息,账号绑定的邮箱也会收到此信息的邮件(论文写作中需要提供此类信息,建议收藏邮件)。接着点击“NEXT”;
接着跳转的页面中显示了2种数据上传方式:Aspera Upload和Private FTP Upload。例如选择Aspera Upload,点击Upload file(之前安装过IBM Aspera Connect 则不需要安装插件,如需安装插件,先点击install plugin下载)。如果仍不知道如何操作,可点击右下角,查看指导视频。
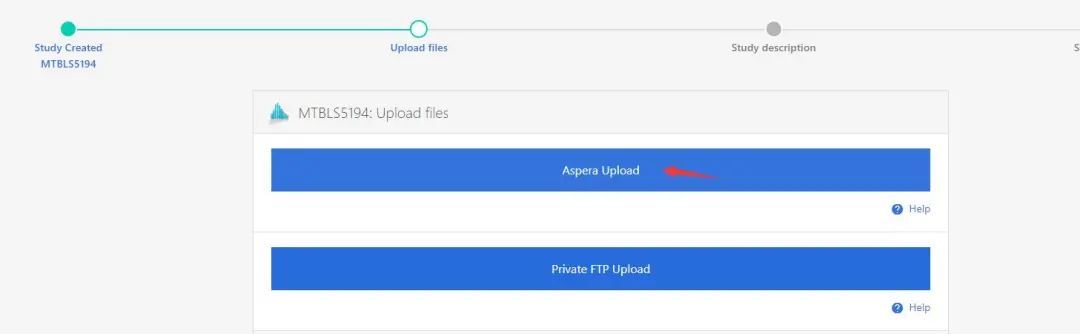
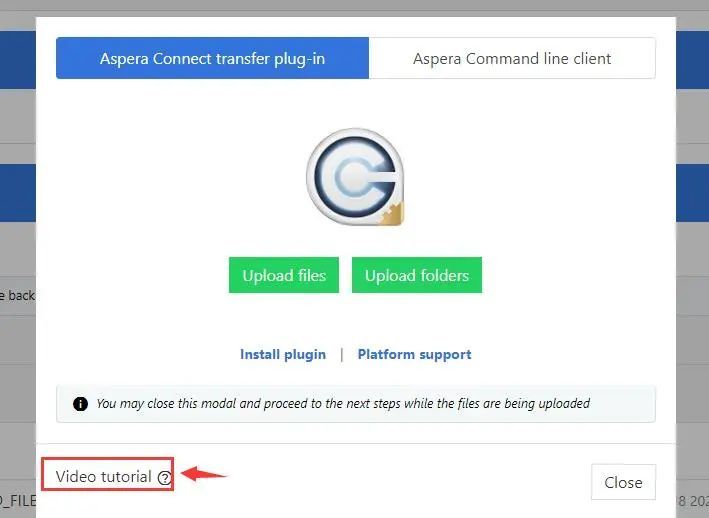
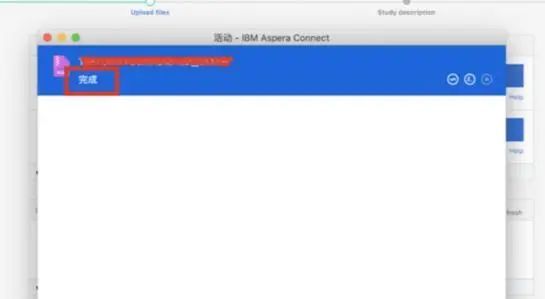
点击Upload file选项后,弹出一个选择文件的窗口,选择需要上传的文件;选中文件后弹出选择窗口,选择允许;点击允许后,上传成功显示界面如下,关闭IBM Aspera Connect窗口。
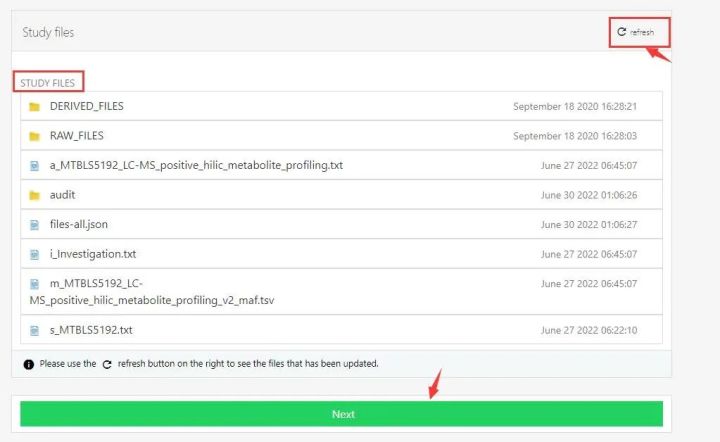
点击refresh按钮,之后显示出上传的文件,如下图红色框所示,表示上传成功,点击NEXT进行下一步;
3 研究项目信息填写
此界面中按照要求填写上项目关键词、摘要及文章发表情况等,点击next进入下个步骤。
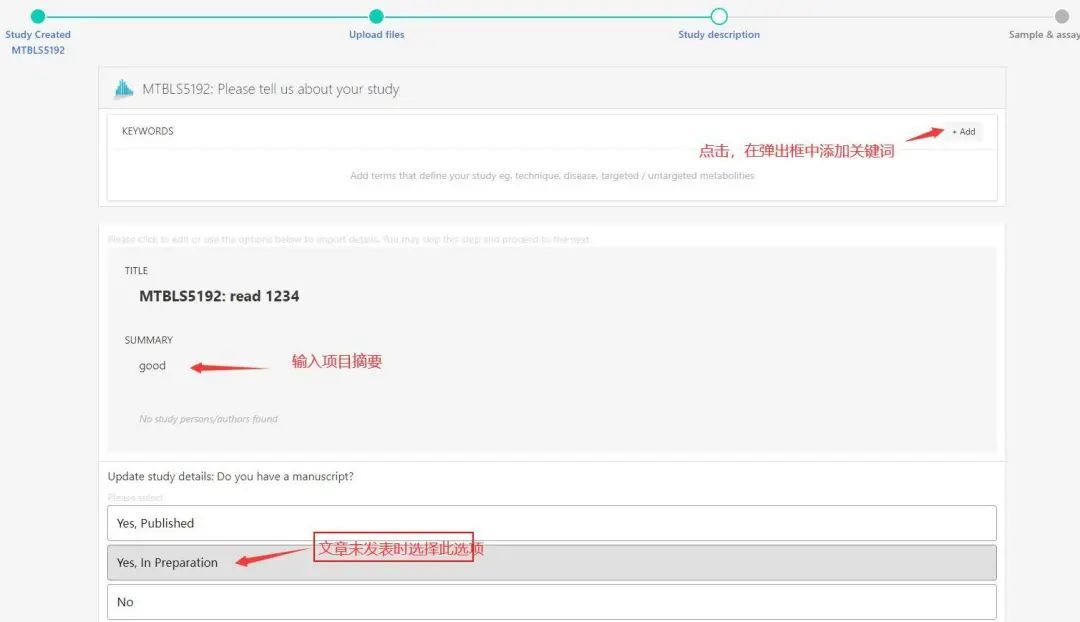
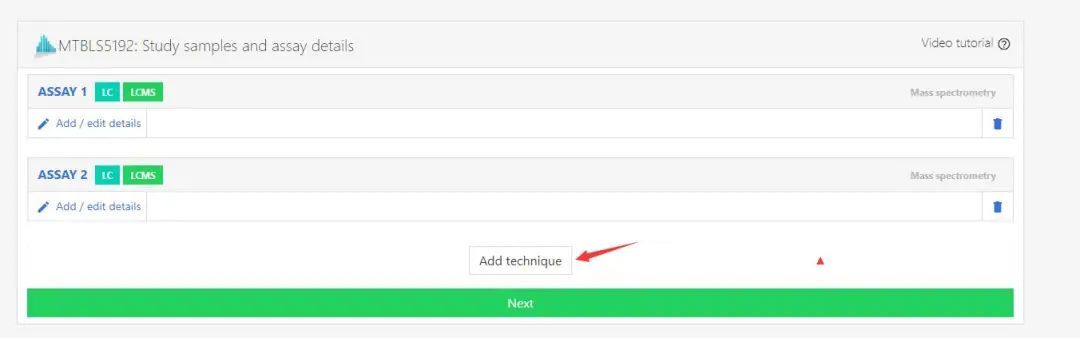
在这个界面点击Add technique,填写信息,例如:
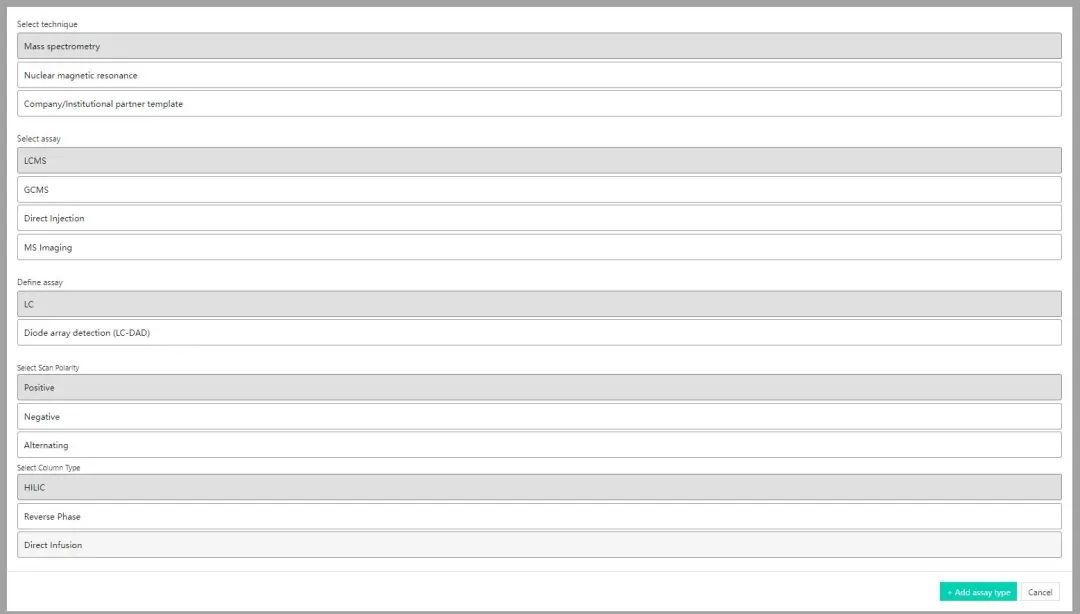
Technique:Mass spectrometry;Assay:LC-MS;Define assay:LC;Scan polarity:Alternating;Column Type:Reverse Phase。之后点击Add assay type;所有的技术添加完后,点击next,弹出框中选择“Confirm”。
页面工具栏:
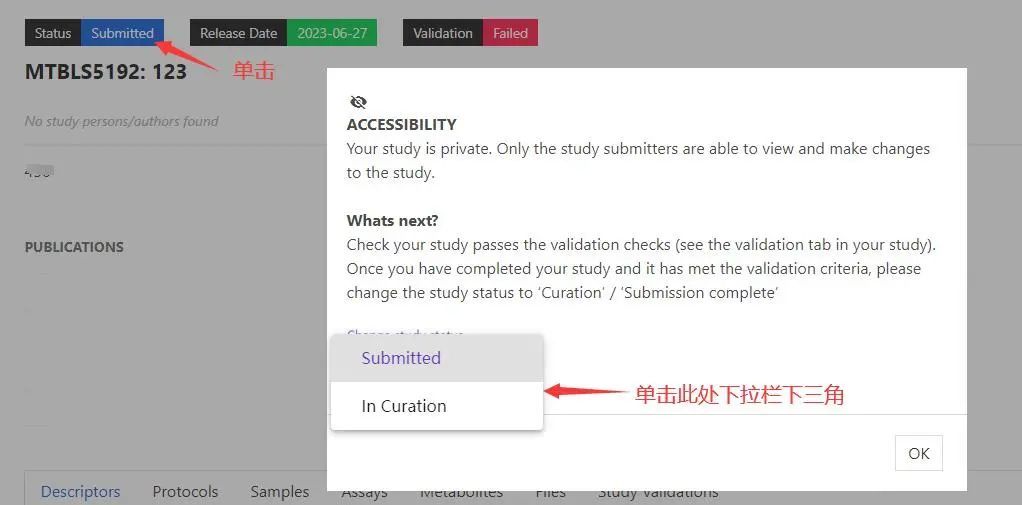
Status:刚创建项目时,页面显示'Submitted'状态,只有按照网站要求准确填写并且最后没有报错(error),你才可以手动把状态改为“In curation”,此后你的项目数据才会呈递到网站审核人手里,由他们进行数据的审核以及补充,只有通过curation,数据最终才能be public
Release Date:这个也是我们自己选择的,你可以根据自己的需要,结合文章online的时间。自行选择数据公开发布的时间点,这个时间咱们是可以更改的。
Validation:网站根据你上传的数据以及你提供的测序参数,审核你数据的完整性、真实性及可靠性,而后给出是否允许你的数据公开的判断,我这里因为还没填写完整,所以现在是'Failed'
Publications: 这里主要涉及该部分数据的文章情况,你要在这里如实填写文章title、key words、author、abstract以及factor信息,值得注意的是factor指该研究的变量(可以是药物类型、药物浓度、基因类型等等),此外,如果你已经有文章稿子了,可以把稿件上传帮助后台审核数据。
之后点击“绿色”按钮,选择数据释放到数据库的时间,点击“OK“,选择”Add Publication”,在新窗口填入论文相应信息,点击“Save“。
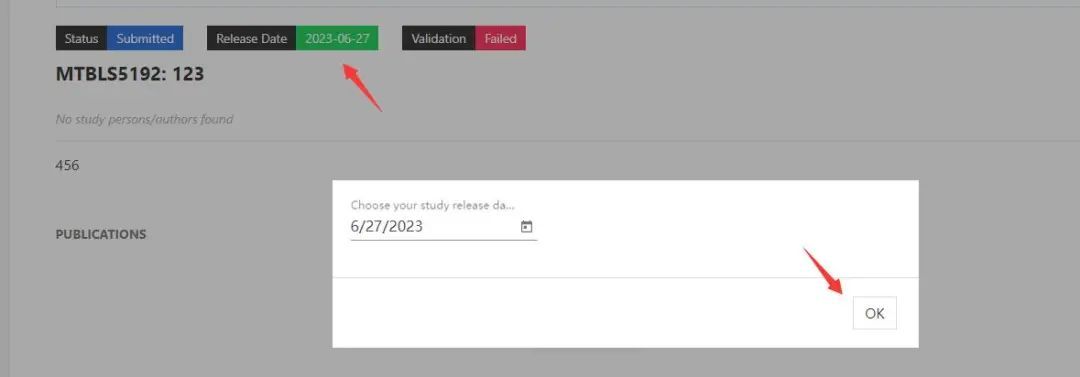
点击“Submitted”,弹出窗口,根据自己项目是否完成决定选择”Submitted”或者“In Curation”,之后点击“OK”;如果信息都填写完整且无误后,手动把状态改为“In curation”,此时你的项目数据才会呈递到网站审核人手里,由他们进行数据的审核以及补充,只有通过curation,数据最终才能发布。
分别在页面中Description,Protocol,Sample,Assays,Metabolites,Files,Study Validation选项卡填入论文相关信息,点击“OK”进行保存;当无误时,”Failed”变成“Success“,表明数据录入成功。如果文章还没发表,PMID、DOI号不填。
Descriptions:把文章的keywords跟factor填上
Protocols:填上实验步骤,每项必须超过两句话,不然就会报错。
Samples:填样品信息
Assay:信息跟前面protocol一样,按照各个步骤填上相应的参数即可
Metabolites:把测序公司提供交付的最代谢物信息表直接上传即可,注意代谢物名称那一列只需要写代谢物名称即可,其他信息删掉
Files:我们可以在这一栏浏览我们上传的所有原始数据文件,以及前面几个条目所填项自动生成的表格
至此数据上传完成,等待网站审核。如果上传的任务信息有二次修改,数据审核周期会有所延长。
5 项目状态查询
点击页面右上角“My Studies”;进入已上传的项目列表页面,点击想要查看的项目编号,进入该项目提交页面。当“Validation“后显示”√“时,表明数据上传成功了。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








