
目录
VISTA-2D 是 NVIDIA 的新基础模型,可以快速准确地执行细胞分割,这项基本任务在细胞成像和空间组学工作流程中至关重要,对所有下游任务的准确性至关重要。
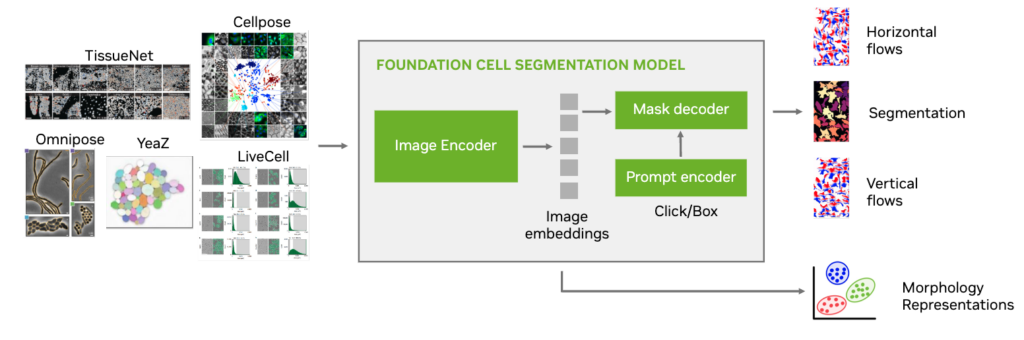
VISTA-2D 模型使用图像编码器创建图像嵌入,然后将其转换为分割蒙版(图 1)。这些嵌入必须包含每个细胞的形态信息。
图 1.VISTA-2D 网络架构
如果可以为每个细胞分割生成嵌入,则可以在所有嵌入上运行聚类,以自动将具有类似形态的细胞分组。
在本文中,我将带您深入了解随附的 Jupyter Notebook,以展示如何使用这些工具首先分割细胞并使用 VISTA-2D 提取其空间特征,然后使用 RAPIDS。这将创建一个自动化流程来快速分类细胞类型。
预备知识
要学习本教程,您需要以下资源:
- 基本熟悉 Python、Jupyter 和 Docker
- Docker 版本 19.03 及更高版本
启动 notebook
此 Jupyter Notebook 的代码位于 /clara-parabricks-workflows/vista2d_rapids_clustering GitHub 库中,并在 NVIDIA 的 PyTorch Docker 容器 内运行。此 Notebook 使用该容器的 24:03-py3 标签构建。使用以下命令运行容器:
docker run --rm -it \
-v /path/to/this/repo/:/workspace \
-p 8888:8888 \
--gpus all \
nvcr.io/nvidia/pytorch:24.03-py3 \
/bin/bash
此命令会启动以下操作:
- 启动 Docker 容器。
- 将资源库的文件夹安装到容器中。
- 将主机上的端口 8888 映射到 Docker 中的端口 8888。
- 将所有可用的 GPU 分配给容器。
- 启动 PyTorch 容器。
- 返回终端。
接下来,您需要一些其他的 Python 包,这些包可以在 requirements.txt 中找到。
fastremap tifffile monai plotly
这些包主要用于辅助函数和绘图,这在本文稍后部分将更加明显。目前,它们可以在 Docker 容器之上安装。
pip install -r requirements.txt
接下来,启动 notebook:
jupyter notebook
现在,notebook 服务器正在运行,并且可以使用 web 浏览器在运行服务器的同一台机器上或在单独的机器上访问 notebook。
在浏览器中,输入服务器所在计算机的 IP 地址,然后输入端口 8888:
<ip-address>/8888
现在 Notebook 已经准备就绪,可以运行了。有关更多信息,请参阅 GitHub 资源库。
使用 VISTA-2D 进行细胞分割和特征提取
本 Notebook 的上半部分结合使用 Live Cell 数据与 VISTA-2D 分割图像中的细胞,并使用 VISTA-2D 模型本身的编码层提取特征。
首先,加载 VISTA-2D 模型检查点,因为本笔记本不专注于训练模型,而是将其用于特征提取的目的。
model_ckpt = "cell_vista_segmentation/results/model.pt"
接下来,加载辅助函数,以避免主笔记本变得太冗长。
from segmentation import segment_cells, plot_segmentation, feature_extract
下几节将详细介绍这些辅助函数的作用。它们均可在segmentation.py中找到。
segment_cells
此函数获取细胞图像,并从头到尾通过 VISTA-2D 运行。这会生成另外两张图像,一张用于完整分割,另一张用于标记图像中发现的细胞数(在 Notebook 中称为pred_mask),从 1 到细胞数之间的每个细胞。这使得细胞能够单独索引,以便向下行提取特征。
img_path="example_livecell_image.tif" patch, segmentation, pred_mask = segment_cells(img_path, model_ckpt)
plot_segmentation
此函数接收segment_cells的输出并显示图像,以便在分割和预测蒙版中直观地验证其准确性。图 2 展示了使用 notebook 中提供的单元图像输出的示例。
plot_segmentation(patch, segmentation, pred_mask)
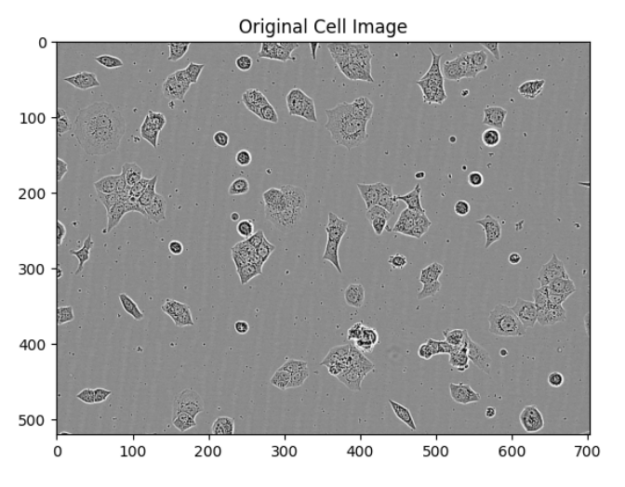
a) 原始细胞图像
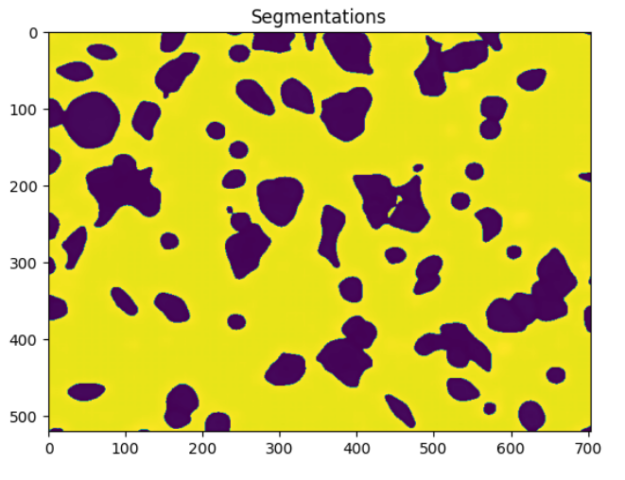
b) 分割
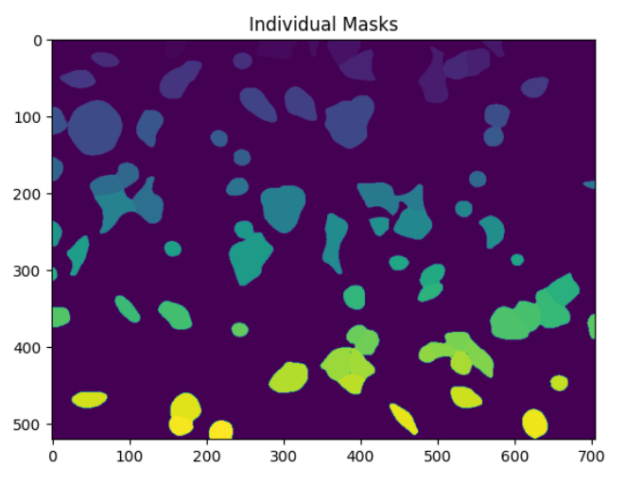
c) 单个蒙版 图 2.VISTA-2D 分割结果
Alt:三张图像显示 VISTA-2D 分割的结果:原始细胞图像,从背景中分割出所有细胞,以及每个细胞的单个蒙版。
feature_extract
此函数获取每个单独的单元分割,并生成特征向量。每个单元都包含在一个裁剪的方形遮罩中,以适应该单元和周围的任何背景。它使用 VISTA-2D 模型的前半部分作为编码器来生成这些特征向量。
我们的想法是,生成的向量包含细胞分割所需的所有信息,因此还必须包含有关每个细胞形态的信息。这些信息作为向量,可以轻松插入聚类算法中。形态相似的细胞应具有类似的特征向量,并被分配给类似的集群。
cell_features = feature_extract(pred_mask, patch, model_ckpt)
这将生成一个包含 num_cells 行和 1024 列的矩阵,该矩阵的列数是每个单元的编码向量的长度。
现在您已经拥有每个单元的特征向量,是时候使用 RAPIDS 通过聚类算法运行它们了。
使用 RAPIDS 进行聚类
RAPIDS 是一个 GPU 加速的机器学习库,具有适用于常用 Python 数据科学库(例如 pandas 和 scikit-learn)的匹配 API。在此 Notebook 中,您仅使用 RAPIDS 的特征降维和聚类部分,但还有更多可用产品。
from cuml import TruncatedSVD, DBSCAN
TruncatedSVD
您从 VISTA-2D 获得的特征向量长度为 1024。然而,考虑到图像中只有大约 80 个单元,因此使用如此多特征来制作集群是不合理的。
您可以使用降维算法来减少这些嵌入的长度,同时最大限度地减少丢失的信息。在此笔记本中,使用TruncatedSVD算法将维度从1024缩减到3。这还可以更轻松地绘制集群,因为您可以在 3D 空间中可视化集群。
dim_red_model = TruncatedSVD(n_components=3) X = dim_red_model.fit_transform(cell_features)
这将生成新的特征向量矩阵 X,现在其大小为[num_cells, 3],而不是 cell_features 中大小为[num_cells, 1024]的原始向量。
DBSCAN
RAPIDS 中提供了许多集群算法。对于此 Notebook,我选择了 DBSCAN。在这里,您将 eps(两个点之间的最大距离)设置为 0.003,并将允许构成集群的最小样本数设置为 2。
model = DBSCAN(eps=0.003, min_samples=2) labels = model.fit_predict(X)
现在,运行 fit_predict 会为图像中的每个单元生成集群标签。如果将标签列表转换为标签字典,则更容易看到哪些单元已被分配给哪些集群。
# Background is 0, so cell IDs start at 1
labels_dict = {x:np.add(np.where(labels==x),1) for x in np.unique(labels)}
# Label -1 means "data was too noisy" so we remove it
labels_dict.pop(-1)
labels_dict
最后,您可以使用 Plotly 配置 3D 交互式图形,以显示每个单元的聚类位置。
import plotly
data = []
for l in labels_dict.keys():
cluster_indices = labels_dict[l][0]-1
# Configure the trace
trace = go.Scatter3d(
x=X[cluster_indices,0],
y=X[cluster_indices,1],
z=X[cluster_indices,2],
name="Cluster "+str(l),
mode='markers',
marker={
'size': 10,
'opacity': 0.8,
}
)
data.append(trace)
# Configure the layout
layout = go.Layout(
margin={'l': 0, 'r': 0, 'b': 0, 't': 0}
)
plot_figure = go.Figure(data=data, layout=layout)
# Render the plot
plotly.offline.iplot(plot_figure)
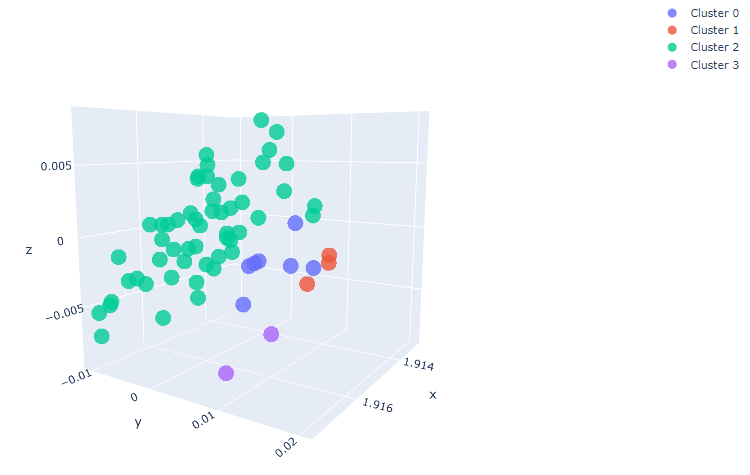
图 3. 由聚类特征向量图生成的交互式三维图





















 66
66

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








