目录:
1.DNA---RNA---protein互相转换

# -*- coding: utf-8 -*-
原创公众号pythonEducation
'''
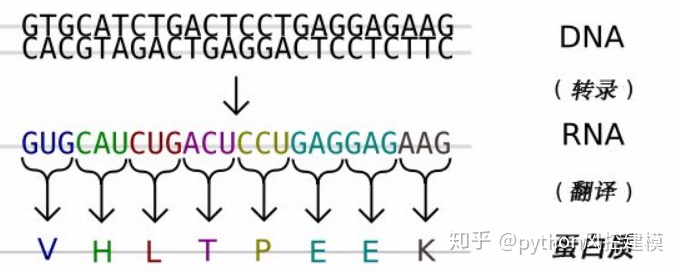
实际的生物学上的转录过程是将模板链反向互补(TCAG → CUGA)生成mRNA。但是,
在Biopython和生物信息学领域,我们通常会直接利用编码链,因为我们可以通过 T → U的转换获得mRNA。
'''
from Bio.Seq import Seq
from Bio.Alphabet import IUPAC
from Bio.SeqUtils import GC
from Bio.Alphabet import generic_alphabet
#生成序列
my_seq = Seq("AGTACACTGGT")
'''
my_seq
Out[5]: Seq('AGTACACTGGT', Alphabet())
print my_seq
AGTACACTGGT
'''
my_seq.alphabet
'''
Out[7]: Alphabet()
'''
#互补
my_seq.complement()
'''
Out[8]: Seq('TCATGTGACCA', Alphabet())
'''
#反向互补
my_seq.reverse_complement()
'''
Out[9]: Seq('ACCAGTGTACT', Alphabet())
'''
my_seq = Seq('GATCGATGGGCCTATATAGGATCGAAAATCGC', IUPAC.unambiguous_dna)
#计算G和C的概率
GC(my_seq)
'''
Out[13]: 46.875
'''
str(my_seq)
'''
Out[14]: 'GATCGATGGGCCTATATAGGATCGAAAATCGC'
'''
protein_seq = Seq("EVRNAK", IUPAC.protein)
dna_seq = Seq("ACGT", IUPAC.unambiguous_dna)
#不同性质序列叠加会报错
#protein_seq + dna_seq
#转化为通用的字母表后,不同序列可以叠加
protein_seq.alphabet = generic_alphabet
dna_seq.alphabet = generic_alphabet
protein_seq + dna_seq
'''Out[27]: Seq('EVRNAKACGT', Alphabet())'''
#创建DNA序列的 编码链
coding_dna = Seq("ATGGCCATTGTAATGGGCCGCTGAAAGGGTGCCCGATAG", IUPAC.unambiguous_dna)
#模板链(反向互补)
template_dna = coding_dna.reverse_complement()
'''template_dna
Seq('CTATCGGGCACCCTTTCAGCGGCCCATTACAATGGCCAT', IUPACUnambiguousDNA())'''
# transcribe 转录方法将编码链转录成对应的mRNA
messenger_rna = coding_dna.transcribe()
'''
Seq('AUGGCCAUUGUAAUGGGCCGCUGAAAGGGUGCCCGAUAG', IUPACUnambiguousRNA())
'''
#Seq 对象还包含了从mRNA逆向转录为DNA编码链的方法。同样,这仅仅是从U → T的替代并伴随着字母表的变化
messenger_rna.back_transcribe()
'''
Out[30]: Seq('ATGGCCATTGTAATGGGCCGCTGAAAGGGTGCCCGATAG', IUPACUnambiguousDNA())
'''
#继续使用在转录那个小节中的例子,现在让我们将这个mRNA翻译成相对应的 蛋白质序列
#rna编码和DNA编码都可以翻译成对应蛋白质
protein_seq1=messenger_rna.translate()
'''
Seq('MAIVMGR*KGAR*', HasStopCodon(IUPACProtein(), '*'))
'''
#直接从DNA翻译为蛋白质
protein_seq2=coding_dna.translate()
#DNA重编程,使用mutable函数
my_seq = Seq("GCCATTGTAATGGGCCGCTGAAAGGGTGCCCGA", IUPAC.unambiguous_dna)
mutable_seq = my_seq.tomutable()
'''
Out[38]: MutableSeq('GCCATTGTAATGGGCCGCTGAAAGGGTGCCCGA', IUPACUnambiguousDNA())
'''
#更改DNA编码
mutable_seq[5] = "C"
'''
MutableSeq('GCCATCGTAATGGGCCGCTGAAAGGGTGCCCGA', IUPACUnambiguousDNA())
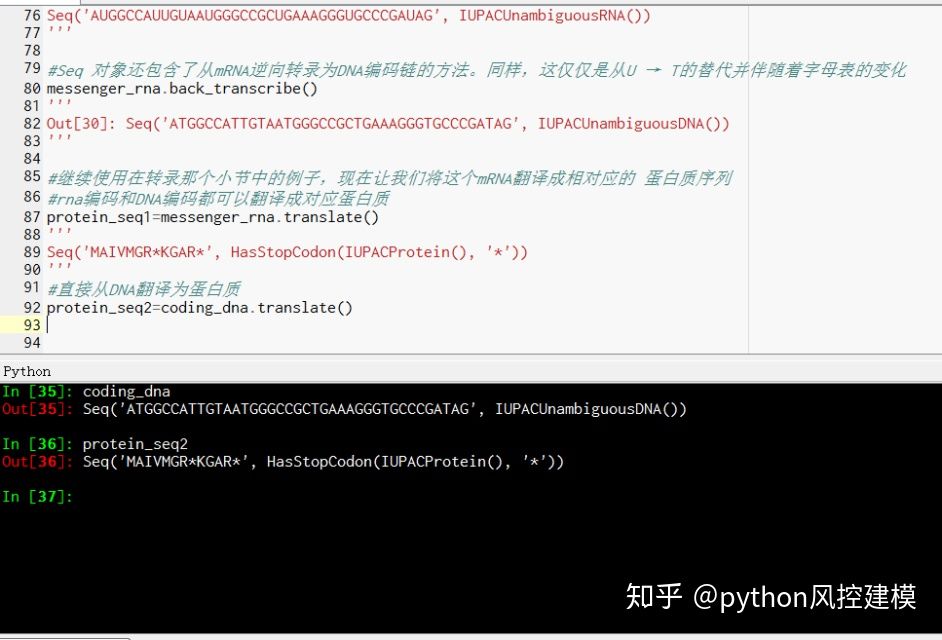
'''
mutable_seq.remove("T")
mutable_seq.reverse()
mutable_seq欢迎各位同学学习python机器学习生物信息学,链接地址为https://edu.csdn.net/combo/detail/1930
























 1672
1672











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










