1-介绍
1、AnnData 不是一个单一的表格,而是一个包含多个表格和矩阵的复杂数据结构,用于处理和存储单细胞基因组数据。
AnnData对象包含以下主要部分:
- var:基因的元数据(Pandas DataFrame格式),包含每个基因的属性信息。例如每个基因的ID、名称、染色体位置等。可以将其看作是基因(列)的属性数据。
- obsp:细胞之间的对称关系矩阵(通常是稀疏矩阵)。
- obsm:细胞的多维数据矩阵(Pandas DataFrame格式),如降维结果。
- X:核心数据矩阵,通常是细胞(行)和基因(列)的表达计数矩阵。
- obs:细胞的元数据(Pandas DataFrame格式),包含每个细胞的属性信息。每个细胞的类型、状态、处理条件。可以将其看作是细胞(行)的属性数据。
- layers:不同类型的计数矩阵(字典格式),如归一化后的计数矩阵、原始计数矩阵等。
- varm:基因的多维数据矩阵(Pandas DataFrame格式)。
- varp:基因之间的对称关系矩阵(通常是稀疏矩阵)。
- uns:未命名数据(字典格式),存储其他任何附加信息。
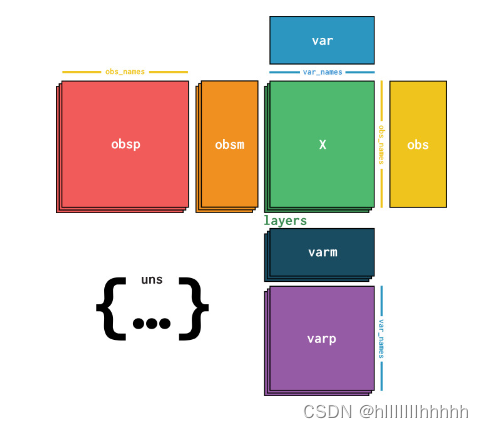
重点:AnnData数据格式,每个部分代表什么;单组学、多组学
2-预处理和可视化
2-1质量控制
1、由于测序深度的限制,受限于基因自身的表达量,我们会发现单细胞测序数据是一个有很多“0”值的矩阵,我们称这种现象为“drop-out”,这些零一方面可能是测序深度不够导致基因没有捕获到,另一方面可能是这些基因本身没有表达。
2、质量控制
第一步是过滤低质量的细胞。当细胞检测到的基因数量较少、计数深度较低且线粒体计数较高时,细胞膜可能会破裂,这表明细胞正在死亡。
第二步双细胞的过滤。双细胞被定义为在相同的细胞条形码(barcode)下进行测序的两个细胞,例如,如果它们被捕获在同一个液滴(droplet)中。这也是为什么我们一直使用barcode而不是cells的原因。双细胞由同型(homotypic)与异型(heterotypic)所构成
第三步环境 RNA 的校正。注:本节可能会导致计数矩阵不为整数,使得部分包失效,所以该分析除非你很明确环境RNA的干扰十分严重,不然我认为是可以被跳过的。
2-2 归一化
思考
-
1、我们在进行移位对数分析的时候,
sc.pp.normalize_total(adata, target_sum=None, inplace=False)中的target_sum使用了默认值,在seurat中默认值是10,000,在一些教程中设定为1,000,000,我们虽然对这个值的意义进行了简单介绍,但你认为不同的值背后的含义是什么?
不同的
target_sum值的意义在于:
基因表达量的比较:较小的值(如10,000)会使较低表达基因的差异更显著,而较大的值(如1,000,000)则更能保留高表达基因的细节。
计算复杂性:较大的值需要更多的计算资源和存储空间。
适应性:选择合适的
target_sum值应该基于具体的研究需求和数据特性。
-
2、我们为什么会使用皮尔森残差来计算归一化值,相对于移位对数而言有什么更好的地方?
我们观察到,scRNA-seq数据中的细胞间变异包括了生物异质性以及技术效应。而移位计数并不能很好的排除两种不同变异来源的混淆误差。
皮尔森近似残差利用了“正则化负二项式回归”的皮尔森残差来计算数据中潜在的技术噪音,将计数深度添加为广义线性模型中的协变量,而在不同的归一化方法的测试中,皮尔森残差法可以消除计数效应带来的误差,并且保留了数据集中的细胞异质性
-
3、你可以找出别的归一化方法,并比较其与移位对数,皮尔森残差的好坏吗?
归一化方法比较
- 移位对数归一化
移位对数归一化是通过对数变换来减小数据中的极端值影响,使得数据变异性更加稳定。这种方法快速高效,适用于大多数常见的scRNA-seq数据,但在处理高变异性数据时可能效果有限。
- 皮尔森残差归一化
皮尔森残差归一化通过计算实际值与预期值之间的残差来标准化数据,更好地处理稀疏数据和零值,对高表达基因的变异性处理也更好。这种方法能够更有效地减少数据中的过度离散性。
- SCRAN
SCRAN方法通过对细胞进行聚类,计算归一化因子,更好地考虑细胞群体的异质性,适用于大规模数据集,能够更好地处理过度离散性。
- SCTransform
SCTransform基于广义线性模型,通过负二项分布对每个基因进行拟合,然后标准化残差,对不同测序深度和技术变异处理效果较好,能够有效减少数据中的过度离散性。
综合比较
- 简单性:移位对数归一化方法最为简单,适用于大多数常见的数据集。
- 处理稀疏数据:皮尔森残差和SCTransform方法对稀疏数据的处理效果较好。
- 计算资源:移位对数归一化所需计算资源最少,SCRAN和SCTransform则需要更多的计算资源。
- 适应性:SCRAN和SCTransform对数据的适应性更广,能够处理不同类型和规模的数据集。
以上,就是本章所要介绍的归一化内容,通过benchmark的测试,我们发现移位对数适用于大多数任务。但是如果我们的分析目标是寻找稀有细胞的时候,可以考虑采用皮尔森残差法来进行归一化。
重点内容
1、在前面的教程中,我们从数据集中删除了低质量的细胞,包括计数较差以及双细胞,并将数据存放在anndata文件中。
由于单细胞测序技术的限制,我们在样本中获得RNA的时候,经过了分子捕获,逆转录还有测序。这些步骤会影响同一种细胞的细胞间的测序计数深度的变异性,故单细胞测序数据中的细胞间差异可能会包含了这部分测序误差,等价于计数矩阵中包含了变化很大的方差项。但在目前的统计方法中,绝大部分模型都预先假定了数据具有相同的方差结构。
“归一化”的预处理步骤旨在通过将“UMI的方差”缩放到指定范围,来调整数据集中的原始UMI计数以实现模型建模。而在真实的单细胞分析中,有不同的归一化方法以解决不同的分析问题。但经验发现,移位对数在大部分数据中的表现良好,这在2023年4月的Nature Method上的基准测试中有提到。
2、两种不同的归一化技术:移位对数变换和皮尔逊残差的解析近似。
移位对数有利于稳定方差,以利于后续降维和差异表达基因的识别。
皮尔森近似残差可以保留生物学差异,并鉴定稀有细胞类型。
1)移位对数归一化:处理单细胞RNA测序(scRNA-seq)数据的方法,目的是使得不同细胞之间的基因表达水平更加可比。
你有一堆装满豆子的罐子,每个罐子的豆子数量不同。有的罐子装得多,有的罐子装得少。为了比较每个罐子的豆子数量,我们先需要让所有罐子的豆子数量变得差不多。然后,我们再去看每个罐子里面各种颜色的豆子分别有多少。
步骤1:调整罐子里的豆子数量——用不同的尺寸因子放缩
步骤2:对数变换——让数量差异变得更小,更加平滑

为什么要做移位对数归一化?
更公平的比较:原始数据中的细胞可能因为不同的测序深度导致总的基因表达量差异很大。归一化处理后,这种差异被消除了,我们可以更公平地比较不同细胞的基因表达水平。
减少数据噪音:对数变换可以减小数据中的极端值影响,使得数据更加平滑,更容易识别出有意义的信号。
后续分析的基础:归一化后的数据更适合进行后续的统计分析,如主成分分析(PCA)和差异表达分析。
2)皮尔森近似残差
问题背景
想象你正在进行一项调查,记录了每个人的苹果数量。但每个人带来的苹果数量不仅受他们自己实际拥有的苹果数量影响,还受测量误差影响。例如,有的人可能多带了一些,有的人可能少带了一些。这些误差使得你难以准确比较每个人之间的实际差异。
传统方法的问题
之前的移位对数归一化方法就像是对每个人的苹果数量进行一个统一的调整,减小了测量误差的影响,但它并不能完全区分开实际拥有的苹果数量和测量误差。
皮尔森近似残差归一化的解决方案
皮尔森近似残差归一化方法就像是你请来了一个统计学专家,帮你更准确地调整每个人的苹果数量,使得这些调整后的数据更能反映每个人实际拥有的苹果数量,同时消除测量误差的影响。
步骤1:预测每个人应该有多少苹果
首先,这个专家根据每个人的具体情况(例如,他们的年龄、身高等信息)预测了每个人“应该”有多少苹果。这就像是给每个人预期的苹果数量。
步骤2:计算差异
然后,专家计算了每个人实际带来的苹果数量和预期苹果数量之间的差异。如果某人实际带来的苹果比预期的多,那








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 4003
4003

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








