GraphBAN:基于图神经网络的化合物 - 蛋白质相互作用预测新范式
榴莲忘返 2014
供稿 | 柠檬青年
审稿 | 吉星
当 AI 遇见药物发现
化合物与靶蛋白相互作用(CPI)预测是药物研发的重要基石。Nature 子刊最新发表的 GraphBAN 框架,通过创新的图神经网络架构创新了传统方法的局限,为新型药物发现带来创新性进展。
核心创新点
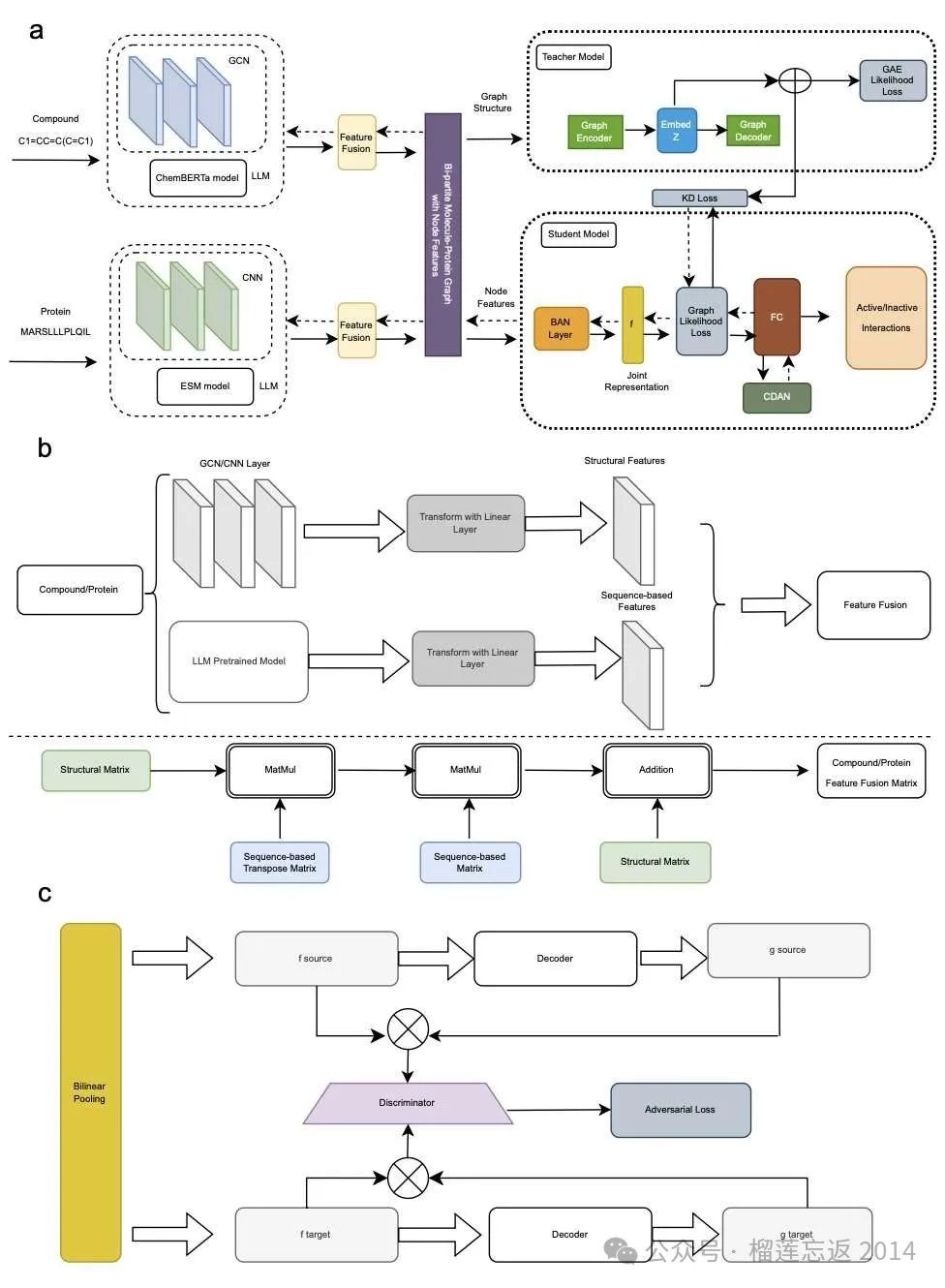
该模型创造性构建了教师 - 学生双模块学习框架:教师模块专注挖掘分子网络拓扑特征,学生模块则精研节点属性表征,二者协同提升预测精度。通过知识蒸馏技术,学生模型能有效继承教师模型对复杂分子互作网络的理解能力。
创新性应用价值
区别于传统方法受限于已知分子互作数据,GraphBAN 展现出强大的外推预测能力——即使面对训练集从未出现的新化合物或新靶点,仍能保持高精度预测。在 Pin1 蛋白(肿瘤治疗重要靶点)的案例验证中,成功从百万级化合物库中筛选出潜在抑制剂,彰显其实际应用价值。
技术融合亮点
• 整合图卷积网络(GCNs)与化学领域大模型 ChemBERTa
• 蛋白质序列特征提取采用 ESM 语言模型
• 双线性注意力网络(BAN)优化特征交互
• 条件域对抗网络(CDAN)增强跨数据集泛化能力
实证研究表现
在五个权威基准数据集测试中,GraphBAN 全面超越十种主流模型。其特有的领域自适应模块,有效解决了不同实验场景数据分布差异的难题,为 AI 驱动的新药研发提供了可靠的技术支撑。

💻 开源代码:https://github.com/HamidHadipour/GraphBAN
📜 原文链接:https://www.nature.com/articles/s41467-025-57536-9
























 1930
1930

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








