Nat. Mach. Intell.|Delete: 一种结合生成式 AI 和结构方法的先导物优化新模型

导读
今天为大家介绍的是发表在《Nature Machine Intelligence》期刊上,由 Chen Shicheng, Zhang Odin 等人于 2025 年发表的题为“Deep lead optimization enveloped in protein pocket and its application in designing potent and selective ligands targeting LTK protein”的研究工作。
药物发现是一个耗时、昂贵且充满风险的过程。而这项研究提出了一个名为 Delete (Deep lead optimization enveloped in protein pocket) 的深度学习模型,旨在通过结合生成式人工智能和基于结构的方法,为先导化合物优化提供一站式解决方案。与现有的从头设计方案不同,Delete 专注于先导化合物的优化,并直接在蛋白质口袋的三维空间中进行。
当前的计算方法大多局限于解决特定的子任务,例如生成二维分子结构,而忽略了关键的三维蛋白 - 配体相互作用。Delete 模型则通过统一的删除(掩蔽)策略来处理先导化合物优化的所有子任务,并通过等变网络设计考虑了复杂的口袋 - 配体相互作用。
通过统计评估和针对各个子任务的回顾性研究表明,Delete 拥有出色的能力,可以使用给定的片段或原子来设计具有更优蛋白质结合能和合理成药性的分子。
研究人员利用 Delete 设计了靶向先前已确定的 LTK 蛋白的抑制剂。在 Delete 设计的配体中,CA-B-1 通过体外和体内实验成功验证为一种强效(1.36nM)和选择性抑制剂。这项工作代表了强大的基于结构的先导化合物优化模型 Delete 在快速和可控的合理药物设计中的成功应用。
文章还指出,虽然人工智能辅助药物设计 (AIDD) 已在抗菌药物研发中展现出有效性,但目前的分子生成工作主要集中于基于配体的从头设计模型,这限制了其在研究较少的靶点和整合化学家先验知识方面的应用。Delete 模型的提出有望克服这些限制,为药物发现提供更有效和更灵活的工具。
引言
Delete 模型将基于结构的范式和先导物优化方法相结合,为先导物优化提供了全面的解决方案。不同于依赖数据记忆识别化学模式的基于配体的范式,该模型侧重于理解潜在的物理相互作用,使其能够有效地研究未见过的靶点。研究者利用几何感知的等变网络作为骨架,使模型输出的几何形状与输入旋转动态对齐,同时保持原子和键拓扑结构不变,从而消除了数据增强的需求,并能同时生成蛋白质 - 配体结合结构。
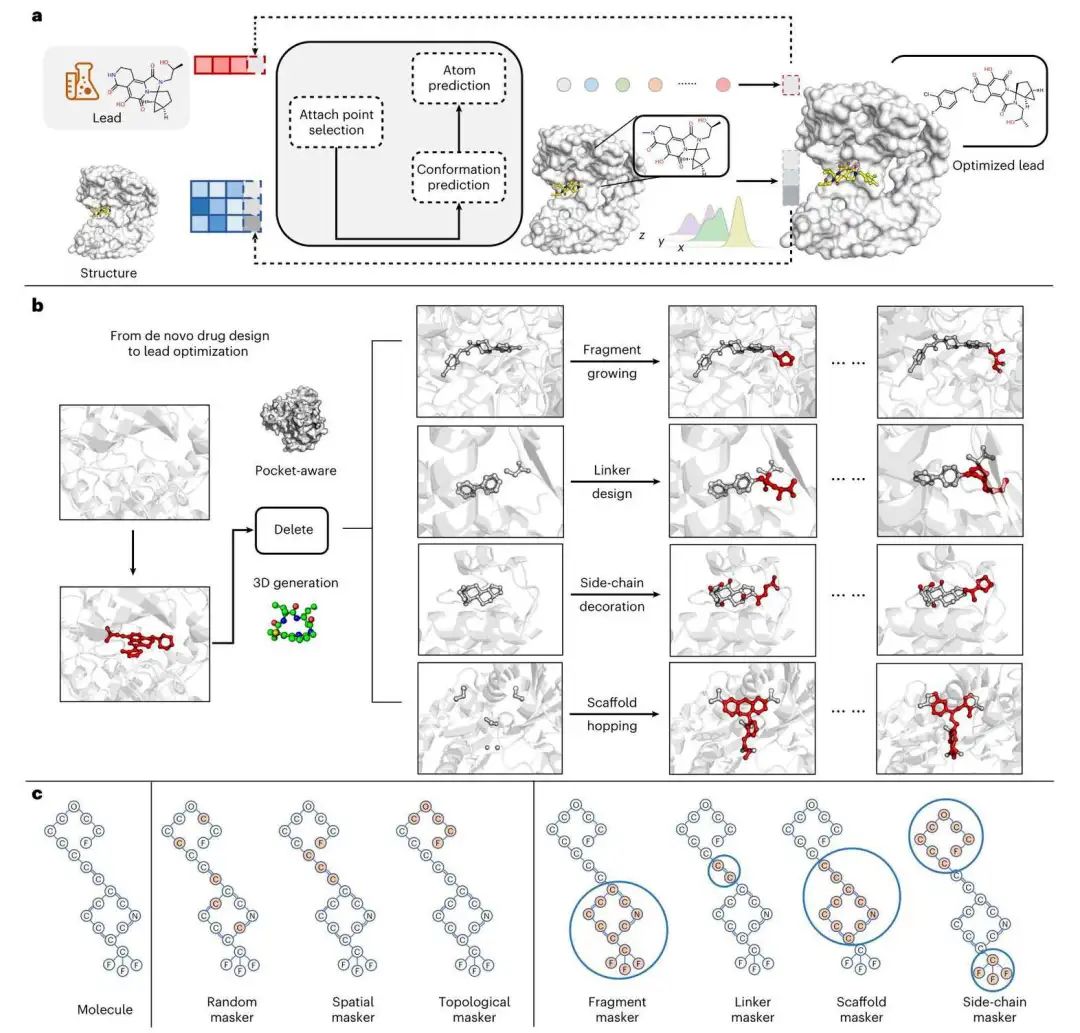
图 1:Delete 是一个用于先导物优化的一体化解决方案。a, Delete 的工作流程。b, Delete 是一个口袋感知和 3D 生成模型,它将从头设计扩展到四个先导物优化任务。c, 统一的删除(掩码)策略图示。前三个是增强策略,后四个是特定于任务的策略。
受掩码图建模和图数据结构预训练策略的启发,作者引入了一种统一的删除策略(如图 1 所示),包括三个增强的掩码器(随机、空间和拓扑)和四个特定于任务的掩码器(连接体、片段、骨架和侧链)。这使得 Delete 能够处理连接体设计、片段扩展、骨架跃迁和侧链修饰等四种不同的先导物优化子任务。
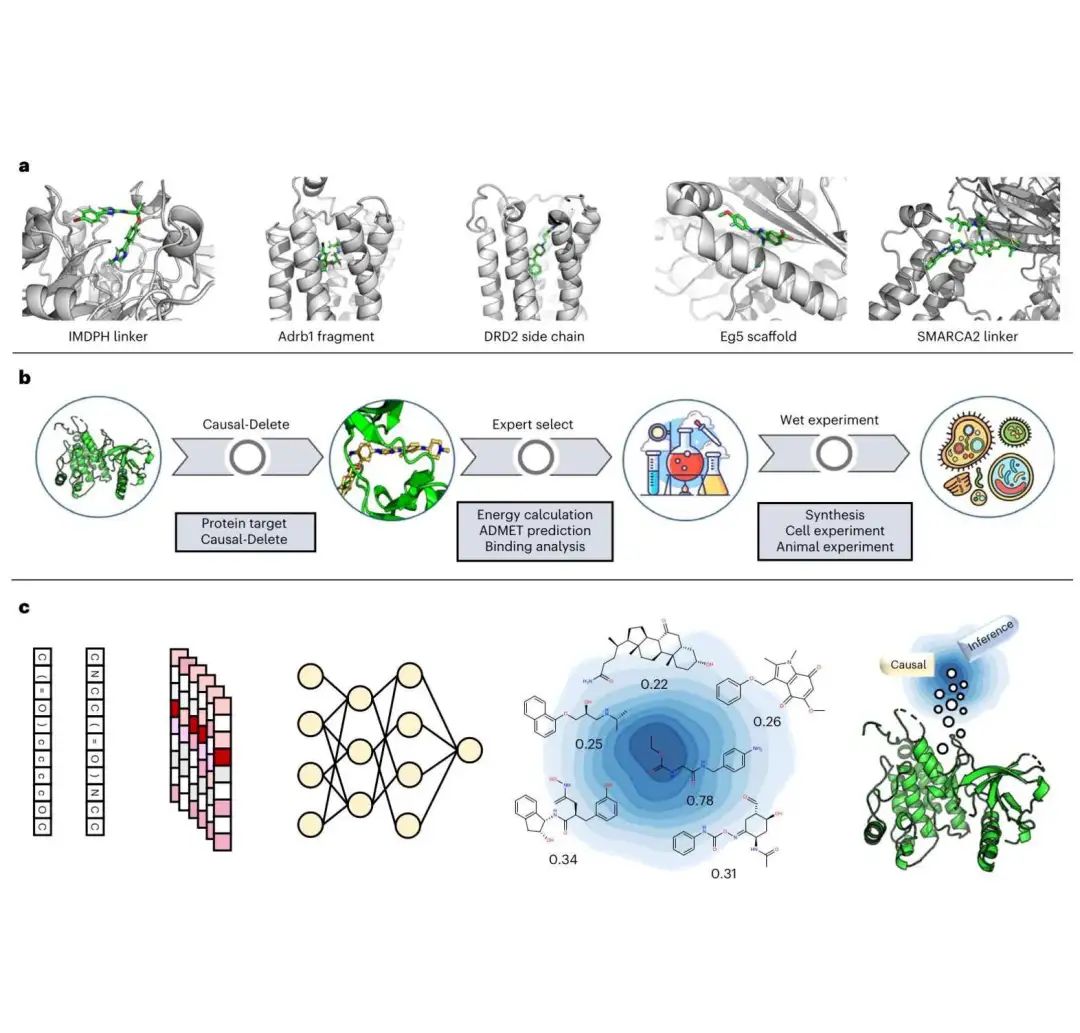
图 2:回顾性研究和迁移学习技术。a,五个回顾性研究的图示。b,将基于配体的方法整合到 Causal-Delete 中,使用 Delete 设计针对先前确定的 LTK 靶点的抑制剂。c,将 ALK 抑制剂的化学空间整合到 Delete 中。在实际应用中使用的迁移学习技术,旨在利用 ALK 的化学空间来帮助 LTK 配体设计。
通过模型比较、回顾性分析(如图 2 所示)和针对白细胞酪氨酸激酶 (LTK) 的抑制剂设计,研究者验证了 Delete 模型的有效性。表 1 中的数据显示,Delete 在四个子任务上均优于其他基线模型。此外,Delete 还成功地复制了五个历史药物开发案例的结果。通过迁移学习技术,Delete 被扩展为基于结构和基于配体的混合模型。最终,Delete 成功设计了一系列针对 LTK 激酶结构域的活性配体,其中最有希望的候选药物 CA-B-1 显示出优异的纳摩尔活性(IC50 = 1.36 nM)。
Delete 模型方法
研究者利用蛋白质口袋(P)和现有结构(Lμ)预测完整配体(L)的分布 P(L|Lμ, P)。为捕捉口袋和配体之间复杂的相互作用,研究者采用了基于欧式距离的 k 近邻方法构建几何图结构,并提取详细的相互作用作为边特征。模型的等变设计确保了必要的物理约束。此外,蛋白质口袋由三角形面片表示,以提取其物理特征。交互模块学习整个系统(包括每个节点及其相邻几何边)的“状态”,生成模块则根据学习到的交互特征建议下一个原子的位置。更多细节可参考补充信息方法。
模型训练采用 PyTorch 和 PyTorch Geometric 框架,使用 Adam 优化器,初始学习率为 2×10⁻⁴,并使用学习率调度器和梯度裁剪。模型在 NVIDIA 3090 GPU 上训练 300 个 epoch,预训练和微调的批量大小分别为 256 和 4。
模型评估主要指标为生成的分子与目标蛋白之间的结合紧密度,通过原位结合能(Scoring)和对接结合能(Docking)来体现。此外,还评估了 QED、合成难度、Lipinski 五规则和 LogP 等分子属性指标。
为将配体信息整合到基于结构的药物设计方法中,研究者采用因果迁移学习技术。通过训练 QSAR 模型来表征 ALK 活性分子的化学空间。该 QSAR 模型使用 ECFP4 指纹表示分子,并采用集成学习方法(包括随机森林、XGBoost、CatBoost、LightGBM、KNN 和神经网络)进行训练,并使用 n 重复 k-bagging 进行交叉验证。此外,利用 ADMET Lab 预测候选药物的吸收和排泄特性(生物利用度和半衰期)。
Delete 模型在重新标记的结合亲和力、生物利用度和半衰期上进行微调,以将活性区域和良好的 ADMET 特性区域纳入搜索空间。微调损失函数如下:
其中,m 是活性分子的数量,li、βi 和 γi 分别表示分子 i 的重新标记的结合亲和力、生物利用度和半衰期。
数据集可通过 Zenodo (https://doi.org/10.5281/zenodo.14586176) 获取,源代码可在 GitHub (https://github.com/HaotianZhangAl4Science/Delete) 获取。
主要结果
Delete 模型充分考虑了蛋白质口袋内的化学环境和几何特征,并通过拓扑表面模型和几何图模型分别对蛋白质和分子进行建模。相互作用表征框架学习蛋白质口袋和先导化合物片段的拓扑和几何相互作用,生成包含几何和能量信息的节点表示。分子生成采用自回归形式,使其在利用口袋信息作为强约束指导分子生成过程之前,能够与拓扑口袋完全相互作用。
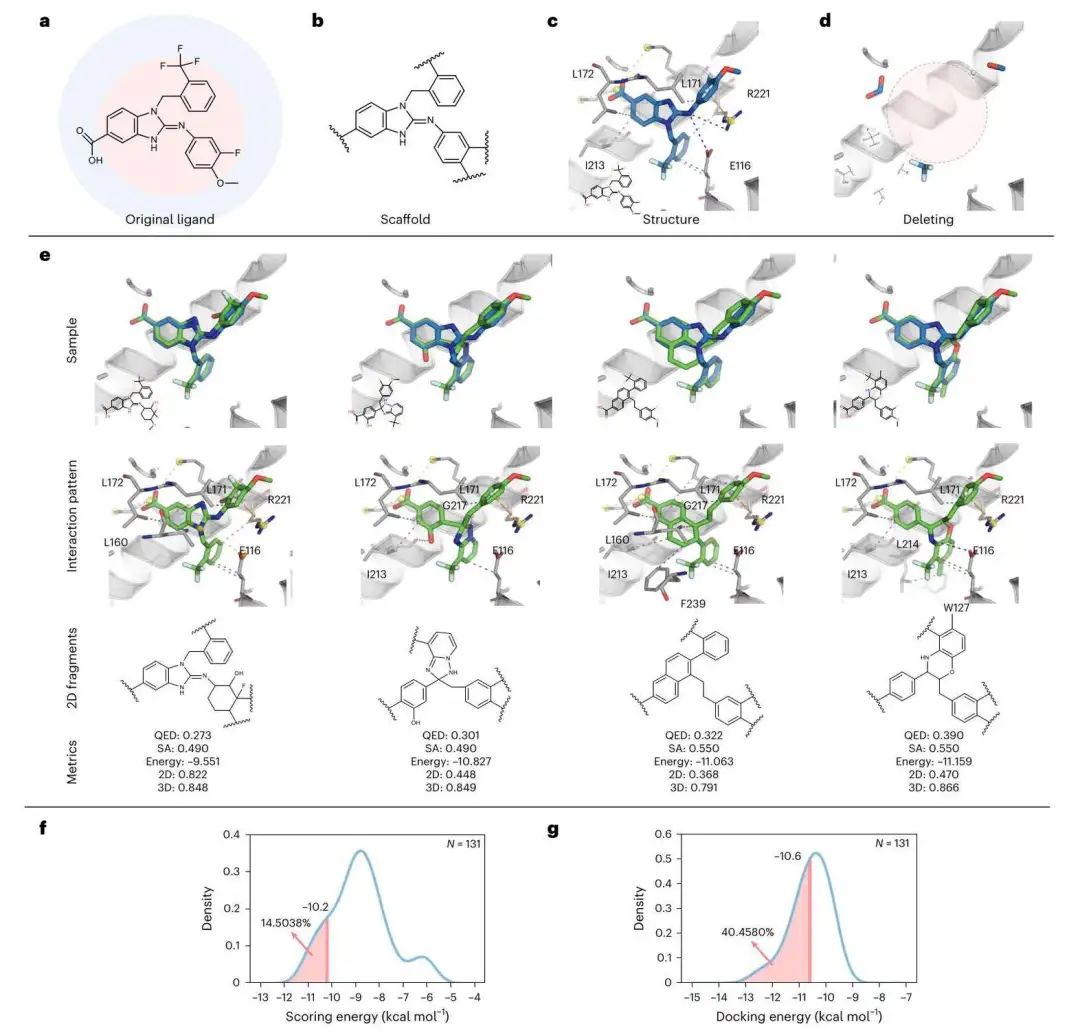
图 3 | Eg5 的骨架跃迁案例研究。a, b, BI8 的二维化学结构 (a) 及其骨架 (b)。红色代表 Bemis-Murcko 骨架,蓝色部分代表 TFs。c, BI8 的晶体结构 (PDB: 3ZCW)。d, Delete 生成的起点,其中红色循环是潜在的生长空间。e, 对四个生成分子的分析。f, g, 评分能量 (f) 和对接能量 (g)。蓝色曲线是生成化合物的预测亲和力分布,橙色线表示原始化合物。SA,合成可及性。
研究者将 Delete 模型与七个其他特定任务模型在四个子任务中进行了比较分析,结果表明,Delete 模型在原位结合能(Scoring)和对接后结合能(Docking)等关键指标上均优于基线模型。此外,Delete 还在分子性质指标上取得了令人满意的结果。
为了进一步验证 Delete 模型的有效性,研究者进行了五项回顾性研究,其中包括针对 Eg5 的骨架跃迁案例。结果显示,Delete 成功生成了与原始化合物空间特征相似的新型化合物,且结合能相当甚至更优。
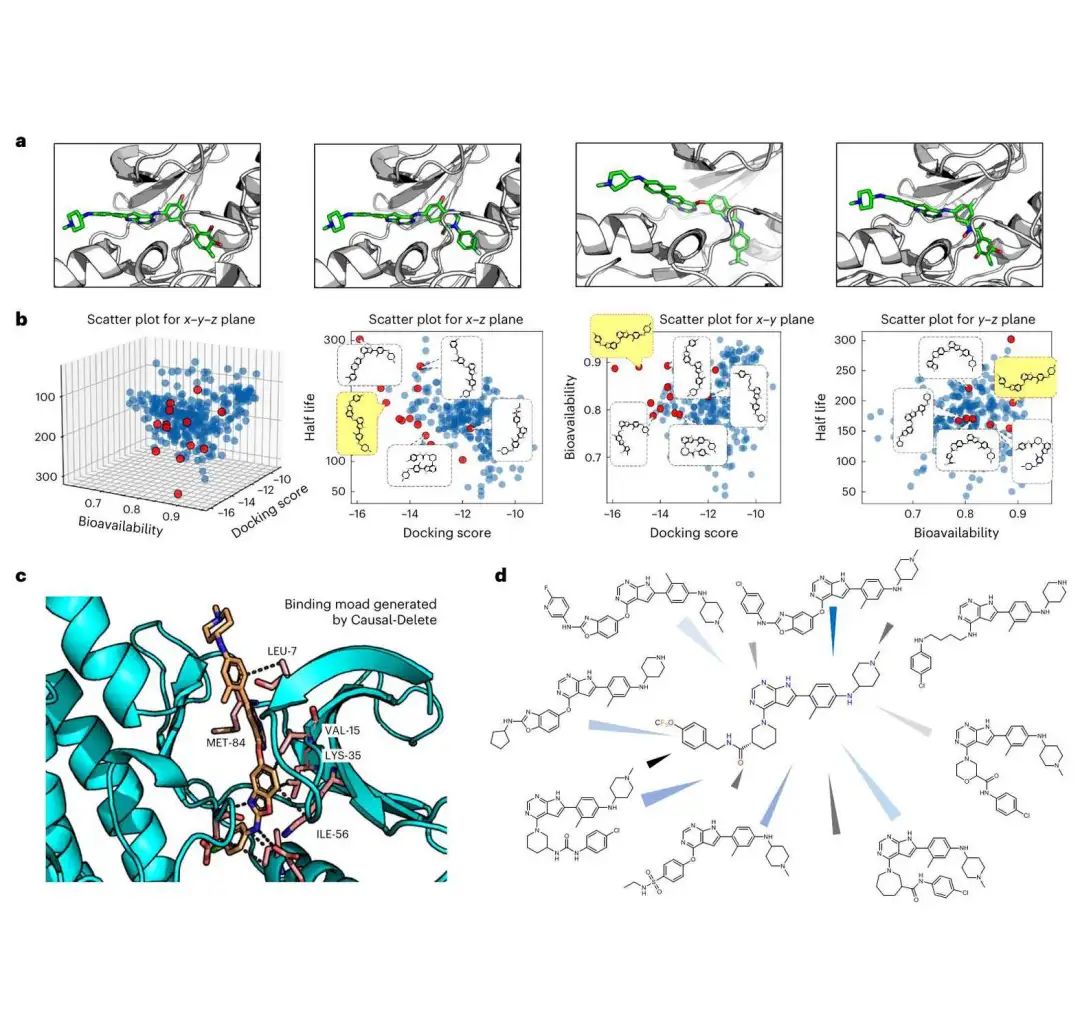
图 4 | Causal-Delete 在 LTK 靶点上的应用。a,生成样本的可视化。构象由 Causal-Delete 直接生成。b,生成分子的化学空间。c,CA-B-1 与 LTK 结合的相互作用模式。d,Causal-Delete 设计的合成抑制剂。
此外,研究者将 Delete 应用于 LTK 靶点药物设计,并成功设计了一种新型 LTK 抑制剂 CA-B-1。CA-B-1 在体外和体内实验中均表现出优异的抗肿瘤活性、高选择性和良好的药代动力学特性。
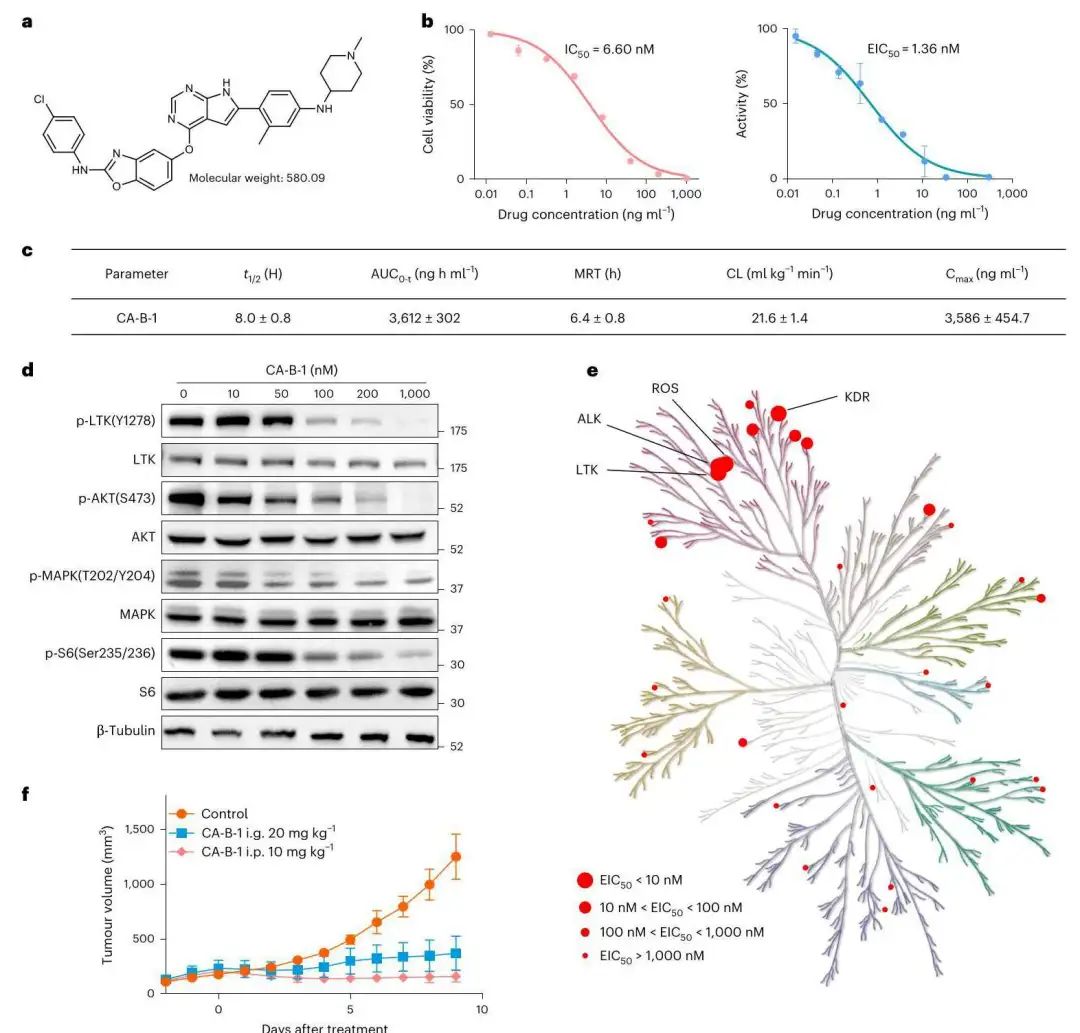
图 5 | CA-B-1 在体外和体内均表现出抗肿瘤功效。a,CA-B-1 的结构。b,CA-B-1 对 MTT 法测定的 Ba/F3-CLIP1-LTK 细胞活力的影响(n = 3),以及 CA-B-1 通过迁移率变化法对 LTK 的抑制效力(n = 2)。数据以平均值±标准差表示。c,在 Sprague Dawley 大鼠中静脉注射(5 mg kg-1)后 CA-B-1 的血浆浓度 - 时间曲线和体内药代动力学参数(n = 3)。d,CA-B-1 对 CLIP1-LTK 激酶活性的抑制以及对下游信号通路 PI3K/AKT 和 RAS/MAPK 的激活。β-微管蛋白用作上样对照。e,CA-B-1 对 31 种激酶的选择性。EIC50 代表化合物对蛋白质活性半数最大抑制浓度。f,CA-B-1 治疗组或载体对照组的肿瘤体积(每组 n = 6 只小鼠)。所有图形条形图均代表平均值±标准差。i.g.,灌胃给药;i.p.,腹腔注射。
尽管 Delete 模型取得了显著成果,但仍存在一些局限性,例如无法考虑构象灵活性以及对大规模数据集的处理能力。未来的研究将集中于改进训练方法、整合更有效的数据和采样技术,以进一步提高 Delete 模型的泛化能力。
总结
药物发现是一个耗时、昂贵且高风险的过程。现代人工智能辅助药物设计 (AIDD) 旨在通过自动发现候选药物来加速这一过程。虽然 AIDD 在 QSAR 模型的应用中取得了一定进展,例如 Collins 团队在抗菌药物研发中的工作,但其受限于已充分研究的药物靶点和充足的分子数据。另一类 AIDD 方法,如 JAEJER、GENTRL 和 GDL-RIPK1 等分子生成模型,通过超越离散化学库,直接在连续化学空间中探索分子。然而,目前经过验证的分子生成工作主要集中在基于配体的从头设计模型。
基于配体的方法同样受限于已充分研究的靶点,需要对已证实对目标蛋白具有活性的分子进行微调。此外,化学家通常会利用先验知识,旨在修饰具有低亲和力或其他不太理想特性的先导化合物或骨架8。然而,从头设计方法通常无法将化学家的先验知识整合到生成过程中,从而降低了他们对这些“黑盒”模型的依赖。
为了克服这些挑战,作者提出了 Delete (Deep lead optimization enveloped in protein pocket) 模型,它结合了生成式人工智能和基于结构的方法,为先导化合物优化提供了一站式解决方案。Delete 模型通过统一的删除(掩蔽)策略处理先导化合物优化的所有子任务,并通过等变网络设计考虑了复杂的口袋 - 配体相互作用。
跨各个子任务的统计评估和回顾性研究表明,Delete 模型能够利用给定的片段或原子生成具有优异蛋白结合能和合理成药性的分子。随后,研究者利用 Delete 设计了靶向先前鉴定的 LTK 蛋白的抑制剂。在 Delete 设计的配体中,CA-B-1 通过体外和体内实验成功验证为一种强效 (1.36nM) 和选择性抑制剂。
要点回顾
Q1:这篇文章提出了什么新的方法,以及该方法的主要优势是什么?
A:这篇文章提出了名为 Delete (Deep lead optimization enveloped in protein pocket) 的深度学习模型,用于药物发现中的先导化合物优化。其主要优势在于:
- 一体化解决方案: Delete 将生成式人工智能与基于结构的方法相结合,可以处理先导化合物优化的所有子任务(例如连接体设计、片段扩展、骨架跃迁和侧链修饰),而无需像现有方法那样依赖多个特定于任务的模型。
- 基于三维结构: Delete 考虑了蛋白质 - 配体在三维空间中的相互作用,通过等变网络设计捕捉复杂的口袋 - 配体相互作用,克服了先前模型主要关注二维分子结构生成的局限性。
- 高效生成高质量分子: Delete 能够生成具有更优蛋白质结合能和合理药物相似性的分子,并在实际应用中成功设计出高效且具有选择性的 LTK 蛋白抑制剂 CA-B-1。
Q2:Delete 模型是如何工作的?
A:Delete 模型的工作流程如下:
- 输入: 蛋白质口袋结构和先导化合物片段。
- 蛋白质和分子建模: 蛋白质使用拓扑表面模型 𝒢p 建模,分子使用几何图模型 𝒢l 建模。
- 相互作用表征: 交互模块学习蛋白质口袋和先导化合物片段之间的拓扑和几何相互作用,生成包含几何和能量信息的节点表示。
- 分子生成: 生成模块以自回归方式建议下一个原子的位置,利用学习到的相互作用特征,并在蛋白质口袋的拓扑信息作为强约束的指导下进行分子生成。
Q3:文章中提到了哪些评估指标,以及 Delete 模型在这些指标上的表现如何?
A:文章中使用的评估指标包括:
- 结合能: 包括原位结合能 (Scoring) 和对接结合能 (Docking),用于评估生成的分子与靶蛋白之间的结合紧密程度。Delete 在这些指标上优于其他基线模型。
- 分子属性指标: 包括定量药物相似性 (QED)、合成可及性、Lipinski 五规则和 LogP,用于评估生成的化合物的成药性。Delete 在这些指标上的表现与测试集中的分子相似,但在某些指标上不及一些二维生成模型。
- 命中率 (Hit): 指生成具有更好结合能的分子比例。Delete 在这方面也表现出色。
Q4:文章中提到的 CA-B-1 是什么,以及它的重要性是什么?
A:CA-B-1 是由 Delete 模型设计的靶向 LTK 蛋白的抑制剂。它的重要性在于:
- 高效和高选择性: CA-B-1 在体外和体内实验中均被证实为一种高效 (1.36 nM) 和高选择性的 LTK 抑制剂。
- 验证 Delete 的实用性: CA-B-1 的成功设计和验证,证明了 Delete 模型在快速和可控的合理药物设计中的实用性和潜力,也突出了生成式 AI 结合基于结构方法的有效性。
Q5:Delete 模型有哪些局限性以及未来的研究方向?
A:Delete 模型的局限性包括:
- 构象灵活性: Delete 未能考虑蛋白质和配体的构象灵活性,生成的配体仅基于结合口袋的刚性几何和能量特性。
- 可扩展性: 将 Delete 扩展到更大或更多样化的数据集可能存在挑战,因为增加的计算需求可能会影响性能。
- 对新蛋白质结构的适用性: 模型对新或特征描述不足的蛋白质结构的有效性仍有待确定。
未来的研究方向包括:
- 使用动态图神经网络来更好地捕捉配体与其靶口袋之间的诱导契合效应。
- 改进训练方法和整合更有效的数据和采样技术,以增强 Delete 对不同蛋白质靶标的泛化能力。
























 1006
1006

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








