1.模型的保存与加载API
保存:
from sklearn.datasets import load_boston
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.externals import joblib
def linear():
lb = load_boston()
x_train, x_test, y_train, y_test = train_test_split(lb.data, lb.target, test_size=0.25)
# 由于x y的特征个数不同,要分别标准化
std_x = StandardScaler()
x_train = std_x.fit_transform(x_train)
x_test = std_x.transform(x_test)
std_y = StandardScaler()
y_train = std_y.fit_transform(y_train.reshape(-1, 1))
y_test = std_y.transform(y_test.reshape(-1, 1))
# 正规方程求解
lr = LinearRegression()
lr.fit(x_train,y_train)
#保存模型
joblib.dump(lr,'./test.pkl')
return None
if __name__ == '__main__':
linear()
加载:
from sklearn.datasets import load_boston
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.externals import joblib
def linear():
lb = load_boston()
x_train, x_test, y_train, y_test = train_test_split(lb.data, lb.target, test_size=0.25)
# 由于x y的特征个数不同,要分别标准化
std_x = StandardScaler()
x_train = std_x.fit_transform(x_train)
x_test = std_x.transform(x_test)
std_y = StandardScaler()
y_train = std_y.fit_transform(y_train.reshape(-1, 1))
y_test = std_y.transform(y_test.reshape(-1, 1))
#模型导入
model = joblib.load("./test.pkl")
# 将标准化后的数据转换为原始数据
y_predict = std_y.inverse_transform(model.predict(x_test))
print("预测结果:",y_predict)
print("正规方程的均方误差:",mean_squared_error(std_y.inverse_transform(y_test),y_predict))
return None
if __name__ == '__main__':
linear()
说明:
- Joblib是一组用于在Python中提供轻量级流水线的工具
- Joblib可以将模型保存到磁盘并可在必要时重新运行
2.逻辑回归概述
原理:通过sigmoid函数
σ
(
x
)
=
1
1
+
e
−
x
\sigma (x)=\frac{1}{1+e^{-x}}
σ(x)=1+e−x1将输入过的数据映射到0~1之间的数字,判断数字是否超过阈值为其打上标签
一般假设函数为:
h
(
x
i
)
=
1
1
+
e
−
(
w
T
x
i
+
b
)
h(\textup{x}^{i})=\frac{1}{1+e^{-(\textup{w}^{T}\textup{x}^{i}+b)}}
h(xi)=1+e−(wTxi+b)1
x
i
x^{i}
xi是测试集第i个数据,
w
w
w是p维列向量,和b都为待求参数;
逻辑回归的损失函数-对数似然损失:
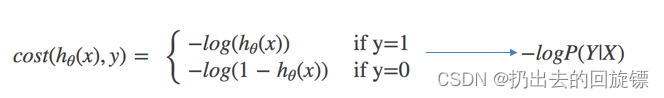
说明:
- 损失函数是体现“预测值”和“真实值”,相似程度的函数
- 损失函数越小,模型越好
- 可能有多个局部最小值
完整的损失函数:
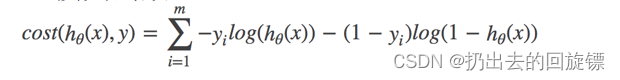
针对多局部最小值的优化方式(不能完全解决问题)
- 多次随机初始化,比较最小结果
- 调整学习率
3.逻辑回归 API
良/恶性乳腺癌种数据分类:
数据描述:

from sklearn.metrics import classification_report
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegression
import pandas as pd
import numpy as np
def logistic():
# 构造列标签
column = ['Sample code number','Clump Thickness', 'Uniformity of Cell Size','Uniformity of Cell Shape','Marginal Adhesion','Single Epithelial Cell Size','Bare Nuclei','Bland Chromatin','Normal Nucleoli','Mitoses','Class']
data = pd.read_csv("./breast-cancer-wisconsin.data",names=column)
# 缺失值处理
data = data.replace(to_replace='?',value=np.nan)
data = data.dropna()
# 数据分割
x_train, x_test, y_train, y_test = train_test_split(data[column[1:10]],data[column[10]],test_size=0.25)
# 标准化
std = StandardScaler()
x_train = std.fit_transform(x_train)
x_test = std.transform(x_test)
# 逻辑回归预测
lg = LogisticRegression(C=1.0)
lg.fit(x_train, y_train)
print(lg.coef_)
y_predict = lg.predict(x_test)
print("准确率:",lg.score(x_test,y_test))
print("召回率:",classification_report(y_test,y_predict,labels=[2,4],target_names=["良性","恶性"]))
return None
if __name__== "__main__":
logistic()
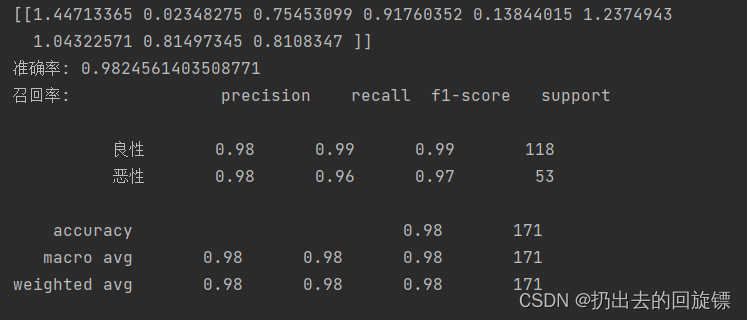
说明:
- 不适合解决多分类问题
- 简单,速度快
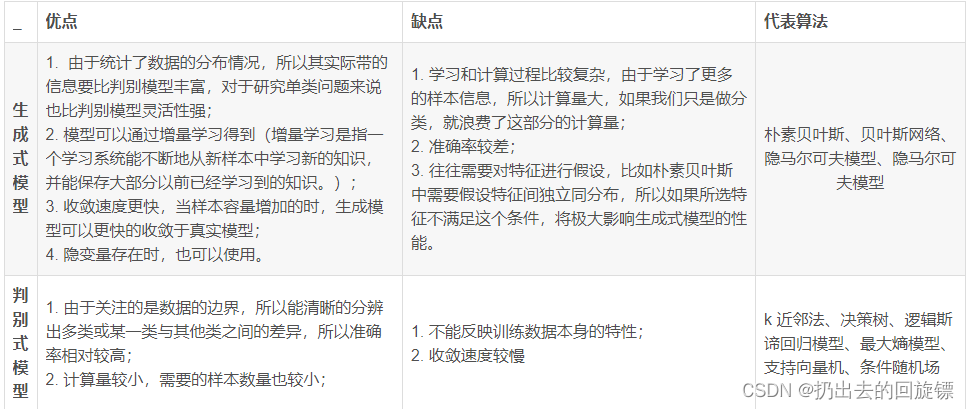
- 生成模型与判别模型对比
- 生成模型一般以概率方式描述数据的产生方式,通过对模型采样就可以产生数据。
- 判别模型对数据之间的映射关系建模,而不考虑数据本身是如何生成的。模型自身并不能产生数据。
- 生成模型对数据本身建模,更基础。判别模型只考虑输入和输出之间的关系,更直接地面向问题。如果希望用生成模型完成判定模型的任务,一般需要额外的步骤。
4.K-means 算法步骤
1、随机设置K个特征空间内的点作为初始的聚类中心
2、对于其他每个点计算到K个中心的距离,未知的点选择最近的一个聚类中心点作为标记类别
3、接着对着标记的聚类中心之后,重新计算出每个聚类的新中心点(平均值)
4、如果计算得出的新中心点与原中心点一样,那么结束,否则重新进行第二步
5.KMeans API
步骤一:库和数据的导入
import pandas as pd
from sklearn.decomposition import PCA
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from sklearn.metrics import silhouette_score
prior = pd.read_csv("./data/order_products__prior.csv")
products = pd.read_csv("./data/products.csv")
order = pd.read_csv("./data/orders.csv")
aisles = pd.read_csv("./data/aisles.csv")
步骤二:合并表项,方便操作
_mg = pd.merge(prior,products,on=['product_id','product_id'])
_mg = pd.merge(_mg,order,on=['order_id','order_id'])
mt = pd.merge(_mg,aisles,on=['aisle_id','aisle_id'])
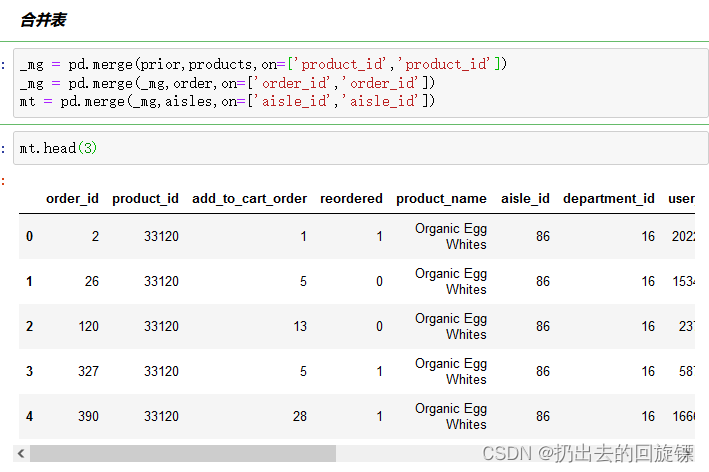
步骤三:交叉表分组
cross = pd.crosstab(mt['user_id'],mt['aisle'])
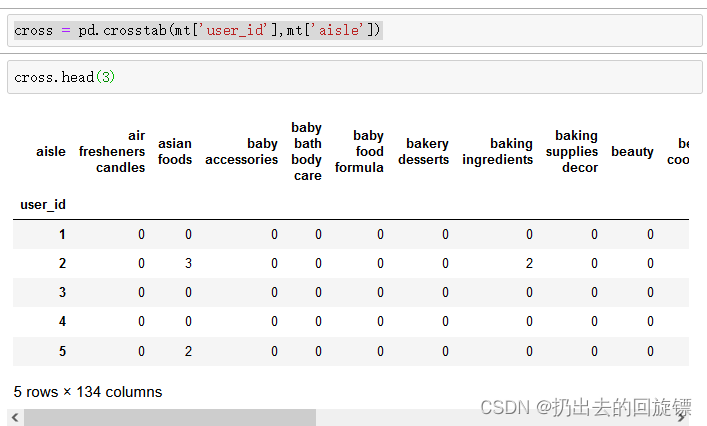
步骤四:主成分分析,并且减少样本数量
pca = PCA(n_components=0.9)
data = pca.fit_transform(cross)
x = data[:900]
x.shape

步骤五:调用kmeansAPI进行聚类
km = KMeans(n_clusters=4)
predict = km.predict(x)
predict
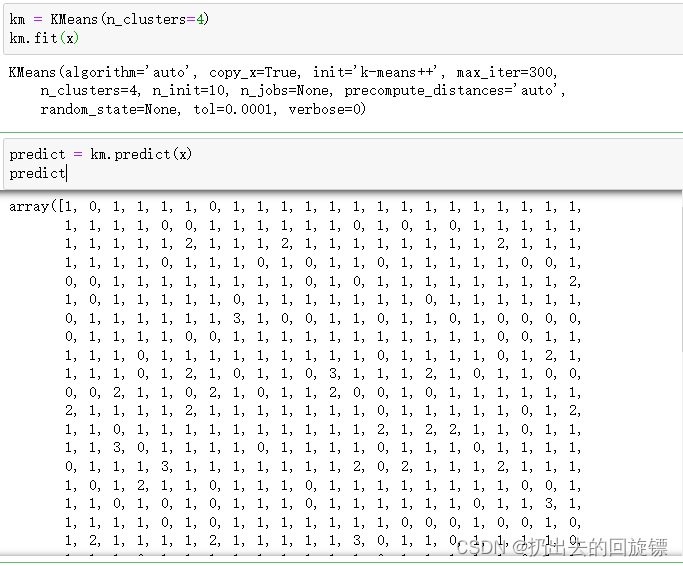
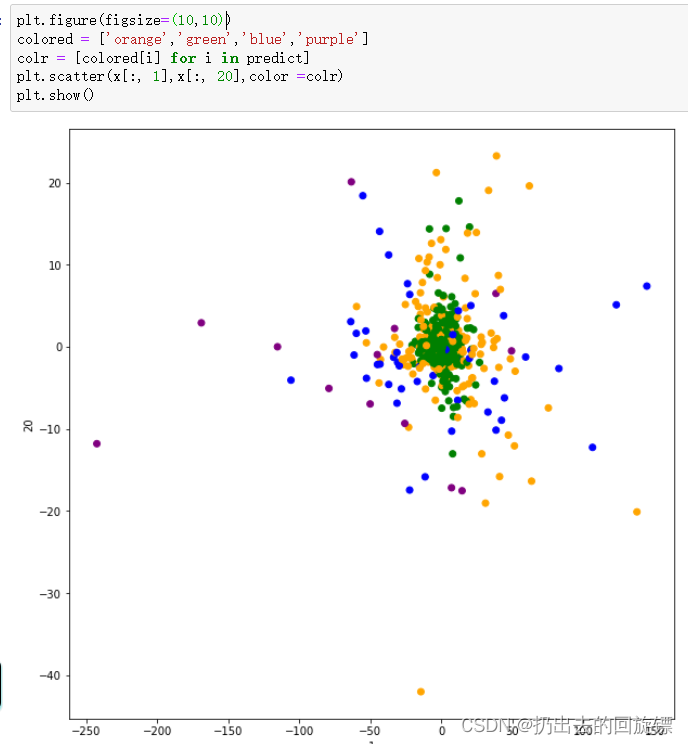
步骤六:图形绘制
plt.figure(figsize=(10,10))
colored = ['orange','green','blue','purple']
colr = [colored[i] for i in predict]
plt.scatter(x[:, 1],x[:, 20],color =colr)
plt.show()
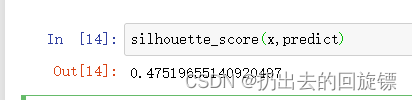
步骤七:效果评估
silhouette_score(x,predict)
说明:
1.聚类评估标准:轮廓系数
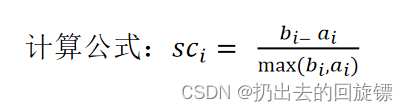
- 对于每个点i 为已聚类数据中的样本 ,bi 为i 到其它族群的所有样本的平均距离,ai 为i 到本身簇的距离平均值
- 轮廓系数的值是介于 [-1,1] ,越趋近于1代表内聚度和分离度都相对较优
2.Kmeans API参数

3.silhouette_score 参数

4.Kmeans的特点
- 容易收敛到局部最优解(多次聚类)
- 采用迭代式算法,直观易懂并且非常实用
- 需要预先设定簇的数量(k-means++解决)






















 308
308











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








