
Miniconda — Anaconda documentation
Visual Studio Code - Code Editing. Redefined
在VSCode上运行Jupyter Notebook_vscode jupyter-CSDN博客
如果需要多个环境:
【anaconda】conda创建、查看、删除虚拟环境(anaconda命令集)_conda 创建环境-CSDN博客
打开powershell

在终端输入:
pip install abagen -i https://pypi.tuna.tsinghua.edu.cn/simple # -i 表示换源;pip相当于就是软件下载工具以下链接为abagen的使用教程以及对应的文章
User guide — abagen 0.1.3-doc+0.g2aeab5b.dirty documentation
Standardizing workflows in imaging transcriptomics with the abagen toolbox | eLife
VScode使用教程:
1.添加中文包
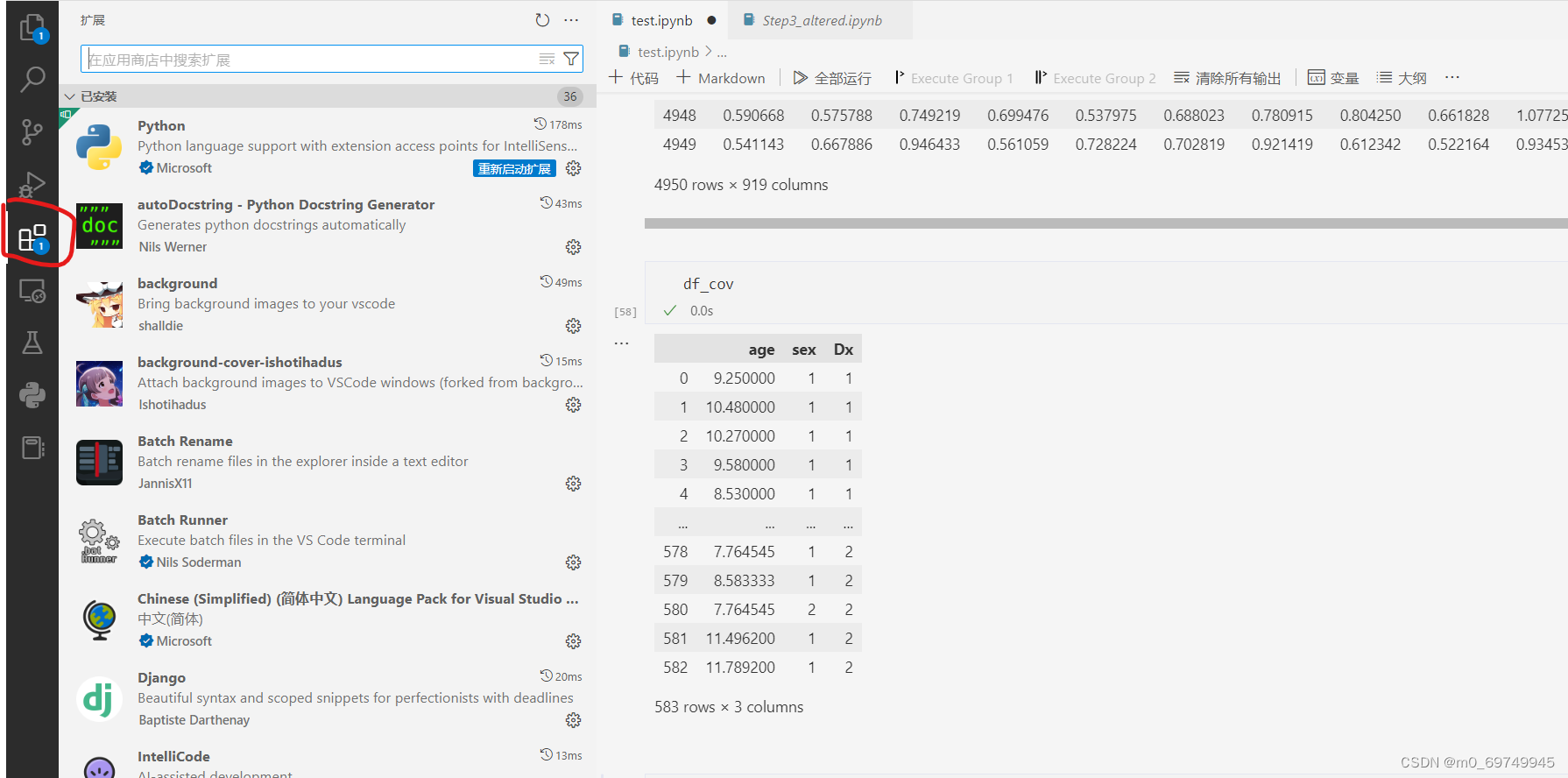
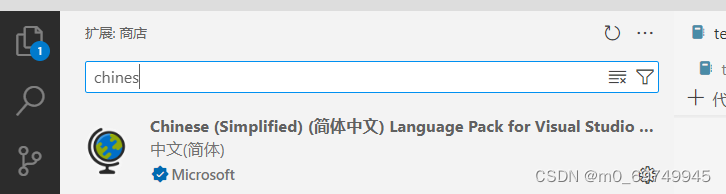
2.新建工作目录
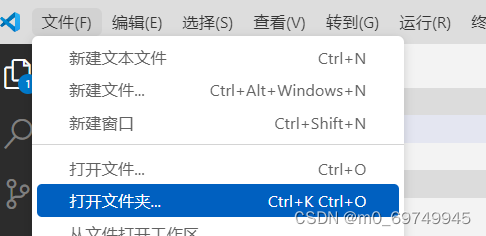
3.新建文件,以.ipynb结尾
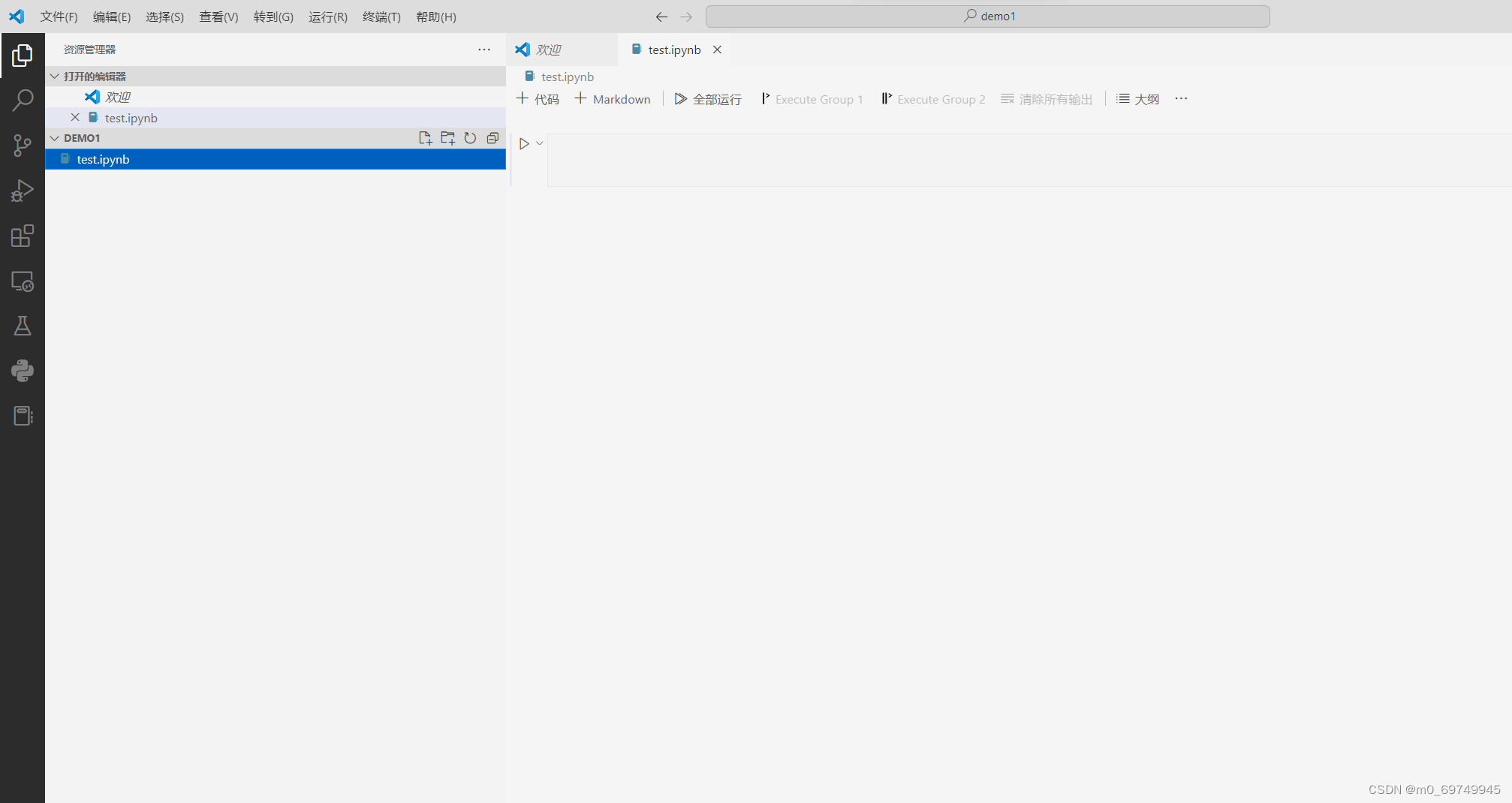
4.选择jupyter内核






















 4708
4708

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








