一、主成分分析(PCA)简介
主成分分析(Principal Component Analysis, PCA)是一种常用的数据降维技术,用于简化数据集,同时保留尽可能多的信息。它通过将原始高维数据转换为较低维度的主成分,从而降低数据的复杂性。这种方法不仅有助于减少计算量,还能帮助我们更容易地可视化和理解数据。
什么是PCA?
PCA是一种线性变换技术,其核心思想是找到数据集中变化最大的方向,即主成分。这些主成分是原始特征的线性组合,按解释方差的大小排序。第一个主成分解释了数据中最多的方差,第二个主成分解释了剩余方差中的最大部分,依此类推。
为什么使用PCA?
- 降维:在处理高维数据时,PCA能够显著减少维度,简化数据结构,降低计算成本。
- 去除噪声:通过保留主要特征,PCA可以去除数据中的噪声,提高数据质量。
- 可视化:PCA将高维数据转换为2D或3D数据,便于可视化分析,帮助我们更直观地理解数据。
二、算法流程
2.1 主要步骤
1. 数据标准化:
- 将数据中心化,即减去每个特征的均值,使得每个特征的均值为0。
- 对数据进行标准化处理(可选),即将每个特征除以其标准差,使得数据的尺度一致。
假设数据集为 X,其中 X 是一个 m×n的矩阵,m是样本数,n 是特征数。
标准化后的数据![]() 为:
为:![]()
其中![]() 是第 j个特征的均值,
是第 j个特征的均值, ![]() 是第 j个特征的标准差。
是第 j个特征的标准差。
2. 计算协方差矩阵:
- 计算中心化数据的协方差矩阵。协方差矩阵反映了各特征之间的相关性。
中心化后的数据 ![]() 为:
为:![]()
协方差矩阵 Σ 为:
3. 计算特征值和特征向量:
- 对协方差矩阵进行特征值分解,得到特征值和对应的特征向量。特征值反映了对应特征向量方向上的数据方差大小。
对协方差矩阵 Σ 进行特征值分解:
![]()
其中,λ是特征值,v 是对应的特征向量。
4. 选择主成分:
- 根据特征值的大小排序,选择前k个最大的特征值对应的特征向量。这些特征向量就是新的低维空间的基底,称为主成分。
选择前 k个最大的特征值对应的特征向量,组成一个矩阵 Vk ,其中每列是一个特征向量。
5. 转换数据:
- 将原始数据投影到选取的主成分上,得到降维后的数据。
将原始数据投影到新的低维空间:
![]()
这样,Xnew 就是降维后的数据。
2.2 步骤计算示例
假设我们有一个2维的数据集:

- 数据标准化:

2. 计算协方差矩阵:

3. 计算特征值和特征向量:

4. 选择主成分:
选择最大的特征值对应的特征向量 v1。
- 转换数据:
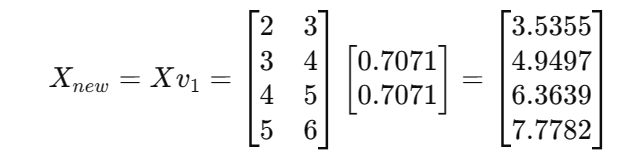
这样,原始数据集就被降维到一维。
三、代码展示
1. 首先,我们需要导入所需的库并加载数据集。这里使用的是著名的Iris数据集。
import numpy as np
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
import time
# 加载示例数据集,这里使用iris数据集
data = load_iris()
X = data.data
y = data.target2. 计算PCA并记录结果
我们将计算不同主成分数量下的累计解释方差比例、计算时间和重构误差。
# 存储不同主成分数量下的累计解释方差比例和计算时间
explained_variances = []
computation_times = []
reconstruction_errors = []
# 计算PCA并记录每次主成分数量下的累计解释方差比例、计算时间和重构误差
for n_components in range(1, X.shape[1] + 1):
start_time = time.time()
pca = PCA(n_components=n_components)
X_pca = pca.fit_transform(X)
explained_variances.append(np.sum(pca.explained_variance_ratio_))
computation_times.append(time.time() - start_time)
# 计算重构误差
X_reconstructed = pca.inverse_transform(X_pca)
reconstruction_error = np.mean((X - X_reconstructed) ** 2)
reconstruction_errors.append(reconstruction_error)
3. 绘制结果图表
接下来,我们绘制累计解释方差比例、计算时间和重构误差的折线图。
# 绘制累计解释方差比例折线图
plt.figure(figsize=(14, 6))
plt.subplot(1, 3, 1)
plt.plot(range(1, X.shape[1] + 1), explained_variances, marker='o')
plt.title('Explained Variance vs. Number of Principal Components')
plt.xlabel('Number of Principal Components')
plt.ylabel('Cumulative Explained Variance Ratio')
plt.xticks(range(1, X.shape[1] + 1))
plt.grid(True)
# 绘制计算时间折线图
plt.subplot(1, 3, 2)
plt.plot(range(1, X.shape[1] + 1), computation_times, marker='o')
plt.title('Computation Time vs. Number of Principal Components')
plt.xlabel('Number of Principal Components')
plt.ylabel('Computation Time (seconds)')
plt.xticks(range(1, X.shape[1] + 1))
plt.grid(True)
# 绘制重构误差折线图
plt.subplot(1, 3, 3)
plt.plot(range(1, X.shape[1] + 1), reconstruction_errors, marker='o')
plt.title('Reconstruction Error vs. Number of Principal Components')
plt.xlabel('Number of Principal Components')
plt.ylabel('Reconstruction Error')
plt.xticks(range(1, X.shape[1] + 1))
plt.grid(True)
plt.tight_layout()
plt.show()
4. 二维PCA散点图
我们将数据降到二维,并绘制散点图来观察不同类别的分布。
# 使用前两主成分进行降维并绘制散点图
pca_2d = PCA(n_components=2)
X_pca_2d = pca_2d.fit_transform(X)
plt.figure(figsize=(8, 6))
plt.scatter(X_pca_2d[:, 0], X_pca_2d[:, 1], c=y, cmap='viridis', edgecolor='k', s=50)
plt.title('2D PCA of Iris Dataset')
plt.xlabel('Principal Component 1')
plt.ylabel('Principal Component 2')
plt.colorbar()
plt.show()
5.完整代码展示
import numpy as np
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
import time
# 加载示例数据集,这里使用iris数据集
data = load_iris()
X = data.data
y = data.target
# 存储不同主成分数量下的累计解释方差比例和计算时间
explained_variances = []
computation_times = []
reconstruction_errors = []
# 计算PCA并记录每次主成分数量下的累计解释方差比例、计算时间和重构误差
for n_components in range(1, X.shape[1] + 1):
start_time = time.time()
pca = PCA(n_components=n_components)
X_pca = pca.fit_transform(X)
explained_variances.append(np.sum(pca.explained_variance_ratio_))
computation_times.append(time.time() - start_time)
# 计算重构误差
X_reconstructed = pca.inverse_transform(X_pca)
reconstruction_error = np.mean((X - X_reconstructed) ** 2)
reconstruction_errors.append(reconstruction_error)
# 绘制累计解释方差比例折线图
plt.figure(figsize=(14, 6))
plt.subplot(1, 3, 1)
plt.plot(range(1, X.shape[1] + 1), explained_variances, marker='o')
plt.title('Explained Variance vs. Number of Principal Components')
plt.xlabel('Number of Principal Components')
plt.ylabel('Cumulative Explained Variance Ratio')
plt.xticks(range(1, X.shape[1] + 1))
plt.grid(True)
# 绘制计算时间折线图
plt.subplot(1, 3, 2)
plt.plot(range(1, X.shape[1] + 1), computation_times, marker='o')
plt.title('Computation Time vs. Number of Principal Components')
plt.xlabel('Number of Principal Components')
plt.ylabel('Computation Time (seconds)')
plt.xticks(range(1, X.shape[1] + 1))
plt.grid(True)
# 绘制重构误差折线图
plt.subplot(1, 3, 3)
plt.plot(range(1, X.shape[1] + 1), reconstruction_errors, marker='o')
plt.title('Reconstruction Error vs. Number of Principal Components')
plt.xlabel('Number of Principal Components')
plt.ylabel('Reconstruction Error')
plt.xticks(range(1, X.shape[1] + 1))
plt.grid(True)
plt.tight_layout()
plt.show()
# 使用前两主成分进行降维并绘制散点图
pca_2d = PCA(n_components=2)
X_pca_2d = pca_2d.fit_transform(X)
plt.figure(figsize=(8, 6))
plt.scatter(X_pca_2d[:, 0], X_pca_2d[:, 1], c=y, cmap='viridis', edgecolor='k', s=50)
plt.title('2D PCA of Iris Dataset')
plt.xlabel('Principal Component 1')
plt.ylabel('Principal Component 2')
plt.colorbar()
plt.show()
四、结果分析
4.1 输出结果图

图一:

图二
4.2 优缺点分析
优点:
- 降维效果显著:
通过解释方差折线图,我们看到前两个主成分可以解释大部分数据方差,展示了PCA在降维方面的有效性。 - 特征提取:
前几个主成分解释了大部分方差,表明PCA提取了数据中的主要特征。 - 可视化:
二维散点图展示了降维后的数据分布,便于观察和分析数据。
缺点:
- 线性假设:
如果数据不是线性可分的,二维散点图可能无法清晰分离不同类别的数据点,展示了PCA在处理非线性数据时的局限性。 - 信息丢失:
当主成分数量较少时,累计解释方差比例未达到100%,重构误差较高,说明降维过程中有信息丢失。 - 解释性差:
PCA转换后的主成分是线性组合,通常缺乏实际意义,难以从业务角度解释
4.3 总结
PCA是一种强大的降维工具,特别适用于高维数据的处理。它可以显著减少数据维度,提高计算效率,并有助于数据的可视化。然而,PCA也有其局限性,例如对非线性数据效果不佳以及主成分解释性差。在实际应用中,选择适当的主成分数量至关重要,以在降维和信息保留之间取得平衡。
通过理解PCA的优缺点,我们可以更好地应用PCA,并在适当的场景中使用其他补充方法,如非线性降维技术(例如t-SNE和UMAP),以获得更好的降维效果。























 46万+
46万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








