在计算机视觉中,YOLOv8是目标检测任务中的强大工具。尽管YOLO系列模型的传统应用主要集中在目标检测上(即定位和分类),但YOLOv8可以通过适当的训练和扩展,结合图像分割技术,实现细胞图像的目标分割、边缘识别、面积计算和灰度值计算等任务。
本文将详细阐述如何使用YOLOv8进行目标分割任务,并给出代码示例。主要内容包括如何标注数据、训练模型、进行推理以及后处理目标分割的步骤。
1. 环境准备和安装
首先,确保你的环境已安装YOLOv8相关依赖。你可以使用以下命令安装YOLOv8所需的库:
pip install ultra
然后,我们需要导入所需的库,并配置一些基本设置。
2. 数据准备与标注
对于YOLOv8进行目标分割任务,首先需要准备和标注数据集。YOLOv8一般使用Bounding Box(边界框)进行目标检测,但是如果需要分割,我们可以通过将标注转化为分割形式,或采用YOLOv8的Mask分支进行目标分割。
-
标注数据格式:YOLOv8的标注格式通常为TXT文件,其中每一行包含一个目标的类别编号及其位置(对于目标分割任务,位置包括目标的多边形顶点坐标)。具体格式如下:
class_id x_center y_center width height对于目标分割(即多边形标注),每个目标需要包含多个点,表示该目标的边界。
示例数据标注(以细胞图像为例):
0 0.5 0.5 0.2 0.2 # 第一类细胞 1 0.7 0.7 0.15 0.15 # 第二类细胞
你可以使用工具如LabelMe或CVAT进行标注,将图像中的细胞或目标转换为上述YOLO格式。
3. YOLOv8模型配置和训练
YOLOv8不仅支持经典的目标检测任务,还扩展了Mask分支支持目标分割。因此,你可以选择训练一个目标分割模型来处理细胞图像分割任务。首先,你需要定义模型配置,设置训练参数。
from ultralytics import YOLO
# 加载YOLOv8模型
model = YOLO("yolov8n-seg.pt") # 加载预训练的分割模型,'n'表示nano版本,也可以选择其他版本
# 设置训练数据和训练参数
data_yaml = {
'train': './train_data', # 训练数据集路径
'val': './val_data', # 验证数据集路径
'nc': 2, # 类别数,例如:细胞1和细胞2
'names': ['cell_type_1', 'cell_type_2'] # 类别名称
}
# 开始训练
model.train(data=data_yaml, epochs=50, batch=16, imgsz=640, device=0)
data_yaml是一个字典,指定训练和验证数据集的位置、类别数及类别名称。train()函数用于训练YOLOv8模型,指定训练的epoch次数、batch大小、输入图像尺寸等参数。
4. 推理与目标分割
训练完成后,你可以加载训练好的模型并进行推理,进行目标分割(检测和分割细胞)。YOLOv8的predict()方法可以直接进行图像推理。
# 使用训练好的模型进行推理
results = model.predict(source='./test_images', conf=0.5, save=True)
# 查看推理结果
results.show() # 展示结果
results.save() # 保存推理结果
在上面的代码中,source指定了待推理的图像或文件夹路径,conf参数用于设定目标检测的置信度阈值。推理结果会保存到默认的输出文件夹中,你可以通过results.show()查看检测和分割的图像。
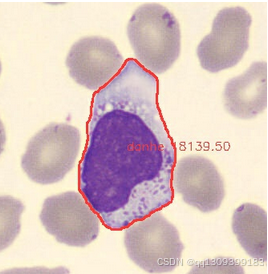
5. 目标分割后处理:边缘识别、面积计算和灰度值计算
YOLOv8不仅可以检测细胞的位置,还可以输出目标的边缘信息(Mask),接下来我们可以对分割结果进行后处理,计算每个目标的边缘、面积和灰度值。
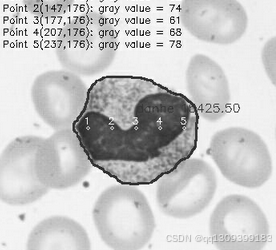
边缘识别
YOLOv8的分割结果包含目标的Mask(分割掩码),这是一个二值图像,表示目标区域。你可以使用OpenCV来进一步处理边缘信息。
import cv2
import numpy as np
# 读取分割结果的Mask
mask = results.masks[0].cpu().numpy() # 假设这是第一个检测到的目标
# 转换为二值图像
binary_mask = (mask > 0.5).astype(np.uint8)
# 使用Canny算法进行边缘检测
edges = cv2.Canny(binary_mask * 255, 100, 200)
# 显示边缘图
cv2.imshow('Edges', edges)
cv2.waitKey(0)
cv2.destroyAllWindows()
面积计算
细胞的面积可以通过计算分割掩码中像素的数量来获得。如果目标区域是二值图像,面积就是白色像素的个数。
# 计算目标的面积
area = np.sum(binary_mask)
print(f"Cell Area: {area} pixels")
灰度值计算
对于灰度值计算,YOLOv8的目标分割结果通常是基于原始图像的像素值,可以提取目标区域的灰度值进行分析。
# 读取原始图像
image = cv2.imread('test_image.jpg', cv2.IMREAD_GRAYSCALE)
# 获取目标区域的灰度值
cell_region = image * binary_mask # 将非目标区域置为0
mean_gray_value = np.mean(cell_region[cell_region > 0]) # 计算目标区域的平均灰度值
print(f"Mean Gray Value of the Cell: {mean_gray_value}")
在这段代码中,binary_mask是YOLOv8分割掩码,image是原始图像的灰度图,通过对目标区域的灰度值进行计算,我们可以得到细胞区域的平均灰度值。

6. 总结
YOLOv8不仅支持目标检测,还可以扩展到目标分割任务,适用于细胞图像分析等应用。在本文中,我们展示了如何使用YOLOv8进行细胞图像的标注、训练、推理,并通过后处理步骤进行边缘识别、面积计算和灰度值计算。
通过YOLOv8的强大功能和简洁的API,细胞图像的分割、分析变得更加高效和精确。这为生物医学研究中的自动化分析提供了有力工具,尤其是在大规模图像分析和实时处理任务中,YOLOv8展现出了其优越的性能和应用潜力。
























 528
528

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








