U-Net: Convolutional Networks for Biomedical Image Segmentation
通过阅读这篇论文了解到在医学图像领域还是有这样一个网络存在, 它是用于获得图像的边缘. 文中说是FCN的延伸, 原谅我的孤陋寡闻,还没有阅读到FCN的原文, 唉,一个人搞这方面的太孤独了,连获得信息的途径都没有,希望广大网友指教,再次谢谢.
U-net这篇论文的作者是参加一个ISBI的竞赛, 获得了不错的效果,然后将其的成果分享给大家,以供大家学习.http://brainiac2.mit.edu/isbi_challenge/ 这是ISBI的官网.
1 这篇论文使用的网络结构:
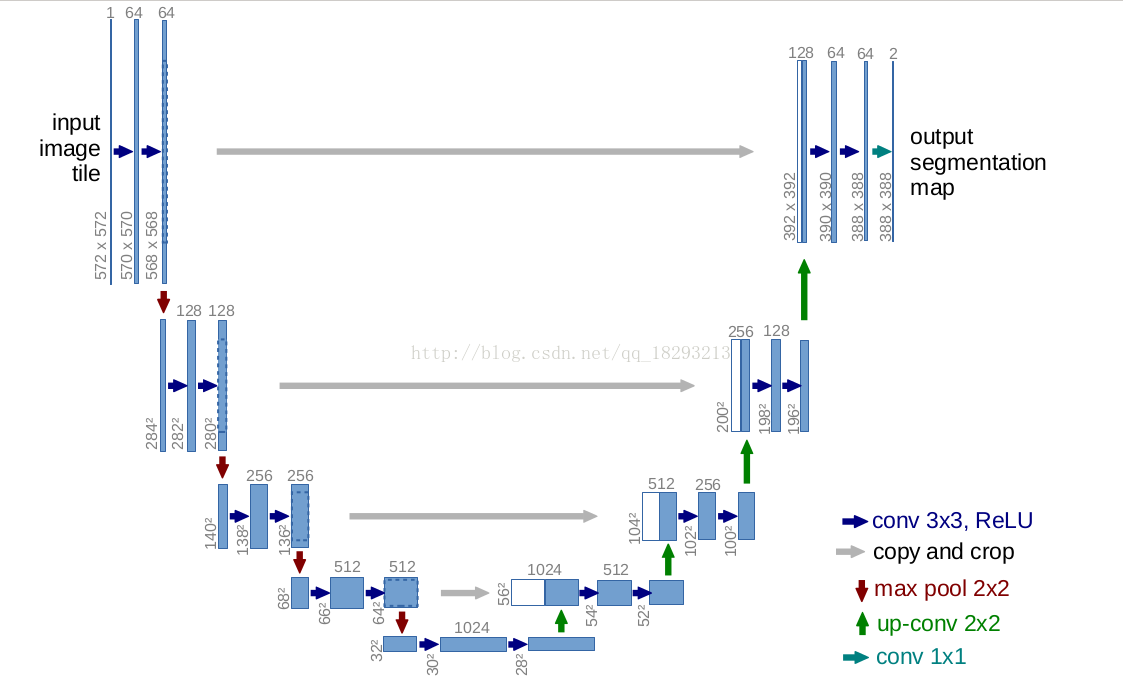
网络结构如图所示, 蓝色代表卷积和激活函数, 灰色代表复制, 红色代表下采样, 绿色代表上采样然后在卷积, conv 1X1代表核为1X1的卷积操作, 可以看出这个网络没有全连接,只有卷积和下采样. 这也是一个端到端的图像, 即输入是一幅图像, 输出也是一副图像. 好神奇.
2 ISBI 竞赛是一个关于细胞分割的竞赛, 或者说是细胞边缘检测的竞赛, 这个比赛官方只提供了30张训练图像, 30张测试图像. 数据量非常少, 怎么办? 我们可以做数据增强, 数据增强之后数据还不是很多, 没问题, U-net适用于小数据集(这个不是很准确,也没有官方的说明).
3 做这个问题的思路, 大约一个多月以前看到交大某位大神的博客(后来了解到竟然是老乡), 他的博文地址
http://blog.csdn.net/u012931582/article/details/70215756 , 我也是根据他的代码来改的,还没达到大神的能力.
(1) 官方提供的是一个tif文件的数据,将30张512X512的图片压缩(暂时理解为压缩吧)或者堆叠在一起,一开始我还以为就一张训练图像,一张label, 一张测试图像,还把我苦恼了一段时间(汗). 首先要安装libtiff这个python包,目前只能在python2上安装成功,(pip install libtiff), python3没有安装成功,又把我苦恼了一段时间.安装之后就可以将这看似一张的图像,转换为30张512x512的图像.方法大致如下:
from libtiff import *
imgstack = TIFF3D.read_image('train-volume.tif')
for i in range(imgstack.shape[0]):
savepath = '../../deform/tarin/' + str(i) + '.tif'
img = TIFF.open(savepath, 'w')
img.write_image(imgstack[i])
同样的方法,我们也可以将label(图像),和测试集分开来.这样训练集,测试集,label(GroundTruth), 都有了.但是数据量太少,怎么办?数据增强把,参考 http://keras-cn.readthedocs.io/en/latest/blog/image_classification_using_very_little_data/ ,这是使用keras实现数据增强的例子(data augmentation).数据增强的时候要注意,因为你的训练集中的每一个图像和label是一一对应的,所以你的每一张训练图像是怎样扭曲加噪声,label就要怎样扭曲加噪声,大神给我提供了一个思路,就是把
label当做训练图像的一个通道,这样他们就可以进行同样的数据增强了.
(2)数据增强之后就可以进行训练网络了,将训练集,label,测试集生成一个npy文件,然后送入U-net就可以了.然后对在测试集上进行测试,跑的特别慢.结果如下:

代码详见:https://github.com/silencemao/detect-cell-edge-use-unet
有问题请批评指正,谢谢.打个广告,新建的语义分割群,群号674968699, 有问题请进群讨论(不仅仅是语义分割), 可能还只有群主一个人(捂脸)






















 6519
6519











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








