I. 1984 Zhang的算法
Ref:1984 ACM Scikit-Zhang-A fast parallel algorithm for thinning digital patterns

文章的主要算法就是这4个条件来一遍一遍的去除边界点, 每遍会分为2步。
我对第一步的评论:而且还包括西北顶点也满足,比如H图FIg4a中的左上角的顶点,在第一次迭代中也满足条件,即P2、P8同时为0的 左上角也满足
第二步是将cd两个条件改为一下这两个。
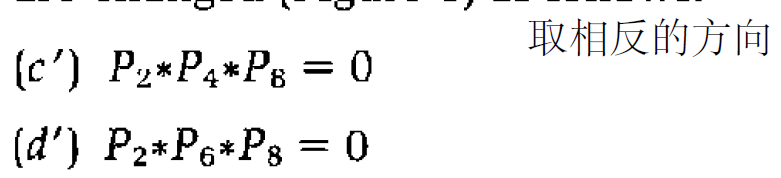
但是scikit中并没有源码的展示用的cython,好在算法比较简单。
skeletonize
return _fast_skeletonize(image)
这两步的区别在于 分别针对 东南 和 西北 边界点。 例如下面对H字母的 两步的结果如下:


II. Lee 1994年 八叉树octree 3D Skeleton
被MATLAB和scikit image收录的算法
Ref:1994 Building skeleton models via 3-D medial surface axis thinning algorithms
但是相比之下,Lee的 八叉树的算法,更符合人的理解, 避免了很多的分支, 估计这也是被scikit 和 MATLAB 收录的原因之一。 这个算法目前还在学习中。

该图来自scikit image的官方sample :linked websit
其源码在实现上 是 对2D图像补充了边缘,使用_compute_thin_image的cy程序来快速计算。
img = np.ascontiguousarray(img)
img = img_as_ubyte(img, force_copy=False)
# make an in image 3D and pad it w/ zeros to simplify dealing w/ boundaries
# NB: careful here to not clobber the original *and* minimize copying
img_o = img
if img.ndim == 2:
img_o = img[np.newaxis, ...]
img_o = np.pad(img_o, pad_width=1, mode='constant')
# normalize to binary
maxval = img_o.max()
img_o[img_o != 0] = 1
# do the computation
img_o = np.asarray(_compute_thin_image(img_o))
# crop it back and restore the original intensity range
img_o = crop(img_o, crop_width=1)
if img.ndim == 2:
img_o = img_o[0]
img_o *= maxval
return img_o






















 2300
2300











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








