摘要
长非编码RNA(lncRNA)参与多种生物学过程,因此其突变和疾病在多种人类疾病的发病机制中起着重要作用。识别与疾病相关的lncRNAs对于疾病的诊断、预防和治疗至关重要。尽管已经开发了大量计算方法,但有效整合多组学数据并准确预测潜在的lncRNA疾病相关性仍然是一个挑战,尤其是对于新的lncRNA和新疾病。在这项工作中,我们提出了一种新的深度多网络嵌入方法,称为DeepMNE,以发现潜在的lncRNA-疾病关联,尤其是对于新疾病和lncRNAs。DeepMNE提取多组学数据来描述疾病和lncrna,并提出了一种基于深度学习的网络融合方法来整合多源信息。
此外,DeepMNE补充了稀疏关联网络,并使用核邻域相似度构建疾病相似度和lncRNA相似度网络。
此外,采用图嵌入方法预测潜在关联。实验结果表明,与其他最先进的方法相比,DeepMNE对新关联、新lncRNAs和新疾病具有更高的预测性能。
此外,DeepMNE在扰动数据集上也具有相当好的预测性能。此外,两种不同类型的案例研究结果表明,DeepMNE可以作为疾病相关lncRNA预测的有效工具。DeepMNE的代码可在https://github.com/Mayingjun20179/ DeepMNE 上获得。
索引项深度网络融合、核邻域相似性、lncRNA疾病关联、多网络嵌入。
Introduction
蛋白质编码基因在遗传信息的存储中起着重要作用,但长期以来,RNA被认为只是一种转录介质。随着RNA分析、细胞类型分离和培养技术的不断改进,我们对RNA众多生物学功能的理解不断发展[1]。RNA代表基因组编码基因的直接输出 信息。
细胞的调节能力很大一部分高度依赖于RNA的合成、加工、运输、修饰和翻译[2]。最近的研究已经证明,约74.7%的人类基因组被转录,但这些基因中只有约1.5%编码蛋白质[3]。这表明绝大多数RNA转录本是非编码的,大量人类基因通过非编码RNA(ncRNA)发挥其功能。因此,非编码RNA(ncRNA)在各种生命过程中起着至关重要的作用,如基因转录和编译等。ncRNA的缺失将导致相邻蛋白质编码基因的特异性显著降低[5]。长链非编码RNA(lncrna)是非编码RNA中最大的部分,由至少200个核苷酸组成,到目前为止,还没有从这些RNA分子中观察到蛋白质编码潜力[6]。lncRNA具有广泛的作用,从胚胎发育、细胞命运决定到维持细胞的生理稳态。在生命的各个阶段,lncRNA甚至参与许多正常生理过程,包括渗透和摄取[7]。越来越多的研究报道,影响lncRNA的突变和疾病与几种人类疾病有关。lncRNA的一级结构、二级结构、表达水平和同源结合蛋白的变化可导致各种疾病,从神经病变到癌症【8】。例如,Dey等人进行的一项研究表明,在成肌细胞中沉默lncRNA H19并敲除H19基因可以显著减少骨骼肌分化[9]。Xin yu等人证明lncRNA MALAT1可以通过miR-101和miR-217实现食管鳞状细胞癌(ESCC)的转录后调节[10]。Gao等人的研究表明,lncRNA 91H通过抑制IGF2的表达参与ESCC的发病机制【11】。因此,确定潜在的lncRNA与疾病的相关性有助于确定lncRNA的确切功能,并从细胞水平深入了解疾病的潜在发病机制。
LncRNA是阐明疾病发生机制的关键。研究人员越来越多地参与lncRNA的研究,并建立了lncRNA疾病相关性数据库,如LncRNADisease【12】、LncRNAdb【13】、Lnc2Cancer【14】、MNDR【15】等。
然而,经实验验证的lncRNA疾病相关性的比例仍然很低。通过实验手段检测lncRNA疾病相关性不仅需要大量的人力和物力成本,而且需要长时间的投资。因此,计算模型已成为首选的调查手段。计算模型不仅节省时间,而且更便于获得候选关联的排名。这些排名可以作为实验验证的指南,这大大减少了寻找新的lncRNA疾病关联所需的成本和时间。由于所有现有数据库仅提供关联证据,因此没有明确证据表明lncRNA与疾病之间缺乏相关性。
因此,如果所有经实验验证的关联都被视为正例,那么lncRNA疾病关联推理问题可以被视为PU学习问题(基于正例和未标记样本的学习)。随后,根据产生负样本的必要性,现有的计算模型可分为两步技术和基于网络的模型。其中,两步技术主要包括两个步骤:产生负样本和建立训练模型。Zhao等人【16】提取了多组学数据,如lncRNA的基因组特征、调节因子特征和转录组特征,拼接这些特征数据,并使用朴素贝叶斯分类器识别疾病相关的lncRNA。Lan等人[17]利用多源信息计算lncRNAs的多个相似度和疾病的多个相似度,利用矩阵的几何平均值来整合这些相似度网络,然后使用bagging SVM建立分类。
基于网络的模型不需要构建负样本,可以有效利用未标记样本信息和网络结构。基于这些事实,人们提出了大量的网络模型。Qingfeng Chen等人[18]使用lncRNA基因关联、疾病DAG网络和lncRNA疾病关联网络计算lncRNA与疾病之间的多重相似性,并采用SVM进行预测,同时使用bagging方法处理类差异平衡问题。Wei Lan等人【19】利用自动编码器来缓解lncRNA(或疾病)特征信息中的噪声,并利用矩阵分解和隐式反馈来预测潜在的lncRNA疾病关联。Lu等人[20]使用疾病GO关联信息计算疾病的Jaccard相似性,从已知lncRNA疾病关联网络中挖掘特征信息,并使用归纳矩阵完成预测潜在关联。Li等人[21]利用疾病的语义相似性和已知的lncRNA疾病关联计算lncRNAs的功能相似性,然后利用网络一致性投影进行推理。Wang等人[22]使用加权K近邻来完成相互作用网络,并使用图正则化非负矩阵分解来预测潜在的lncRNA疾病关联。Xie等人【23】提出了加权矩阵lncRNA疾病关联预测模型(WLDAP)。与其他方法不同,WLDAP仅使用已知的lncRNA疾病关联进行预测。Yue等人[24]回顾了图嵌入方法在生物医学网络中的应用,并将11种图嵌入表示方法应用于3种生物医学链接预测任务。研究发现,图嵌入方法在不使用任何生物特征识别的情况下取得了有竞争力的性能。Zhang等人[25]应用多模态深层自动编码器从多个生物网络中学习统一表示,并结合随机森林分类器实现生物链接预测,取得了良好的预测结果。
尽管基于网络的方法在lncRNA疾病关联的推断方面取得了巨大成功,但其应用仍存在一些局限性。首先,许多模型仅利用已知的关联网络和单一疾病信息进行预测,未能充分利用多源信息。
其次,对于多个相似网络,许多模型仅采用线性方法进行融合,无法适当挖掘不同网络之间的非线性结构。
第三,大多数模型在预测新的lncRNAs或新疾病方面较弱。
基于此,本文提出了一种深度多网络嵌入模型(DeepMNE)来推断潜在的lncRNA疾病关联,该模型也适用于新的lncRNA和新的疾病。为了全面、客观地描述lncRNA和疾病,DeepMNE采用疾病的有向无环图、疾病GO注释和疾病基因关联构建疾病相似网络,并实现lncRNA的序列特征和表达谱特征构建lncRNA相似网络。为了挖掘不同网络之间的非线性关系,DeepMNE提出了一种基于深度学习的网络融合方法,以整合多个网络信息。此外,为了消除网络稀疏性以获得更准确的特征信息,DeepMNE对稀疏关联网络进行了补充,并使用核邻域相似度来挖掘lncRNA(disease)的关联相似度。实验结果表明,与其他模型相比,DeepMNE对新的关联、新的疾病和新的lncRNAs具有良好的预测能力。
材料和方法
A.方法回顾
为了计算lncRNA疾病的潜在关联,我们在此介绍DeepMNE模型,该模型包含以下三个步骤,如图1所示。

Fig 1 DeepMNE预测潜在lncRNA疾病相关性的框架
B. 疾病相似性
1) 疾病的语义相似性
d disease TA 代表着疾病d的集合和他的祖先 Ed代表DAG中的一条疾病和其edge
根据疾病网格描述符,所有疾病都可以投影到有向无环图directed acyclic graph (DAG)中。
DAG中的每个节点对应一种疾病,DAG的任何有向边都指向从一般疾病项到更具体疾病项的方向。
如果有任何疾病𝑑, 相应的有向无环图表示为DAG𝑑=(𝑑,𝑇𝑑,𝐸𝑑) ,哪里𝑇𝐴 表示包含疾病的节点集𝑑 以及它的所有祖先𝐸𝑑 表示DAG中这些节点的有向边集。 Wang等人【26】相信DAG𝑑,== 距离越短𝑑, 对于d来说语义表达更好==。 . 因此,对于任何疾病𝑡 以DAG为单位𝑑 , 语义对疾病的贡献值𝑑 具体如下:

哪里𝐷𝑑(𝑡) 表示疾病的语义贡献值𝑡 致疾病 disease 𝑑, 疾病的语义贡献值𝑑 到自身为1。同时𝑤 表示语义贡献因子(通常,𝑤 = 0.5) [26]. 疾病的语义价值𝑑 定义为DAG中所有疾病的==语义贡献值之和𝑑 具体如下: ==

两种疾病在DAG中的常见部位越多,其语义相似度越大。因此,对于任何两种疾病𝑑1和𝑑2、语义相似度 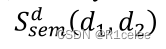
可计算如下:
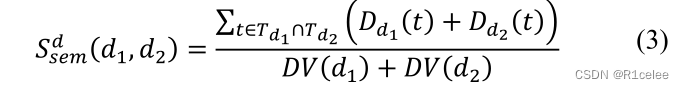
Dd1(t) 和D d2(t)代表着疾病t对 d1和d2两种疾病的语义贡献程度(semantic contribution)
DV(d1)和DV(d2)代表的d1和d2的语义值
2) 基于有向无环图的疾病功能相似性
基于GO项的有向无环图,James Z.Wang等人[27]提出了一种计算模型来度量GO项的语义相似度。Insuk Lee等人【28】提供了一个大规模的人类基因功能关联网络,该网络使用每条边的相关对数似然得分(likelihood score)(LLS)来衡量任何两个基因之间关联的强度。根据基因的对数似然得分,肖等[29]通过简单的处理建立了基因相似性网络。为了获得更准确的疾病功能相似网络,我们从疾病GO关联和疾病基因关联两个角度计算了疾病的功能相似性。为便于解释,本节采用疾病基因功能相似度的计算(𝑆𝐷𝑔en) 例如。
首先,相似性𝑆𝑔(𝑔, G) 基因𝑔和基因集G的定义如下【30】:


其次,让G1和G2表示与疾病相关的基因集𝑑1和d2。在这些条件下,基因功能相似
计算如下 :
其中|∙| 表示元素数。类似地,根据(4)和(5),使用疾病-GO关联和GO项目的语义相似性,疾病的GO功能相似性
可获得 .
(属于G1的和G2的相似度+属于G2的和G1的相似度)/(G1和G2的种类数)
C. lncRNAs的相似性
研究表明,尽管功能性lncRNAs缺乏线性同源性,但它们通常具有相似的k-mers(长度为k的核苷酸子序列)。这些k-mers与lncRNA结合蛋白和亚细胞定位有关[31]。序列k-mer通常用于描述序列的短期顺序信息,而序列的全局或长期信息可以通过核苷酸的物理和化学性质来描述[32]。为了整合长期和短期特征,本文提取了lncRNA序列的一般平行相关伪二核苷酸组成(PC-PseDNC)特征,并使用核邻域相似性(KSNS)[33]
构建序列相似性网络lncRNAs的Sexpl。此外,我们还提取了lncRNAs在24种人体组织或细胞类型中的表达谱,并计算了表达谱相似度Sexpl由KSNS提供。
D、 基于深度学习的网络融合
本研究使用多源数据构建多个相似度网络,每个网络从不同的角度描述相似度之间的关系。开发一种有效的方法,从这些网络中提取常见和重要的信息,消除网络噪声,已成为当务之急。改进的扩散分量分析(clusDCA)[34]使用重新启动随机游走和奇异值分解(SVD)来实现网络融合。在此过程中,SVD进行降维,通过线性投影将原始高维空间映射到新的低阶表示空间。然而,由于实际问题的复杂性,这些浅层、非线性技术往往难以捕捉复杂、高维的非线性网络结构。最近,Vladimir等人[35]使用多模式深度自动编码器(deepNF)从多个相互作用网络中提取蛋白质的高级特征。与其他浅层模型相比,deepNF能够有效捕获复杂且高度非线性的网络结构。
基于上述事实,本文提出了一个基于深度学习的网络融合框架,如图2所示。

该框架主要包括两个步骤:
首先,对每个预处理的异构网络,采用叠加自动编码的方法获得其高层低维特征表示;
其次,对所有低维特征进行集成,利用堆叠式自动编码器获得集成的低维特征,并利用KSNS构建融合网络。
该框架结合了DNGR(用于图形表示的深度神经网络)[36]和KSN的优点。一方面,利用DNGR的随机浏览和堆叠自动编码器对加权图进行处理,不仅降低了网络噪声,而且挖掘了多个网络的非线性结构。另一方面,利用KSNS重构融合网络,不仅可以计算样本的结构相似度,还可以分层挖掘邻域和非邻域的非线性信息。
该过程主要包括三个关键技术特征:网络预处理、堆叠式自动编码器和核邻域相似度。
首先,参考DNGR【36】,预处理步骤包括重新启动随机浏览和计算移位()正逐点互信息(positive pointwise mutual information )(PPMI)矩阵。为此,让
P
t
k
ϵ
R
m
∗
m
P_{t}^{k}\epsilon R^{m*m}
PtkϵRm∗m
表示第k个网络在第t步的转移概率矩阵,其计算公式如下:

𝑆𝑘 ∈ 𝑅m*m 表示𝑘-th相似性网络,以及𝑚 表示网络大小。 α 表示转移概率 。𝑃0 = 𝐼m×m 表示大小的单位矩阵𝑚乘𝑚。 在T次迭代后,最后的概率共生矩阵如下:


数据集
LncRNADisease数据库提供了大量lncRNA疾病关联、circRNA疾病关联以及lncRNA、mRNA和miRNA之间的转录调控信息【12,43】。LncRNADisease目前已更新至2.0版。与版本1.0相比,更新版本包含更多的lncRNA、疾病信息和相关关联[43]。首先,本文从LncRNADisease 2.0数据库下载了经实验验证的lncRNA疾病关联,选择了“智人”物种,并删除了重复关联,以获得5714个lncRNAs和423种疾病之间的7696个关联。其次,为了实现对疾病的全面和准确描述,我们从比较毒理基因组学数据库(CTD)中提取了网状描述符和疾病层次信息【44】,还提取了疾病分子功能注释数据和疾病基因关联数据。然后,为了获得lncRNAs的相关信息,我们从非编码数据库中搜索lncRNAs的转录ID,并提取其序列数据和表达谱数据。最后,我们排除了没有MeSH描述符的疾病和没有转录ID的lncRNAs,并观察到714个lncRNAs和241种疾病之间总共有1692个关联,这是本文交叉验证和案例研究的基准数据集。
B.实验步骤
为了全面评估模型的预测性能,我们在之前研究的基础上,在以下三种情况下分别对lncRNA疾病关联预测进行了五重交叉验证(CV):
(1) “配对预测”情景,预测已知lncRNAs与疾病之间的未知关联。将所有已知的lncRNA疾病关联对随机分成5等份,其中4份用于训练,另一份用于测试。
(2) “新lncRNA”情景,预测与新lncRNA相关的疾病。将所有lncRNA随机分成五等份,其中四对lncRNA疾病关联对用于训练,其余对用于测试。
(3) “新疾病”情景,有助于预测与新疾病相关的lncRNAs。将所有疾病随机分成五等份,其中四对lncRNA疾病关联对用于训练,其余的lncRNA疾病关联对用于测试。
在“配对预测”场景中,原始lncRNA疾病关联非常稀疏。因此,我们选择了一倍的已知关联作为阳性示例,并进一步随机选择了相同数量的未知关联作为阴性示例,这两个关联都被用作测试集。训练集由剩余的正面示例和所有未知关联组成。以ROC曲线下面积(AUC)、精确召回率曲线下面积(AUPR)和F1测度(F1)作为评价指标。对于“新lncRNA”情景和“新疾病”情景,我们倾向于更加关注排名靠前的候选lncRNA(疾病)中已知关联的比例,即𝐻itrate【47】,如下所示:

参数分析
DeepMNE共有4个参数,即重要性级别参数𝑐 , 特征正则化参数α,邻域正则化参数𝜆, 和潜子空间维数𝑖. 其中,𝑐 控制已知交互相对于未知交互的重要程度。典型地,当𝑐 = 1、两者的相互作用同等重要;α控制潜在特征的贡献;𝜆 描述相似性网络的影响。为了准确确定这些参数的影响,我们评估了模型在所有参数组合下的“成对预测”情景下的预测性能。明确地𝑐 从{1,3,5,7,9}中选择,α从{0.01,0.1,1,10,100}中选择, 𝜆 从{0.01,0.1,1,10,100}中选择,以及r 从{50100}中选择。根据不同的参数组合,DeepMNE的平均AUPR值如图3所示。

结合图3中的(a)和(b),可以看出r= 100 , 𝑐= 3.𝜆 = 10,α=10,模型达到其最优AUPR值。如图(a)所示, 当r = 100,AUPR值的趋势𝑐 (蓝色曲线)更接近r = 50(红色曲线),但前者的最大AUPR值略好。无论是否r = 100或r = 50,模型在以下位置达到最大AUPR值:𝑐 = 3,表明重要性级别参数的设置在一定程度上提高了预测性能。如图(b)所示,𝜆和α对模型的影响较大。将参数设置为𝜆 =10 随着α的增加,模型的AUPR逐渐增加。当α=10时,得到模型的最大AUPR值。类似地,当α=10时,模型的AUPR随着𝜆. 什么时候𝜆= 10,模型的AUPR值达到最大。此外,当10≤ 𝜆 ≤ 100和10≤ α ≤ 100时,模型的AUPR值可以保持在0.93以上,这进一步证明了模型对正则化参数具有很强的鲁棒性。
基于以上分析,我们选择r = 100, 𝑐 = 3.𝜆 = 10,α=10作为DeepMNE参数,并进行后续实验。
模型比较
许多先进的计算模型已经被提出用于lncRNA疾病预测。在此,我们选取了一些较新的lncRNA疾病关联预测方法,以及一些最新的生物医学链接预测方法,即NCPLDA【21】、LDGRNMF【22】、LDA-LNSUBRW【48】、GRGMF【49】和GCN-CRF【50】,简要描述如下:
NCPLDA:基于疾病语义相似性和已知的lncRNA疾病关联,采用网络一致性预测来预测lncRNA疾病关联
⚫ LDGRNMF:基于疾病的语义相似性和已知的lncRNA-疾病关联,采用加权的K已知近邻和图规整的非负矩阵分解来进行预测。
⚫ LDA-LNSUBRW :基于疾病语义相似性和已知的lncRNA-疾病关联,使用线性邻域相似性和不平衡双随机Walk进行预测。
⚫ GRGMF:用于生物医学链接预测的图形正则化的广义矩阵因子。
⚫ GCN-CRF:用于微生物药物关联预测的条件随机场的图卷积网络。 然后,利用原始文件提供的代码和参数设置来进行预测。在10个随机种子下进行CV,并计算所有指标的平均值和方差.
然后,利用原始文档提供的代码和参数设置执行预测。在10个随机种子下进行CV,计算所有指标的均值和方差。
如表1所示

DeepMNE模型的平均AUPR、AUC和F1值高于其他模型。与NCPLDA、LNSUBRW、LDGRNMF、GRGMF、GCN-CRF的平均AUPR值分别为0.8720、0.7533、0.8893、0.9234和0.9297相比,DeepMNE的平均AUPR值达到了0.9505,分别增长了9%、26.18%、6.88%、2.93%和2.24%;DeepMNE模型的平均AUC值达到0.9462,与其他模型相比,分别增加了6.72%、50.05%、8.53%、3.6%和2.65% NCPLDA为0.8866,LNSUBRW为0.6306,LDGRNMF为0.8718,GRGMF为0.9133,GCN-CRF为0.9218。此外,DeepMNE模型的平均F1值也高于其他模型。对于“新lncRNA”和“新疾病”,我们计算了10个随机种子下前2%、4%、6%、8%候选lncRNA(或疾病)的平均命中率。具体结果如表2所示。

如表2所示,对于“新lncRNA”的预测,GCN-CRF在前2%、4%、6%和8%的候选疾病中的平均命中率最高。DeepMNE的预测结果仅次于GCN-CRF,两者非常接近。对于“新疾病”情景,DeepMNE的结果明显高于其他方法。具体而言,对于前2%的候选lncRNAs,DeepMNE的平均命中率达到0.3841,而NCPLDA的平均命中率为0.0529,LNSUBRW的平均命中率为0.3372,LDGRNMF的平均命中率为0.2013,GRGMF的平均命中率为0.3276,GCN-CRF的平均命中率为0.3465,分别增长了626.09%,13.91%,90.81%,17.25%和10.85%。同时,DeepMNE在前{2%、4%、6%、8%}候选lncRNAs上的平均命中率也高于其他方法。
与其他方法相比,DeepMNE的良好性能主要归功于以下三个方面:
(1)DeepMNE提取多源数据,从多个角度描述lncRNAs(疾病)之间的关系。多源数据的信息补充可减轻因数据丢失而导致的错误。
(2) 深度多网络融合框架结合了DNGR和KSNS的优点。也就是说,它不仅可以挖掘多个网络的非线性结构,而且可以分层挖掘邻域和非邻域特征之间的非线性关系。
(3) NRLMF的邻域运算、重要性水平设置和潜在特征完成都在一定程度上提高了模型的预测能力
E、 模型稳健性分析
由于技术限制、实验误差等原因,已知的lncRNA疾病关联网络往往含有噪声,具体表现为关联信息缺乏、关联信息不真实等。为了检验模型对已知相互作用的依赖性,基于之前的研究【51】,将噪声添加到经过训练的lncRNA疾病关联网络中,以生成扰动数据集,并在扰动数据集上进行实验,以评估模型的预测能力。具体而言,随机选择20%的已知关联和相同数量的未知关联作为测试集,并将1%的噪声添加到训练网络中(即,改变训练网络中1%的lncRNA疾病对的标签)。然后,计算10个随机种子下所有模型的AUPR和AUC值。箱线图如图4所示。

从图4可以看出,在受干扰的数据集上,DeepMNE获得了更好的平均AUPR和AUC值。具体而言,DeepMNE的平均AUPR值为0.9317,分别比NCPLDA的0.8734、LNSUBRW的0.8644、LDGRNMF的0.7323、GRGMF的0.8574和GCN-CRF的0.8357高出6.67%、7.77%、27.22%、8.66%和11.48个百分点。同时,DeepMNE模型的平均AUC值为0.9196,高于NCPLDA的0.8637、LNSUBRW的0.8334、LDGRNMF的0.7384、GRGMF的0.8302和GCN-CRF的0.8158。
与表1中的结果相比,DeepMNE的AUPR和AUC值有所降低。主要原因是受扰动的数据集含有一定的噪声,导致关联相似度和完成的关联矩阵出现偏差,但DeepMNE模型仍然取得了令人满意的结果
Discussion
本研究提出了一个深度多网络嵌入(DeepMNE)模型来预测潜在的lncRNA-疾病关联。该模型的新颖之处在于将深度网络融合、核邻域相似度和图嵌入技术相结合,预测lncRNA-疾病关联概率。DeepMNE整合多组学信息构建多个疾病相似网络和多个lncRNAs相似网络,并利用深度学习模型实现多网络融合,以挖掘这些网络中的非线性结构相似性。此外,DeepMNE对稀疏关联网络进行了补充,利用核邻域相似度构建lncRNA(疾病)的相似网络,并计算lncRNA疾病关联得分。总之,DeepMNE利用多源信息的融合在网络之间进行信息交换,并利用图嵌入方法确定lncRNAs和疾病的低维特征,以减少数据噪声造成的错误。为了评估我们的DeepMNE模型的性能,我们基于最新的lncRNA疾病关联数据集进行了广泛的实验,并与其他最先进的lncRNA疾病计算方法进行了各种比较。实验结果进一步证实了DeepMNE模型的有效性。例如,对基准数据集的五倍交叉验证结果表明,对于新的关联、新的lncRNAs和新的疾病,DeepMNE可以产生更好的AUPR、AUC和F1值。此外,在扰动数据集上的实验表明,DeepMNE获得的AUPR和AUC值高于其他方法。案例研究1的结果表明,通过DeepMNE模型获得的前10、前20、前30和前40个候选关联在新数据库中得到了进一步验证。案例研究2中三种疾病的预测结果进一步证明,DeepMNE将是预测lncRNA疾病相关性的有效工具。








 DeepMNE是一种新型深度多网络嵌入方法,用于预测潜在的lncRNA-疾病关联,尤其适用于新关联、新lncRNAs和新疾病。它综合了多组学数据,利用深度学习融合多源信息,通过核邻域相似性处理网络稀疏性。实验证明,DeepMNE在新关联预测方面优于其他先进方法。
DeepMNE是一种新型深度多网络嵌入方法,用于预测潜在的lncRNA-疾病关联,尤其适用于新关联、新lncRNAs和新疾病。它综合了多组学数据,利用深度学习融合多源信息,通过核邻域相似性处理网络稀疏性。实验证明,DeepMNE在新关联预测方面优于其他先进方法。
















 2340
2340

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








