基于深度信念网络的表示学习用于lncrna -疾病关联预测
Deep Belief Network based representation learning for lncRNA-disease association prediction
摘要
在本文中,我们从lncRNA、疾病和miRNA的相互作用出发,提出了一种基于DBN(深度信念网络)的lncRNA-疾病关联预测模型(DBNLDA)。该体系结构包含三个主要模块:网络构建、基于DBN的特征学习和基于神经网络的预测。首先,我们构建了三个异质网络,如lncRNA-miRNA相似性(LMS)、疾病- mirna相似性(DMS)和lncrna -疾病关联(LDA)网络。通过相似网络的节点嵌入矩阵,通过两个基于DBN的子网网络分别学习lncrna -疾病表示。lncRNA-disease的联合表示由第三个DBN从上述两个子网的输出中学习。该联合特征表示用于ANN分类器预测关联得分。
结果:在使用最先进的方法进行标准数据集测试时,该方法的AUC为0.96,AUPR为0.967。对乳腺癌、肺癌和胃癌病例的分析也证实了DBNLDA在预测lncrna -疾病显著相关性方面的有效性。
1 介绍
lncRNA-disease关联的工作主要有三大类。第一类研究利用了lncRNA功能相似性的知识,假设功能相似的lncRNA与相似的疾病相关。基于这一假设,我们构建了一个lncrna -疾病关联网络。然后使用机器学习和社会网络分析算法进行lncrna疾病预测。例如,RWRlncD[7]、IRWRLDA[8]、BRWLDA[9]等方法采用各种随机游走算法,KATZLDA[10]采用Katz page排名算法分析相似度网络。所有这些方法都依赖于网络结构特征,结果都偏向于具有高度和中心性的节点。
对lncRNA机制的深入研究表明,lncRNA的调控在很大程度上是由共同表达的mirna[11]决定的。第二类研究研究了各种疾病中lncrna、基因和mirna的表达水平。早期的[12]方法使用了实验验证的疾病相关基因/ mirna和lncRNA共表达数据。这些方法对于没有经过实验验证的基因/miRNA相互作用的lncrna无效。在这类研究中,最近的研究使用了一些数学模型,如矩阵完成[13]、矩阵分解[14]和基于图形的算法(TPGLDA [15], DisLncPri[16])来预测lncrna与疾病的关联。
第三类作品基于lncRNA、miRNA和mRNA的功能相似性和关联性构建了异质相互作用网络。机器学习技术,如随机漫步[12],支持向量机[17],和拉普拉斯正则最小二乘法[18]已经被用于分析这些网络。上述方法的主要挑战是如何有效地表达lncrna疾病特征。深度学习模型的引入消除了特征提取的需要,实现了无监督表示学习。其中,CNNLDA[19]使用卷积神经网络,GCNLDA[20]使用图卷积神经网络[21]学习lncRNA、miRNA和疾病节点的全局表示。最近一项名为GAMCLDA[22]的研究使用了图形自动编码器和矩阵完成来预测lncrna -疾病关联。
深度学习算法如深度信念网络(DBN)最近被用于计算机视觉和文本挖掘,以学习数据[23]的潜在表示。DBN模型由限制玻尔兹曼机(RBM)堆叠而成,包含可见层和隐藏层,以计算概率分布作为潜在表示。基于DBN的模型成功应用于生物信息学预测药物靶点[24]、多种类型的mirna -疾病关联[25,26]和癌症亚型[27]预测,但未应用于lncrna -疾病关联预测领域。
在本研究中,我们提出了一种基于DBN的表示学习模型(称为DBNLDA)来预测lncrna -疾病关联。DBNLDA利用lncrna与疾病的功能相似性、共表达、相互作用等异质性信息进行预测。DBNLDA的体系结构包含3个模块:-(i)网络构建,(ii)基于DBN的特征学习和(iii)关联预测。图1描述了提议的体系结构的概要。与之前的研究不同[12,20],DBNLDA构建了lncRNA-miRNA (LMS)、疾病- mirna (DMS)和lncrna -疾病关联(LDA)三个功能性相似网络。然后,对于每一对lncRNA-疾病,DBN子网-1从lncRNA与mirna的功能关联中学习其和疾病表征。同样,DBN subnetwork-2从LDA网络中学习lncrna疾病表达。将子网学习到的表示结合起来,使用第三个DBN (DBN组合)学习更高级别的表示。为了减少稀疏网络学习特征的阻碍影响,利用注意层对dbn组合的特征进行重新计算。在最后阶段,使用基于神经网络的分类器,根据联合特征表示来预测lncrna与疾病的关联。对癌症数据集的5倍交叉验证和案例研究表明,DBNLDA显著提高了表现和潜在的lncrna -疾病关联预测。

图1:DBNLDA的架构。LMS: LncRNA-miRNA相似;DMS: Disease-miRNA相似;LDA: LncRNA-Disease协会;L:从LMS中嵌入lncRNAs;D: DMS疾病的嵌入;LD: lncrna的嵌入与LDA引发的疾病;DBN:深度信念网络。
2 材料与方法
2.1 数据集
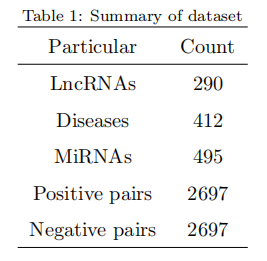
使用之前的报告[12,20,14]的数据集进行lncRNADisease相关性、lncRNA- mirna相互作用、lncRNA功能相似度和疾病语义相似度的分析。从两个参考数据库LncRNADisease[28]和lnc2cancer[29]下载LncRNA-disease关联。MiRNA-lncRNA相互作用和miRNA-disease相互作用分别从miRNet[30]和Starbase[31]数据库下载。然后,我们对240种lncRNA、412种疾病、495种mirna和2697种已知lncRNA-疾病相互作用的所有下载的关联和相似性进行了汇编。这些已知的交互对构成了训练模型的正例子。所有其他未在参考数据库中列出的lncrna与疾病的配对都被认为是负相互作用。我们从负交互作用中随机选择2967个样本来构建一个平衡数据集。数据集汇总见表1。
2.2 LMS、DMS和LDA网络的构建
DBNLDA体系结构的第一步是构建三个相似网络,如第1节定义的LMS、DMS和LDA。设nl、nm、nd为数据集中lncRNAs、miRNAs和疾病的数量。利用lncRNA-lncRNA相似性和lncRNA-miRNA相互作用构建LMS网络(包含nl lncrna和nm mirna)。本文采用Chen s方法[32]计算的LncRNA功能相似性,如果相似度评分大于0则添加一条边。对于lncrna的列表,我们使用从miRNet下载的已知miRNA靶点作为lncRNA-miRNA的边缘。
与LMS类似,利用疾病相似性和已知的疾病- mirna相关性构建了DMS网络(与nd疾病和nm mirna有关)。lncRNA -疾病关联(LDA)网络包含nl lncRNA和nd疾病,其中一个非定向边缘被用来代表lncRNA和疾病之间的已知关联。
2.3 计算节点嵌入网络特征
2.7 超级参数
不同的超参数决定了DBNLDA在不同模块中的性能。这些超参数的值是在绩效评估后根据经验设定的。对于所有网络,node2vec嵌入的维数e设置为64,其他参数保持[33]中的默认值。在[26]实现后,本研究中的DBN子网使用三层RBM堆叠,每层h = 128个节点。合并DBN的维数h0 = 256。我们发现,很低的值h和h0会降低性能,较高的值对性能没有影响(详见图2)。在分类模块中,两个隐藏层的神经元数均设置为128。分类器迭代了30多个迭代,因为模型的性能在第30个时代之后变得稳定。

图2:DBNLDA模型在DBN子网中不同节点数(h)与DBN组合(h0)的准确率比较点用(h, h0)值标注
DBNLDA步骤和实现细节的详细描述可以在SupplementaryFile-S1.pdf中找到。
3 结果与讨论
3.1 性能评估指标
我们使用5倍交叉验证来衡量和比较模型的性能。数据集包括5394对lncRNA-disease(2697对阳性关联和2697对阴性关联)。采用接受者工作特征(auco - roc)曲线下面积来比较预测模型的整体性能。以精确召回曲线下面积(AUPR)和平均准确率作为预测指标。

图3:5折交叉验证不同折线的学习曲线
3.2 整体性能
DBNLDA经过超过30个时期的训练,学习曲线(见图3)在所有交叉验证的折叠中显示出一致的特征。模型经交叉验证后平均AUC为0.96,ROC曲线如图4所示。模型的精度为0.957,AUPR值为0.968。
为了分析基于DBN的特征如何提高预测模型的性能,我们对不同层次的特征组合进行了重复实验。由表2可知,基于DBN的学习方法的引入显著提高了预测模型的精度。

图4:5折交叉验证不同折线的ROC曲线
3.3 与其他方法比较
基于ROC曲线下面积(area under ROC curve, AUC)和精确召回曲线下面积(area under precisionrecall curve, AUPR),比较了DBNLDA和其他常用方法如RFLDA[12]、GCNLDA[20]、SIMCLDA[13]、Ping s Method[34]、MFLDA[14]、LDAP[17]、GAMCLDA[22]和CNNLDA[19]的性能。以上方法都是利用来自不同数据源的异质性信息的知识来预测lncrna -疾病关联,未考虑网络结构特征。所有LDA预测模型的AUC和AUPR值见表3。除DBNLDA外,其他方法的AUC和AUPR值均取自[12]和[22]。从表中可以明显看出,DBNLDA报告的AUC是第二好的(0.96),与RFLDA报告的最高AUC值接近(少1.6%)。在AUPR值方面,DBNLDA优于其他所有方法,DBNLDA的AUPR值为0.968,比第二高的RFLDA高出18.9%。这些结果表明DBNLDA能有效预测lncrna -疾病的相关性。
表2:基于特征组合的模型精度

表3:与最先进方法的性能比较

3.4 案例研究
为了进一步研究DBNLDA预测lncRNAdisease显著相关性的能力,我们对乳腺癌、肺癌和胃癌进行了个案研究。在本研究中,首先在包含所有lncrna -疾病关联的数据集上训练DBNLDA模型,但lncrna与相关疾病(乳腺癌/肺癌/胃癌)之间的关联已得到验证。然后,使用训练过的模型计算所有lncrna与特定疾病的关联得分,并分析每种疾病的前15个候选lncrna。表4、表5、表6显示了DBNLDA预测的乳腺癌/肺癌/胃癌中排名前15位的候选lncrna。证据栏显示的是对参考数据库或文献关联的参考。
表4:前15位DBNLDA预测了与乳腺癌相关的lncrna


我们发现DBNLDA预测的13个(86.67%)与乳腺癌相关的lncrna也被lnc2cancer或LncRNADisease数据库证实。对于这两个未经证实的预测,我们可以从最近的出版物中找到证据。在肺癌病例中,15个预测量最高的lncrna中,有10个被文献数据库证实,其余均为近期文献报道。在胃癌病例中,DBNLDA可以预测参考数据库或文献报道的12种相关性。这三个新的关联(HCP5, HCG4和MIR99AHG,表6中*所示)可以作为进一步实验室验证的新建议。DBNLDA对LDA预测的详细比较见补充表2.xls。
表5:前15位DBNLDA预测肺癌相关lncrna

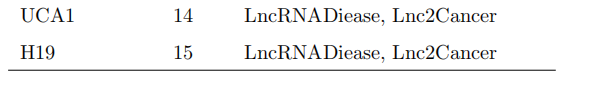
表6:前15位DBNLDA预测胃癌相关lncrna。

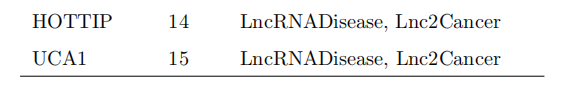
4. 结论
识别与疾病相关的lncRNA是识别lncRNA在疾病机制中的功能作用的必要步骤。在这项工作中,我们开发了DBNLDA,一种基于深度信念网络的lncrna -疾病关联预测模型。这项工作整合了lncRNA、miRNA、疾病相互作用和功能相似性的信息来构建异质网络。然后,利用基于DBN的lncrna与疾病的潜在表征来准确预测lncrna与疾病的关联。交叉验证证实DBNLDA在AUC方面有相当的表现,在AUPR方面有显著的改善。乳腺癌、肺癌和胃癌的病例研究表明DBNLDA具有预测潜在疾病相关lncrna的能力。该模型可以进一步扩展为多模态数据,如lncRNA药物-靶标相互作用和lncRNA-表观遗传学-疾病相互作用。





















 406
406











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








